《细胞》重磅:迄今最强肠癌基因变异现身,将刷新对促癌基因的认知!
我们对促癌基因的认知,又要刷新了。
今天,由哈佛医学院Po-Ru Loh和Ronen E. Mukamel,以及MIT-哈佛大学博德研究所Steven A. McCarroll和Robert E. Handsaker领衔的研究团队,在顶级期刊《细胞》上发表重磅研究成果[1]。
他们分析了英国生物库(UKB)中418136名参与者和GTEx数据库中838参与者的全基因组和表型数据,发现了两个重要的非编码可变数目串联重复序列(VNTRs),一个位于TMCO1内含子和另一个位于EIF3H下游,它俩分别与青光眼和结直肠癌风险增加2倍以上有关。
据研究人员统计,在目前已知的所有常见遗传变异中,这两个VNTRs对青光眼和结直肠癌的发生,贡献了最大的遗传力量。更重要的是,这些结果表明,非编码VNTRs在人类健康中发挥了重要的作用,而这一作用以前并未被我们所认识。简单来说,这一研究有望打开我们对癌症等疾病认知的新大门。

论文首页截图
从名字上就可以看出,可变数目串联重复序列(VNTRs)是一些重复几次到几百次的核苷酸序列串联在一起组成的。由于VNTRs的大小因人而异,一直以来受测序技术的限制,VNTRs与人体表型之间的关系很难研究。
近年来,技术的进步和研究方法的改进,让VNTRs与表型之间的研究变得可以实现。就在两年前,Loh团队就基于外显子组测序数据发现,可编码的VNTRs是身高、血清尿素和头发卷曲等不同表型的已知最强遗传基础[2],相关研究成果发表在顶级期刊《科学》上。这一次,他们想基于全基因组测序数据,探索非编码和可编码VNTRs在塑造人类表型和基因表达中的作用。
通过对人类参考基因组和人类基因组结构变异联盟(HGSVC)中部分数据的分析,Loh团队确定了9561个VNTRs用于后续的分析。他们先调用了迄今最大的自闭症数据库Simons Simplex Collection(SSC)中自闭症谱系障碍和UKB中118种神经发育表型数据,在关联分析中,他们意外地发现,9561个VNTRs对包括阿尔茨海默病在内的常见神经发育障碍的影响有限。
随后,他们将研究重点放在了非编码VNTRs上,并从中筛选了3个与表型相关性特别强的VNTRs,它们分别位于TMCO1的内含子、EIF3H的下游和CUL4A的内含子。
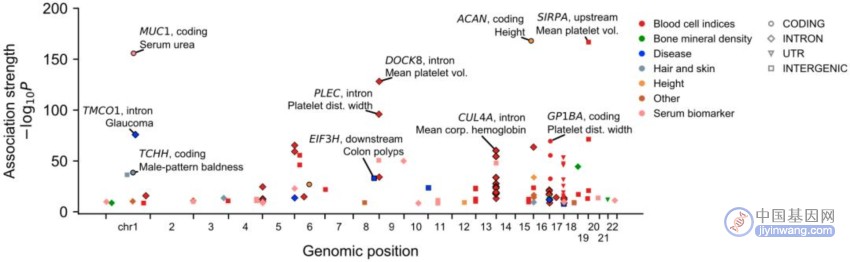
与不同性状相关VNTRs的所在位置
咱们先来看EIF3H下游这个VNTR,它与肠癌有关。
Loh团队认为,从遗传的角度看,大肠癌是一种复杂疾病,目前已发现一百多个常见风险位点,不过这些对肠癌风险的影响都很微弱(OR<1.2)[3]。
相比之下,EIF3H下游大约20kb的地方,长度为27bp的重复序列(通常2-6个重复单元),与结直肠癌和结肠息肉的患病风险密切相关。与基因组中跟肠癌有关的任何常见SNP或插入/缺失突变相比,最长的那个VNTR(6个重复单位)都与结直肠癌风险升高最多相关(OR=1.34)。通过比较,他们发现该位点的解释力在全基因组范围内的所有结直肠癌相关位点中排名第一。
值得注意的是,肠癌和息肉风险随VNTR长度的增加而线性增加,每增加一个重复单位,肠癌风险就增加14%(11%-17%),肠息肉风险增加9%(7%-10%)。而且两个等位基因的影响是叠加的,EIF3H下游的VNTR会导致不同个体的结直肠癌风险增加2倍以上。
此外,Loh团队还发现,在这个英国队列中,EIF3H下游的VNTR对英国肠癌患者的遗传性贡献了20%的力量。如果将EIF3H下游的VNTR纳入肠癌风险的多基因预测分析,可以将准确性提高25%。
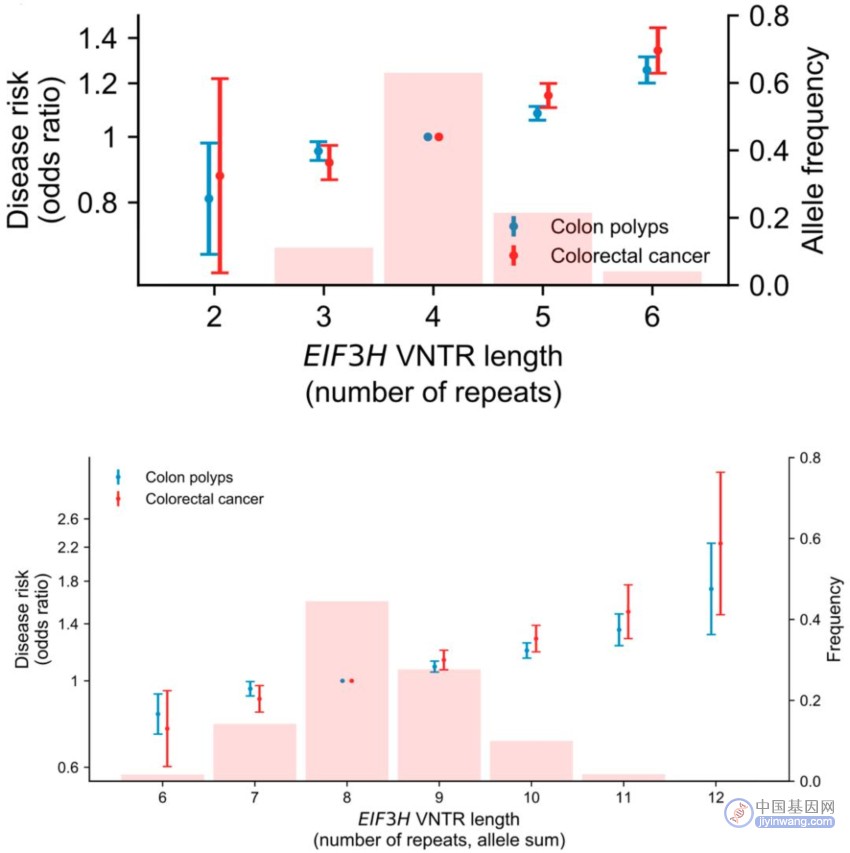
EIF3H下游VNTR与肠癌之间的关系
从Loh团队的研究结果来看,TMCO1内含子中的VNTR与青光眼风险的相关性,比其他任何相关SNP或插入/缺失突变都强。具体来说,与没有重复扩增的个体相比,长VNTR的纯合个体患青光眼的风险增加了2倍以上(OR=2.27)。
至于这两个非编码VNTR与疾病密切相关的分子机制,Loh团队没有找到。以EIF3H下游VNTR与肠癌之间的关系为例,EIF3H下游的VNTR对附近和远端基因的表达没有任何影响。这就需要科学家在后续的研究中去探索这些非编码VNTR促癌的具体机制。不过,在本研究中,Loh团队发现VNTR可以通过多种不同的机制影响基因剪接。
总的来说,Loh等人的这项研究成果让我们认识到了非编码VNTR对人类的健康有重要的影响,与癌症等疾病的发生密切相关。这一发现不仅让我们对癌症等疾病的发生有了新的认知,以此为基础开展的机制研究有望发现治疗癌症等疾病的新靶点。
参考文献:
[1].Mukamel et al., Repeat polymorphisms underlie top genetic risk loci for glaucoma and colorectal cancer, Cell, 2023, https://doi.org/10.1016/j.cell.2023.07.002
[2].Mukamel RE, Handsaker RE, Sherman MA, et al. Protein-coding repeat polymorphisms strongly shape diverse human phenotypes. Science. 2021;373(6562):1499-1505. doi:10.1126/science.abg8289
[3].Huyghe JR, Bien SA, Harrison TA, et al. Discovery of common and rare genetic risk variants for colorectal cancer. Nat Genet. 2019;51(1):76-87. doi:10.1038/s41588-018-0286-6
声明:本站所有文章资源内容,如无特殊说明或标注,均为采集网络资源。如若本站内容侵犯了原著者的合法权益,可联系本站删除。