哈佛团队研发新型表观组测序法,能同时检测DNA分子序列和基因调控信息
最近,在哈佛大学医学院丹娜法伯(Dana-Farber)癌症中心从事博后研究的清华校友吴婧怡和所在团队,利用三代测序技术修正了基因组的组装错误,发现了一个新的基因调控元件。
该技术可同时检测 DNA 分子上的序列和基因调控信息,在临床上有望用于部分遗传疾病的分子诊断以及父母本溯源,并有望在癌症早期检测癌细胞的组织来源。
(来源:Nature Genetics)
01
半个世纪的三代测序技术
据介绍,测序技术起源于 20 世纪 70 年代。1970 年,中国工程院外籍院士、著名华裔科学家、美国康奈尔大学教授吴瑞报道了一项新型测序技术,即在人为复制 DNA 的过程中,加入放射性标记的核苷酸,从而区分出 DNA 上不同位置的内容,进而读出 DNA 的序列。
随后,两度获得诺奖的英国生物化学家弗雷德里克·桑格(Frederick Sanger)和其他学者,联合改进了这一方法。改进后的方法被更多的科学家使用,第一代测序技术 Sanger 测序由此诞生,桑格教授也因此获得 1980 年诺贝尔化学奖。
但是,这种测序成本高、效率低,难以用于大规模基因组例如人类基因组的测序。20 世纪 90 年代,科学家开发了多种新型测序方法。相比于第一代测序,这些技术仍然依赖于合成 DNA 的过程,故被称为“边合成、边测序”。
这些技术的好处在于,不再依赖放射性元素,而是以显微镜可捕捉的荧光信号,来读取合成中的 DNA 信息,极大提高了测序效率。
随后,大量初创公司加入测序技术的商业化竞争,包括 、 和 等公司。最终, 公司的测序平台被 公司于 2007 年收购,并以具有竞争力的价格和高质量的数据垄断全球市场,成为了当今的主流测序技术,也被称为第二代测序技术。
如今,第二代测序技术已被广泛用于人类生活,科研人员用其检测新冠病毒如何变异,医生用其检测准父母是否携带遗传疾病的基因,公司用其提供个性化的 DNA 测序服务。
但是,第二代测序技术存在的最大问题是,它只能一次读取 100 个左右碱基长度的 DNA。人类基因组一共有 30 亿个碱基,二代测序难以精确定位基因组大片段的变异、以及高度的重复序列。
于是,学界开始思考如何一次性测出更长的 DNA 分子。第三代测序技术也应运而生,目前市场上主流的三代技术是单分子实时荧光测序和纳米孔测序。这两种测序方式都可以直接读取较长的 DNA 片段并和 DNA 上的化学修饰。
DNA 上的化学修饰往往具有生物学功能,能够直接调控基因的表达,越来越多的生物学家开始青睐于这项技术。
02
为教科书增加新内容
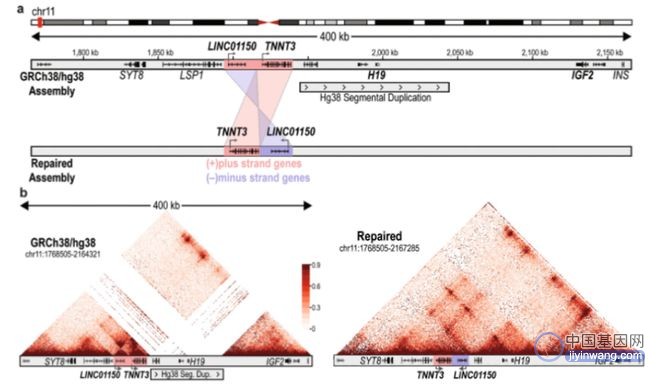
吴婧怡和所在团队开发的新方法正是基于纳米孔测序。首先,他们利用三代测序成功修正了一些人类基因组中的错误拼接和注释。
其次,相比于直接进行三代测序,本次方法做了两个主要改进。一是课题组用酶修饰了基因组上蛋白结合的地方(蛋白结合的区域通常是调控基因表达的区域),且这种修饰可以被三代测序检测到。二是他们用基因编辑技术,截取了感兴趣的位点进行深度测序。
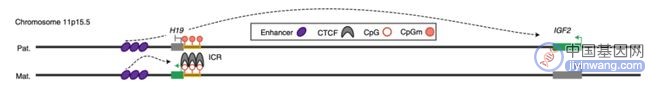
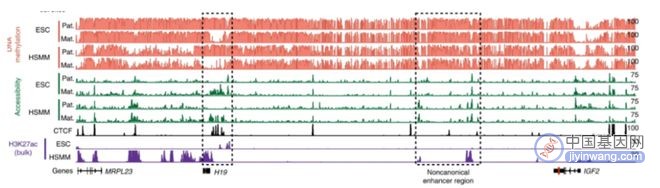
最后,该团队结合深度测序的序列信息和上面的蛋白结合信息,成功区分出了 DNA 的父母本来源,借此发现了一个调控 IGF2 基因的新元件。
(来源:Nature Genetics)
据悉,IGF2 基因主要调控生长发育,其表达量需要精确的调控,才能实现正常的功能。过多或者过少,都可能导致发育缺陷或者癌症等严重疾病。
在大部分组织里面,这个基因的表达只来源于父本 DNA。但是,该课题组发现在一些需要快速增长、或者和代谢相关的组织中,IGF2 的表达来源于父母双方的 DNA,并且母本的 DNA 的表达可以被一个灵长类特有的调控元件控制。
评审专家读完论文表示,能够获取超长的 DNA 片段(>100kb),并从中读取出如此多的信息,是该领域的一个重要进步。同时,IGF2 一直被作为教科书上经典的基因调控范式,故此次发现也为教科书增加了新的内容。
此外,这一技术也是 CRISPR-Cas9 基因编辑技术、表观遗传学以及第三代测序的完美结合。值得一提的是,在分析很多基因组数据的时候,科学家发现总有些区域信息缺失,而这篇论文指出了其中的一个原因是主流基因组(hg38)组装存在着错误。
近日,相关论文以《动态、组织特异性和等位基因特异性调控元件的长程定相》()为题发表在 Nature Genetics 上 [1],索菲亚·巴塔利亚(Sofia Battaglia)、Kevin Dong、吴婧怡担任共同一作,哈佛医学院细胞生物学教授布拉德利·伯恩斯坦()担任通讯作者。
相关论文(来源:Nature Genetics)
03
CRISPR-Cas9 基因编辑技术、表观遗传学以及第三代测序的完美结合
研究中,该团队首先要在方法上实现本次技术,然后要建立分析数据的方法,最后则要使用本技术发现新的生物学机制。
吴婧怡表示:“在第一个阶段,我们当时面临的两个主要技术难点:一是如何提取和保留超长片段的 DNA,二是如何富集我们感兴趣的区域进行深度测序。”
为解决第一个问题,该团队在实验上尝试了很多条件,最后发现要保留长片段的 DNA,主要得在整个 DNA 处理的过程中保持一种温和的条件,以避免那些会打断 DNA 过程的剧烈物理震动和剪切。
为了富集 DNA,该团队使用 CRISPR-Cas9 技术,对去磷酸化的 DNA 进行剪切。这样一来,只有剪切后的 DNA 能连接上测序的接头,从而实现特定区域的富集。
当此次技术被成功开发出来后,他们开始进行大量测序。在第二阶段的数据分析中,遇到的最大难题则是信噪比的困扰。
“我们最感兴趣的是基因表达调控蛋白和 DNA 的结合位点。但是,一些核小体包裹的 DNA 附近也会出现假信号。后来,通过蛋白结合和核小体包裹的 DNA 在长度上的不同特征,成功地对数据进行去噪,最终拿到了理想数据。”吴婧怡说。
有了成熟的技术和分析管线后,接着要用它研究一些调控位点。当时,他们发现可以通过 DNA 的序列信息和调控蛋白的结合信息来区分父母本。因此,首选的研究位点是基因印记位点。
基因印记位点,是一些 DNA 上带有化学修饰的位点。这种化学修饰通常只修饰来源于父母本一方的 DNA 并调控其表达。而本次技术则能清晰地区分 DNA 的父母本来源,并能鉴定印记基因中已知和未知的调控元件。
但是,当他们深入探索 IGF2 这个经典印记基因区域的时候,发现 ENCODE 数据库的所有数据都是缺失的。随后,大家的兴趣开始转向别的基因。
吴婧怡说:“当时因为另外一个课题的原因,我的基因组浏览器里面保留了基因组大片段拷贝的注释。当我在浏览 IGF2 这个区域时,发现这个区域的缺失和一个大片段拷贝的注释是完美重合的,这意味着基因组上有多个 IGF2 基因,所以数据库的数据比对到了基因组另外的地方。”
后来,她和所在团队利用三代测序的数据重新组装了这一区域,发现这其实是利用短序列组装基因组时的一个错误。基因组上并没有其他的 IGF2,这意味着所有 ENCODE 数据又能重新回到正确位置。
当第一次看到这个区域上的信息时,大家都非常兴奋。而当所有数据都比对到 IGF2 附近以后,IGF2 的下游出现了一个增强子(基因调控元件)的信号。
“我当时看这些信号时感到很诧异,这里怎么会有增强子信号?IGF2 的增强子应该在 H19(另外一个基因) 的下游。” 吴婧怡对这个数据提出了质疑。
她的质疑,引起了团队其他成员的重视。接下来半年,大家的主要工作内容基本就在回答上述问题。期间,他们分析了 ENCODE 数据库的 200 多个细胞系、以及 GTEx 数据库的 17000 多个人体组织数据,并通过功能实验证明这是一个在快速增殖的细胞中调控 IGF2 的增强子,而且是灵长类动物中特有的。
而之前的大部分研究都集中在小鼠中,但是人类基因组在这里又出现了错误。可以说,这个增强子就这样一直默默地在基因组中发挥作用,但却没有被人发现。
(来源:Nature Genetics)
吴婧怡在博后期间还有一个新发现:即在肿瘤中有一些 DNA 重复序列,会在异常情况被表达出来,进而被呈递到肿瘤细胞表面,将肿瘤在体内的位置暴露出来,让人体免疫细胞能够识别到它们,并对肿瘤予以清除[2]。
但是,这种重复序列在基因组上有多重拷贝、而且也很难定位,这阻碍了学界对其激活机制的研究。
她说:“前人和我们自己目前的工作都显示在肿瘤中存在一些非常特异的激活型 DNA 重复序列。但是,对于它们激活的原因目前尚不清楚。
成立独立实验室以后,我们的第一个工作将利用本技术,来定位这些 DNA 重复序列和它的蛋白结合模式。如能掌握这些 DNA 重复序列的表达模式,将有望为药物设计或肿瘤疫苗提供新思路。”
参考资料:
1.Battaglia S,Dong K,Wu J,et al.Long-range phasing of dynamic, tissue-specific and allele-specific regulatory elements.Nat Genet 54,1504-1513(2022). https://doi.org/10.1038/s41588-022-01188-8
2.Griffin, G. K., Wu, J., Iracheta-Vellve, A., et al. Epigene C silencing by SETDB1 suppresses tumour intrinsic immunogenicity.Nature 595,309-314(2021).https://www.nature.com/articles/s41586-021-03520-4
声明:本站所有文章资源内容,如无特殊说明或标注,均为采集网络资源。如若本站内容侵犯了原著者的合法权益,可联系本站删除。