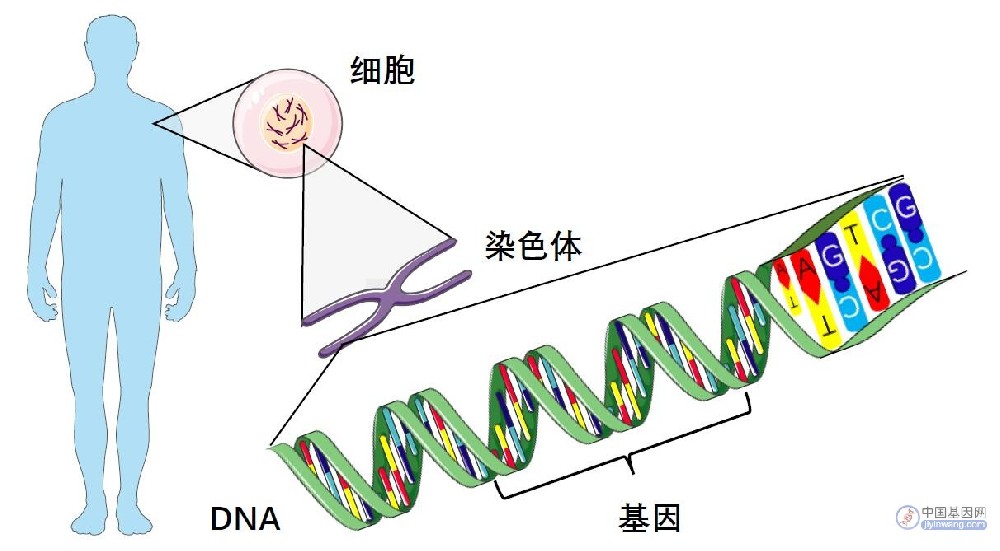
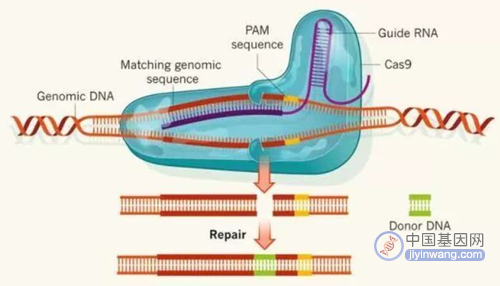
基因科普:基因测序技术发展史和技术原理,一文汇总!
从单个转录本到整个转录组
从单个生物到复杂的宏基因组
从模式生物到科学家们研究的所有物种
从浩浩荡荡的人类基因组计划到个体化基因检测
这一系列变化都离不开测序技术的发展
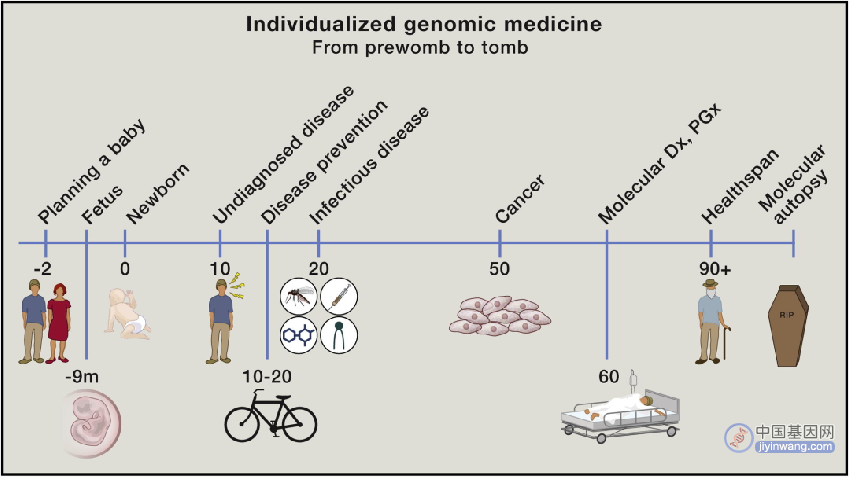
个体化基因检测在医学中的应用
工欲善其事,必先利其器。
测序技术的不断进步使人类可以破解生命的密码,是分子生物学迅速发展的强大推动力之一。
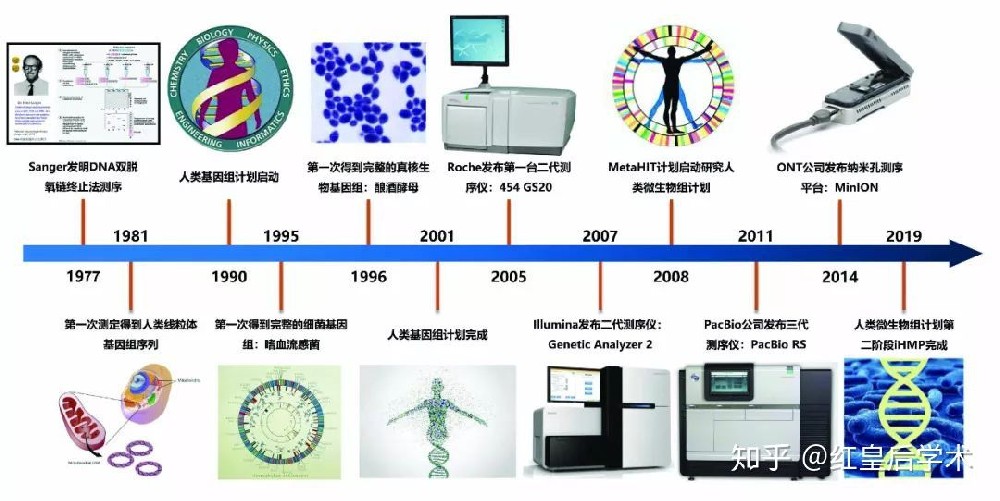
测序技术的发展历程
1952年
人们早已知道有两种核酸:DNA和RNA,但在很长一段时间里人们更倾向于蛋白质是遗传物质。
1944年,美国微生物学家艾弗里(Oswald Avery,1877-1955)和他的同事通过实验发现DNA是肺炎双球菌转化实验的关键,证实有活性的遗传物质是DNA。赫尔希(Alfred Hershey,1908-1997)和蔡斯(Matha Chase,1927-2003)通过同位素标记的T2噬菌体增殖实验,证实DNA才是遗传物质。这一实验结果很快被人们普遍接受,甚至被当成了DNA是遗传物质的最后证明。
1953年
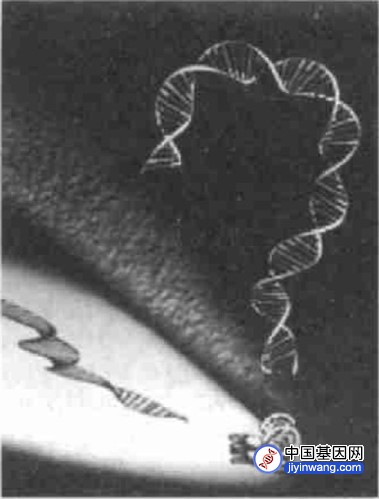
威尔金斯(Maurice Wilkins,1916 - 2004)和富兰克林(Rosalind Franklin,1920 - 1958)采用X-射线衍射技术分析DNA晶体,沃森和克里克通过一张清晰的DNA衍射照片找到了一种可能的DNA结构,并在Nature发文。
在这篇只有一页多的paper之中,沃森和克里克给出了改变整个生物界的一个图片:DNA双螺旋结构。沃森与克里克这篇仅1000字的论文被誉为“生物学的一个标志,开创了新的时代”。
这是在生物学历史上唯一可与达尔文进化论相比的最重大发现,它与自然选择一起,统一了生物学的大概念,是科学史上的一个重要里程碑,标志着分子生物学的诞生。1962年,沃森、克里克、威尔金斯三人因DNA双螺旋结构的发现而获得当年的诺贝尔生理学或医学奖。遗憾的是富兰克林已于1958年因乳腺癌去世。
1965年
酵母丙氨酸转移核糖核酸(tRNA)具有完全的生物活性,既能接受丙氨酸,又能将所携带的丙氨酸参入到蛋白质的合成体系中,因此在蛋白质生物合成中有着重要作用。
tRNA由76个核苷酸组成,其中除了4种常见的核苷酸外,还有7种稀有核苷酸。美国科学家霍利(Robert Holley,1922-1993)等人确定了第一个完整的核酸序列,即来自酿酒酵母的丙氨酸tRNA。霍利与尼伦伯格、科拉纳共同获得了1968年诺贝尔生理学或医学奖。
1972年
比利时分子生物学家Walter Fiers(1931-2019)测定了人类史上第一个完整的编码蛋白质的基因序列,即噬菌体MS2的衣壳蛋白序列,并于1976年获得了噬菌体MS2的完整基因组。
一代测序技术
1977年,DNA测序技术进展有了重大突破,英国化学家桑格(Frederick Sanger,1918-2013)发明了双脱氧链终止法,吉尔伯特(Walter Gilbert,1932-)和他的学生Allan Maxam(1942-)发明了化学降解法,这两种测序方法都被称为一代测序技术。
(1)化学降解法:即Maxam-Gilbert化学降解法,其原理为:将DNA片段的5’端磷酸基使用放射性同位素标记,再分别采用不同的化学试剂处理修饰和裂解特定碱基,从而产生一系列长度不一而5’端被标记的DNA片段,这些以特定碱基结尾的片段群通过聚丙烯酰胺凝胶电泳分离,再经放射线自显影,确定各片段末端碱基,从而得出目的DNA的碱基序列。
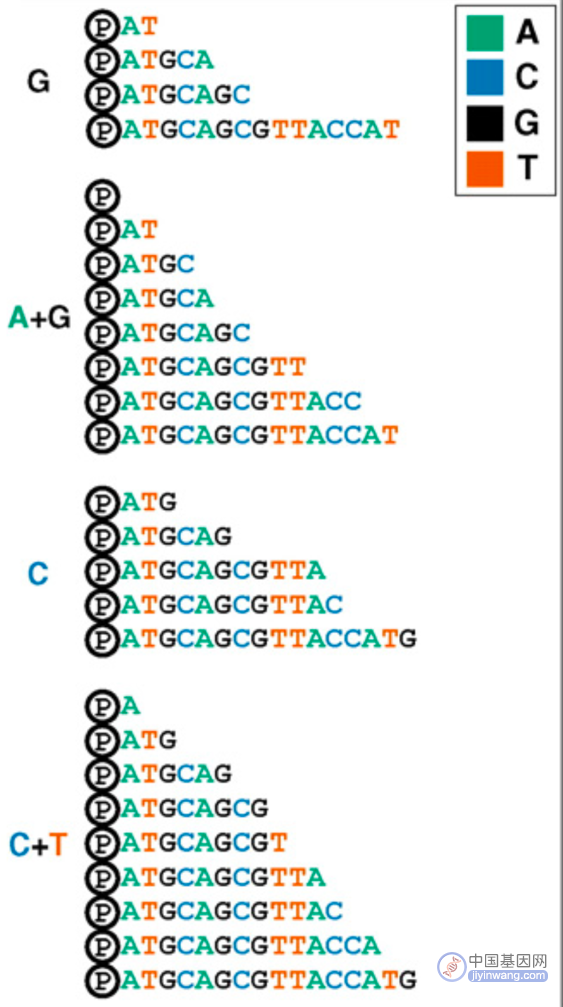
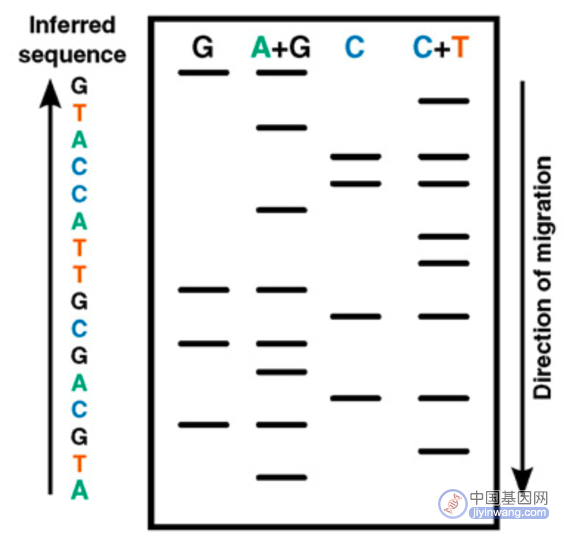
化学降解法测序原理
(2)双脱氧链终止法:即Sanger法,采用DNA复制原理。Sanger测序反应体系中包括目标DNA片段、脱氧三磷酸核苷酸(dNTP)、双脱氧三磷酸核苷酸(ddNTP)、测序引物及DNA聚合酶等。其技术核心是ddNTP的使用,由于缺少3’-OH基团,不具有与另一个dNTP连接形成磷酸二酯键的能力,这些ddNTP可用来中止DNA链的延伸。此外,这些ddNTP上连接有放射性同位素或荧光标记基团,因此可以被自动化的仪器或凝胶成像系统所检测到。
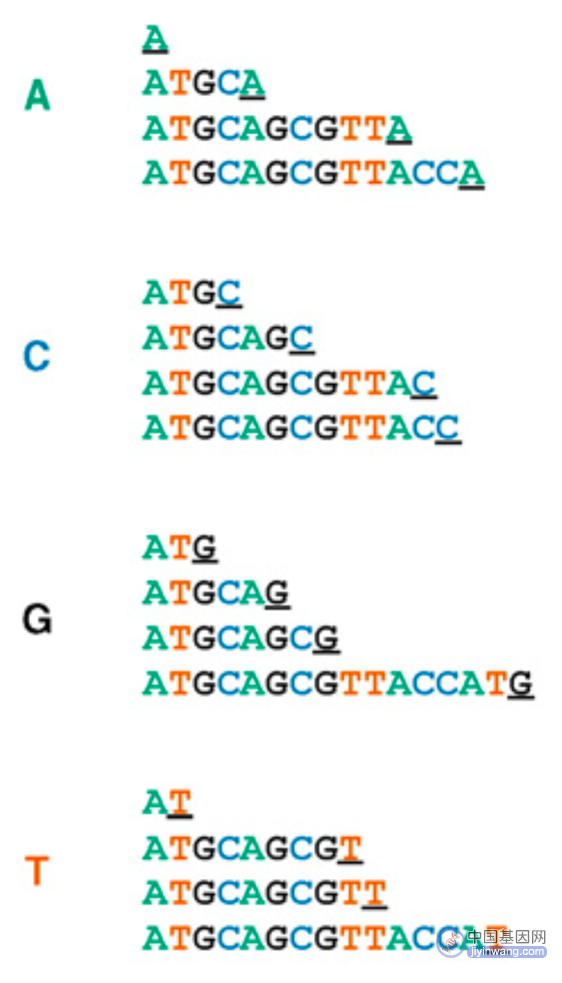
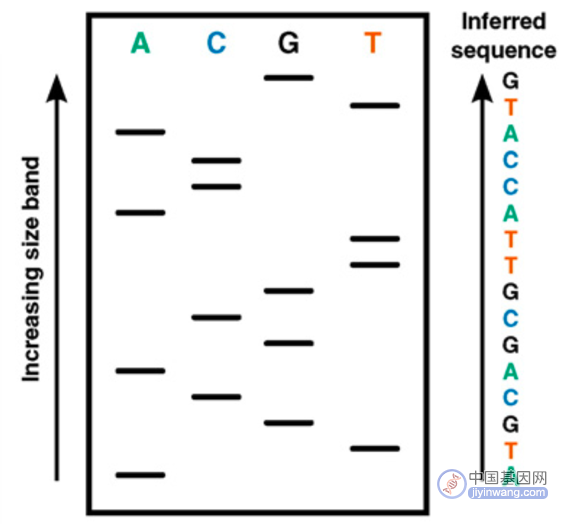
双脱氧链终止法(Sanger法)测序原理
Sanger的双脱氧链终止法广为人知并且沿用至今,而化学降解法如今已被人逐渐遗忘。
双脱氧链终止法的特点是测序读长可达1000bp,准确性高达99.999%,但测序成本高,通量低等方面的缺点,严重影响了其真正大规模的应用。但由于高精度,一代测序仍然是现今基因检测的金标准,也是对新一代测序结果进行评估验证的主要手段。
而在当时,正是一代测序技术使得基因组的研究成为了可能,浩浩荡荡的人类基因组计划就是利用一代测序技术完成的,人类基因组草图于2001年发表,被认为是人类基因组计划成功的里程碑,比预计时间提前了几年。随着测序技术的不断进步,以及T2T联盟的科研组织的不断努力,2022年4月,《Science》杂志连发六篇特刊,刊登了人类迄今为止完整且无间隙的人类基因组测序结果,填补了人类基因组计划留下的最后8%的序列图谱空白。
Sanger曾经在1958年及1980年两度获得诺贝尔化学奖,是第四位两度获得诺贝尔奖,以及唯一获得两次化学奖的人。其第一次获奖是凭借定序胰岛素的氨基酸序列,证明蛋白质具有明确构造,而第二次获奖就是因为双脱氧链终止法的发明。利用这个技术他成功测定了噬菌体φX174的基因组序列。
Gilbert研究出测定DNA和RNA分子中核苷酸顺序的方法,获1980年诺贝尔化学奖。此外,他也创造了外显子(exon)与内含子(intron)两个名词,来区分mRNA分子中的两个部分。
二代测序技术
二代测序(NGS)技术基于大规模平行测序技术(massive parallel analysis,MPS)方法对大量的目的基因同时进行测序,二代测序平台主要采用的技术有以下三种:边合成边测序(sequencing by synthesis,SBS),连接法测序(sequencing by ligation,SBL,又名SOLiD)和半导体测序(Ion Torrent)。
常见测序技术与平台
Roche公司 454焦磷酸测序
2005年,454生命科学公司(454 Life Sciences)推出了革命性的基于焦磷酸测序法的超高通量基因组测序系统——Genome Sequencer 20 System,开创了边合成边测序的先河。
2007年初,罗氏诊断(Roche Diagnostics)收购454公司。Roche 454测序系统是第一个商业化运营二代测序技术的平台。
2013年,罗氏诊断宣布关闭454测序业务,并于2016年终止相关服务。
Illumina公司 Solexa/HiSeq/MiSeq/NextSeq测序
Illumina公司的二代测序仪应该说是目前全球使用量最大的第二代测序机器。
ABI公司 SOLiD测序
SoLid测序技术是ABI公司于2007年开始投入用于商业测序应用的仪器,目前该平台已淡出市场。
华大基因 Complete Genomics/DNBSEQ测序
华大基因于2013年收购美国Complete Genomics(CG)公司,获取了其测序核心技术和其他核心知识产权,随后自己组建研发团队,不断增强自身实力。
Ion Torrent 半导体测序
2007年,454创始人罗森伯格创办Ion Torrent公司。
2010年,Ion Torrent推出了世界上第一台半导体测序仪——个人染色体检测仪PGM。
2010年,Life Technologies收购了Ion Torrent。2012年,Life公司再接再厉推出了功能更为强大的Ion Proton测序仪。
2013年,赛默飞世尔科技公司(Thermo Fisher Scientific)收购Life Tech。
(1)Roche 454焦磷酸测序:使用边合成边测序技术,避免了Sanger法存在的宿主菌克隆问题。
即首先将目的DNA片段打断成300-800bp的小片段,然后在5’端加上一个磷酸基团,并将3’端变成平端,再在两端加上衔接子组成目的DNA的样品文库。
之后将目的DNA片段固定到磁珠上,将磁珠包被在单个油水混合小滴中进行独立的扩增,从而实现所有目的DNA片段进行平行扩增PCR。
随后将这些DNA放入PTP反应板中共进行后继测序,这里面包含了化学光反应所需的各种酶和底物。
测序开始时,将T、A、G、C按顺序循环单分子进入PTP 板,如果发生配对,则会释放一个焦磷酸盐分子,其在后续与ATP磷酸化酶和虫荧光素反应产生光信号,此光信号被捕获以确定碱基序列。
(2)Illumina Solexa合成测序:使用克隆单分子阵列技术。
首先将目的DNA片段打断成100-200bp,随机连接到固相基质上,经过Bst 聚合酶延伸和甲酸胺变性的桥式PCR循环,生成大量的DNA簇。
之后的反应与Sanger法类似,每次延伸所产生的光信号被标准的阵列光学检测系统分析测序,下一次循环中把终止剂和荧光标记基团裂解掉,然后继续延伸dNTP,实现了边合成边测序技术。
(3)ABI SOLiD连接法测序:
首先制备DNA文库,可以使用片段文库和配对末端文库。第二阶段与焦磷酸测序相同,加入磁珠等反应元件进行emPCR平行扩增,不同的是该方法的磁珠只有1 µm。
在连接测序中,底物是 8 个碱基的八聚体单链荧光探针,在5′末端分别标记了CY5、Teaxs Red、CY3、6-FAM这四种颜色的荧光染料。3′ 端的第 1、2 位碱基类别排序分别对应着一个固定的荧光染料,第 3、4、5 位碱基“n”是随机碱基,第 6、7、8 位碱基“z”是可以和任何碱基配对的特殊碱基。一次测序中包括了五轮连接反应,可以减小测序误差。
(4)华大基因Complete Genomics测序:
华大智造测序仪采用先进的DNBSEQ测序核心技术,通过仪器气液系统先将DNA纳米球(DNA nanoball,DNB)泵入到规则阵列芯片(Patterned Array)并加以固定,然后泵入测序模板及测序试剂。
测序模板与芯片上的 DNB 的接头互补杂交,在DNA聚合酶的催化下,测序模板与测序试剂中的带荧光标记的探针相结合。
然后由激光器激发荧光基团发光,不同荧光基团所发射的光信号被相机采集,经过处理后转换成数字信号,传输到计算机进行处理,可以获取待测样本的碱基序列信息。
(5)2.5代测序技术/半导体测序技术:半导体测序技术由Ion Torrent研制开发。由于使用到了Emusion PCR技术,其实质介于二代和三代测序技术之间。
该技术使用一种高密度半导体芯片,每个芯片单独的进行测序。
实验时先将芯片置于一个离子敏感层和离子感受器之上,当DNA聚合酶在每一个单分子模板链上滑动时,发生聚合反应,释放出氢离子,最终离子感受器就会捕捉到这种信号,从而读出DNA序列。
三代测序技术
常见测序技术与平台
单分子荧光测序技术
美国Helicos BioSciences公司的SMS技术
美国Pacific Bioscience公司的SMRT技术
纳米孔测序技术
英国Oxford Nanopore Technologies公司
电子显微镜观察法
美国ZS Genetics公司
美国Electron Optica公司
(1)单分子荧光测序技术:代表是美国螺旋生物(Helicos BioSciences)(该司已停止营业)的SMS技术和美国太平洋生物(Pacific Bioscience)的SMRT技术。其通过将脱氧核苷酸用荧光标记,实时地记录荧光的强度变化。当荧光基团被掺入DNA链的时候,它的荧光就同时能在DNA链上探测到。当它与DNA链形成化学键的时候,它的荧光基团就被DNA聚合酶切除,荧光消失。这种荧光标记的脱氧核苷酸不会影响DNA聚合酶的活性,并且在荧光被切除之后,合成的DNA链和天然的DNA链完全一样。
测序过程包括文库构建和上机两步。文库构建是将长片段DNA分子与测序接头连接成茎环结构,然后加上与接头互补的测序引物及DNA聚合酶。上机测序是将构建好的文库复合物载入SMRT Cell的纳米孔中,通常一个纳米孔固定一个DNA分子,DNA聚合酶通过共价连接的方式固定在纳米孔底部。
(2)纳米孔测序技术:Nanopore测序是基于电学的检测,区别与Illumina和PacBio的光学。
代表是英国牛津纳米孔(Oxford Nanopore Technologies,ONT)公司。新型纳米孔测序法(nanopore sequencing)是采用电泳技术,借助电泳驱动单个分子逐一通过纳米孔来实现测序的。
由于纳米孔的直径非常细小,仅允许单个核酸聚合物通过,四种核苷酸的空间构象不一样,因此当它们通过纳米孔时,所引起的电流变化不一样。由多个核苷酸组成的DNA或RNA链通过纳米孔时,检测通过纳米孔电流的强度变化,即可得到碱基序列。
纳米孔测序仪MinION是现在最便携的测序仪,最小的版本和人的手掌差不多大,可以直接插在电脑上进行使用。ONT公司将MinION称之为世界上唯一的实时(real-time)测序仪。判断通过纳米孔的核苷酸类型,从而进行实时测序。
(3)电子显微镜观察法:2012年,首次报道通过电子显微镜鉴定完整DNA分子的碱基结构。
ZS Genetics(该司已停止营业)使用透射电子显微镜(TEM)通过检测标记的重金属标签观测DNA分子,但当重金属标签相距较近时很难分辨并会相互影响,而且透射电子显微镜发射的高能电子束易破坏DNA分子结构。
Electron Optica公司使用低能量电子显微镜LEEM,可以减少对DNA分子结构的破坏,且不使用重金属标签,在测序时发生的错误较少,但获得的图片分辨率低。
各代测序技术的优缺点
一代测序
优点:方法简便,分辨率高,测序片段长,流程细致,质控环节多,污染低,结果直观可视,假性结果极低。在单个基因测序方面具有优势。金标准。
缺点:测序试剂昂贵,通量低,处理比较长的同聚物时也有自身的问题。
二代测序
优点:通量高,一次可对几十万到几百万条核酸分子进行序列测定;单条序列成本低。
缺点:序列读长短;建库过程中有PCR过程,会引入错配碱基;要想得到准确和长度较长的拼接结果,需要较高的测序覆盖率,会增加成本和错误率。
三代测序
优点:
连贯性:连贯性对基因组的组装非常重要,如果连贯性比较好,能够准确的反应出基因结构之间的关系(外显子、基因簇、转移元件、调节序列等)。
完整性:如果一个基因组的测序深度>50X,理论上每一个碱基都会被测到。但实际上,基因组仍然会有很多缺失区域,比如即便是最新的人类参考基因组,其中仍然会有超过百万的“N”。读长的提升能够有效提高基因组组装的完整性。
准确性:基因组组装的准确性可以在核酸水平或者结构变异水平进行描述。Illumina的三代测序技术的准确性非常高,每个碱基准确性大于99.9%,PacBio和Nanopore的准确性在足够测序深度的情况下,经过算法校正之后也能够达到99.9%。
缺点:单读长的错误率偏高,需要重复测序以纠错;依赖DNA聚合酶的活性;成本较高;生信分析软件不够丰富,数据积累少。
纳米孔测序
优点:
超高的便携性,MinION可以做到即插即用,非常适合流行病疫区的即时检测;
超长的读长,MinION可以达到150kb,可用来填补之前测序的gap,可用于从头组装基因组,可变剪切等;
广泛的应用,可以直接对DNA,RNA和蛋白质序列进行测序;碱基判读准确率较高,R10纳米孔数据质量值超过Q40(即错误识别的概率是0.01%,即错误率0.01%),一致性(Identity)质量值达Q50 (99.999%的碱基准确率)。
缺点:低准确度,尤其是2D reads,虽然可以通过降低DNA通过速度或一致性检测提高准确度,但仍无法和二代测序相比
总结与展望
2004年至2010年间的测序能力每五个月翻一番,测序成本以超摩尔定律下降。
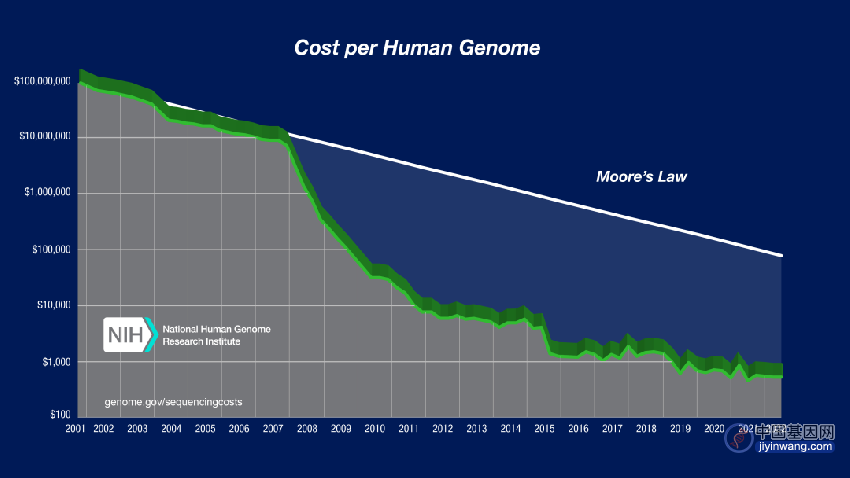
测序成本以超摩尔定律下降
未来测序将更加专一化,具有更高的通量,测序设备微型化、高度集成,测序数据分析能力进一步提高,测序对象范围扩展,测序成本更低。测序技术以及平台的研究还有很多问题需要解决,相信未来在各应用领域将对人类产生更大的作用。
References:
Heather JM, Chain B. The sequence of sequencers: The history of sequencing DNA. Genomics. 2016 Jan;107(1):1-8.
Topol EJ. Individualized medicine from prewomb to tomb. Cell. 2014 Mar 27;157(1):241-53. doi: 10.1016/j.cell.2014.02.012.
Molecular Structure of Nucleic Acids: A Structure for Deoxyribose Nucleic Acid
https://www.jianshu.com/p/926d02f94c78
https://blog.csdn.net/u010608296/article/details/111475760
https://www.genome.gov/about-genomics/fact-sheets/Sequencing-Human-Genome-cost#:~:text=Based%20on%20the%20data%20collected%20from%20NHGRI-funded%20genome-sequencing,generate%20a%20whole-exome%20sequence%20was%20generally%20below%20%241%2C000.
https://ibook.antpedia.com/x/477335.html
声明:本站所有文章资源内容,如无特殊说明或标注,均为采集网络资源。如若本站内容侵犯了原著者的合法权益,可联系本站删除。