人类基因组进化最快的区域也参与塑造人类对特定疾病的易感性
直立行走、颅面形态和认知能力等表型可以把我们和我们的类人猿祖先加以区分,但膝关节炎、精神分裂症等人类特有疾病易感性也随之而来。最近的工作指出人类和黑猩猩在氨基酸序列上几乎没有差异,因而人们假设差异主要存在于非编码区【1-3】。人类加速进化区(human accelerated regions,HARs)是一组在人类基因组中高度保守的DNA片段,不偏码蛋白,但其序列改变会影响基因表达【4】。然而,局部突变率和阳性选择的变化通常被认为是HARs产生的两种相互排斥的解释,那么究竟是两种原因单独的作用还是协同作用的结果尚不清楚,或许可以将人群遗传数据整合到比较基因组工作中来解决这一问题。

2022年11月23日,来自杜克大学医学中心的Craig B. Lowe团队在Cell杂志上发表了一篇题为Adaptive sequence divergence forged new neurodevelopmental enhancers in humans的文章,他们将比较基因组学与来自人群的遗传变异数据相结合,以证明人类基因组中进化最快的区域,称之为“人类祖先快速进化区(human ancestor quickly evolved regions,HAQERs)”。HAQERs富含二价结构域,与时空受限的发育或环境响应调节元件相关。研究人员开发了体内单细胞自转录活性调节区测序(scSTARR-seq)对发育中小鼠大脑皮层进行多重单细胞增强子测定,以证明HAQER锻造了人科动物独有的功能元件。此外,HAQERs还富含与疾病相关的变异,这表明它在塑造人类特定的疾病易感性方面发挥着重要作用。

研究人员通过一种计算筛选来识别人类谱系中进化最快的区域,通过对类人猿的全基因组多重比对以推算人类-黑猩猩祖先在每个比对位置的每个核苷酸状态的概率,确定了能将人类和祖先基因组分开的1581个平均长度为892 bp的HAQER,这些区域先前未被表征,代表了人类基因组中进化最快的区域。由于基因组区域中的序列差异可以通过局部突变率的变化或通过正选择产生,作者使用可用的高覆盖人群测序数据来确定这些因素在HAQER进化中的相对影响,发现HAQERs 中的序列进化是由尼安德特人和智人“分道扬镳”之前的突变率升高和定向正选择驱动的。
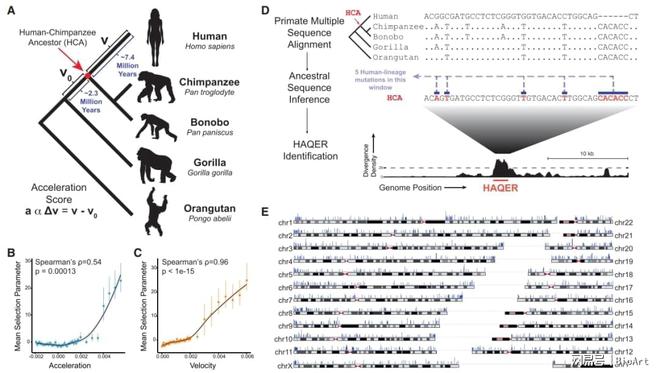
图1. 鉴定HAQERs是人类基因组中进化最快的区域
HAQERs通过定向正选择进化的结论暗示了这些区域的适应功能,通过分析127个参考表观基因组中染色质状态的全基因组富集和缺失模式,作者发现HAQER强烈富集二价染色质状态,即含有多梳抑制标记H3K27me3和活性启动子标记 H3K4me3和活性增强子标记H3K4me1。在HAQER中观察到的差异可能是发育基因调控功能进化的基础,虽然发育中组织的跨物种表观基因组图谱尚未广泛获得,但由于发育中的大脑皮层与人类认知有关,作者着重将这一部位作为HAQER 功能体内分析的目标组织。于是作者基于之前的方法【5】开发并利用了scSTARR-seq以同时测量多个测试序列在体内发育中脑组织中特定于每种细胞类型的增强子活性,结果显示测试的13个HAQER序列中有5个在至少一种细胞类型中显示出人源序列中增强子活性的显着增加,比如HAQER0710 在兴奋性神经元中表现出人源特异性增强子活性。
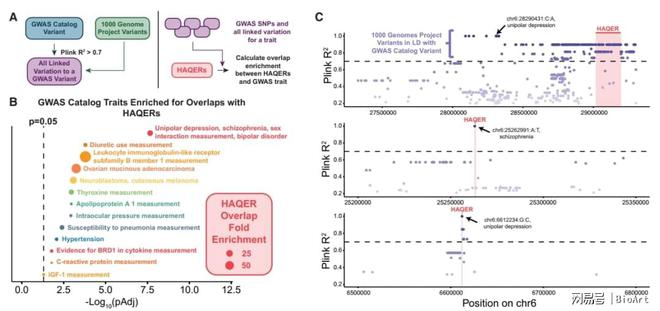
图2. HAQERs在与人类疾病相关的基因变异附近富集
为了确认HAQERs是否与疾病之间存在相关性,作者进一步通过GWASs计算了HAQERs的分离变异是否与人类疾病和障碍相关的SNPs相关。作者对与注释性状相关的所有GWAS SNPs执行计算(具体见文章方法部分),以获得特定GWAS性状的所有观察到的连锁变异集。结果显示HAQER 高度富集与GWAS特征相关的变异,如高血压、神经母细胞瘤和单相抑郁症/精神分裂症/双相情感障碍。因此,HAQER与疾病变异的关联表明快速分化产生了人类特异性疾病易感性作为结果。
总之,这项工作确定了整个人类基因组中进化最快的区域,并推测在整个脊椎动物进化史中,突变率、正选择和物种特异性疾病易感性之间存在一般对应关系。
https://doi.org/10.1016/j.cell.2022.10.016
参考文献
1. King, M.C., and Wilson, A.C. (1975). Evolution at two levels in humans and chimpanzees.Science188, 107–116. https://doi.org/10.1126/science.1090005.
2. Wray, G.A. (2007). The evolutionary significance of cis-regulatory mutations.Nat. Rev. Genet.8, 206–216. https://doi.org/10.1038/nrg2063.
3. Carroll, S.B. (2008). Evo-devo and an expanding evolutionary synthesis: a genetic theory of morphological evolution.Cell134, 25–36. https://doi.org/10.1016/j.cell.2008.06.030.
4. Pollard, K.S., Salama, S.R., King, B., Kern, A.D., Dreszer, T., Katzman, S., Siepel, A., Pedersen, J.S., Bejerano, G., Baertsch, R., et al. (2006). Forces shaping the fastest evolving regions in the human genome.PLoS Genet.2, e168. https://doi.org/10.1371/journal. pgen.0020168.
5. Doan, R.N., Bae, B.-I., Cubelos, B., Chang, C., Hossain, A.A., Al-Saad,S., Mukaddes, N.M., Oner, O., Al-Saffar, M., Balkhy, S., et al. (2016). Mutations in human accelerated regions disrupt cognition and social behavior.Cell167. 341.e12–354.e12. https://doi.org/10.1016/j.cell.2016.08.071.
声明:本站所有文章资源内容,如无特殊说明或标注,均为采集网络资源。如若本站内容侵犯了原著者的合法权益,可联系本站删除。