全基因组序列分析注释自闭症的基因组结构
自闭症谱系障碍(ASD)是一种神经发育疾病,主要表现为社交和沟通困难、重复行为和兴趣受限,男性发病率大约是女性的4倍,针对双胞胎的研究认为遗传率在64%–91%之间,其在遗传上也是异质的,涉及到大量基因。之前的WGS已经评估了不同类型突变体在疾病中的贡献,但通常一次只分析一或几个类别,无法完全捕捉ASD的基因组结构。
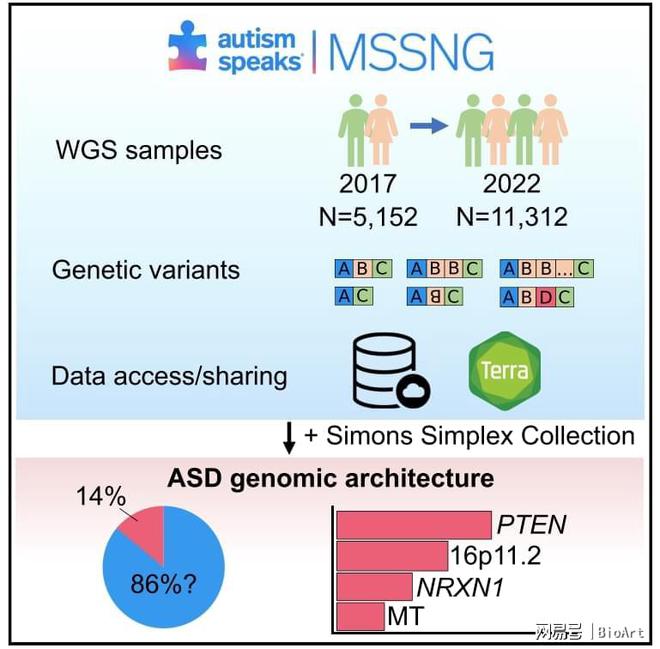
2022年11月10日,来自加拿大多伦多儿童医院的Stephen W. Scherer团队在Cell杂志上发表了题为Genomic architecture of autism from comprehensive whole-genome sequence annotation的文章,他们展示了包括来自 5100名ASD患者和6212名非ASD父母及兄弟姐妹WGS数据的Autism Speaks MSSNG资源最新版本,分析了来自MSSNG和Simons Simplex Collection(SSC)中的各种遗传变异,在MSSNG的718/ 5100名ASD患者 (14.1%) 和SSC的350/2419名ASD患者 (14.5%) 中发现了ASD相关的罕见变异,其中,52%是核序列水平变异,46%是核结构变异(SVs)(包括拷贝数变异、倒位、插入、单亲同二体和串联重复扩增),2%是线粒体变异。总之,这项研究对于探索携带ASD相关罕见变异的家庭的基因型-表型相关性,以及剖析特发性ASD人群的病因提供了重要资源。

与之前的版本【1】相比,当前MSSNG 包含两倍的ASD个体(5100对2613)和总个体(包括家庭成员;11312对5152)的WGS数据,研究人员合并了包括MSSNG、SPARK和自闭症测序联盟(ASC)等在内的多个队列的数据共鉴定到134个ASD相关基因。67个新基因中的大多数为从头蛋白质截短变体(PTV)、从头破坏性错(DMis)变体和过量PTV的混合。作者分别根据基于读取深度的工作流程(检测CNVs≥1 kb)和采用拆分读取和基于双端映射的算法(检测删除、重复、插入和倒位≥50 bp)将罕见SVs进行划分,在约6%的ASD 患者中检测到致病性SVs。
随后,作者评估了致病变异、单倍群和异质性以研究它们与ASD的关联。在ASD个体中发现了23例已知致病性变异,异质性>5%,其中17例为从头变异,定义为母体异质性不可检测或<5%,另外的4个家族中致病性变异在患有ASD的母亲和她的孩子中均存在>5%的异质性。作者比较来自MSSNG和SSC的ASD个体与来自SSC的非ASD同胞不同变异类型情况,从头PTV和DMis、PTV-PTV双等位基因事件在ASD个体中显著富集。为了检查基因型-表型关联,作者比较了适应性行为标准评分、FSIQ、全球能力共识估计和社会化标准评分,几乎所有类别ASD相关罕见变异都与这四种检测的较低分数相关。
为了详细了解基因组结构,作者对受影响的基因区域进行分析,最常受ASD患者显性PTV(从头和遗传)和DMi变体(仅从头)影响的基因包括PTEN、KDM5B、MIB1和CHD8。除了21三体和性染色体非整倍体外,还观察到其他几种染色体异常,包括多重易位。受SVs影响最大的ASD相关基因包括NRXN1、PTCHD1-AS和AUTS2,而最常受致病变异影响的mtDNA基因是MT-TL1和MT-ND6。随后,作者根据它们对增强子、启动子、拓扑关联域和其他调控元件的影响来注释罕见的序列水平变体。在MSSNG中,预测会损害启动子的变体被过度传播给患有ASD的个体。在SSC中,在患有ASD的个体或非ASD的兄弟姐妹中没有观察到过度传播。
除了罕见的遗传和从头基因变异外,常见的变异也有助于ASD的遗传力【2】。作者使用最近ASD WGS的汇总统计数据来计算MSSNG、SSC的多基因风险评分(PRS)。为了评估技术再现性,作者比较了来自MSSNG的10对同卵双胞胎的PRS。相对于整个PRS范围,所有双胞胎之间的差异都很小。MSSNG、SS中的PRS分布相似。调整性别后,MSSNG和SSC中的ASD个体的平均PRS高于SSC中的非ASD同胞。PRS与上述共识表型测量之间未检测到显著关联。最后,该团队也建立了一个供研究人员协作使用的基于云的数据平台Terra(https://terra.bio),由于MSSNG数据已经存储在GCP中,因而Terra非常适用于探索基于云的ASD研究。
总之,这项工作对MSSNG的分析加强了对之前ASD基因组结构的认识并提出了新见解。随着越来越重视基因型表型相关性,个体水平的理解将是早期诊断的关键,这可能通过允许早期治疗来改善结果,这项研究产生的分子靶点也可能最终为药物干预提供信息。
https://doi.org/10.1016/j.cell.2022.10.009
参考文献
1. Yuen, R.K.C., Thiruvahindrapuram, B., Merico, D., Walker, S., Tammimies, K., Hoang, N., Chrysler, C., Nalpathamkalam, T., Pellecchia, G., Liu, Y., et al. (2015). Whole-genome sequencing of quartet families with autism spectrum disorder.Nat. Med.21, 185–191. https://doi.org/10.1038/nm.3792.
2. Gaugler, T., Klei, L., Sanders, S.J., Bodea, C.A., Goldberg, A.P., Lee, A.B., Mahajan, M., Manaa, D., Pawitan, Y., Reichert, J., et al. (2014). Most genetic risk for autism resides with common variation.Nat. Genet.46, 881–885. https://doi.org/10.1038/ng.3039.
声明:本站所有文章资源内容,如无特殊说明或标注,均为采集网络资源。如若本站内容侵犯了原著者的合法权益,可联系本站删除。