科学家开发三维人脑类器官筛选系统,将自闭症基因研究提至新高度
人的大脑宛如宇宙一样,复杂而神秘。我们的大脑发育究竟经历了怎样的过程?各种脑类疾病又是如何产生的?从大学起,李冲就对神经系统产生了浓厚的兴趣,并一直致力于疾病的致病机理研究。
最近,在奥地利科学院分子生物科技研究所从事博士后研究的他以共同第一作者和共同通讯作者在 Nature 上发表了一篇论文。
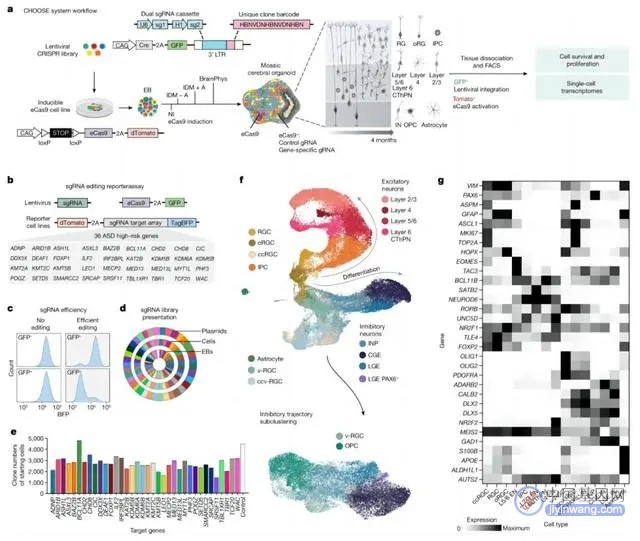
研究中,李冲和所在团队将高通量基因编辑 CRISPR/CAS9 技术和单细胞组学技术联合应用在 3D 人脑类器官中,开发出一种名为 CHOOSE(CRISPR-human organoids-scRNA-seq)的系统,并利用这一系统探索了自闭症(又名孤独症)的发病机制。

这一成果能帮助人们进一步理解自闭症可能的致病原因,加深对于自闭症致病基因的功能的认知。
同时,本次系统对于在类器官中进行高通量基因编辑和单细胞组学的联合应用,起到了重要的推进作用。
此外,这一系统也能用于不同的类器官系统里比如心脏、肺、小肠等,能够加速对于基因功能在复杂组织里的全面研究,并能加速人类对于致病基因功能的认识。
同样重要的是,类器官技术是一个以人类组织为平台的研究模式。相比以往在动物模型里进行研究,本次工作更能观察到一些人类特有的生物现象。
(来源:Nature)
01
发现与自闭症基因相关的细胞类型和调控网络变化
详细来说,CHOOSE 这一系统可以平行地在类器官中的每一个细胞中进行不同的基因敲除。假如将其想象成一种马赛克组织,组织中的每一个细胞都有一个特定的基因被编辑。
经过一段时间的培养和组织分化以后,就可以对类器官中每一个进行过基因编辑的细胞进行单细胞转录组测序。
单细胞转录组测序技术,是一种在近年来得到快速发展的基因组学技术。它可以全面、定量地针对细胞类型、细胞状态、细胞内的生物学通路,在单细胞水平上开展分析,从而提供大量的生物学信息。
在 CHOOSE 系统的帮助之下,可以一次性针对大量基因同时进行深入、全面的表型分析,从而大大加速对于致病基因的研究速度和研究深度。
研究中,利用 CHOOSE 这一系统课题组研究了自闭症的致病基因。据了解,自闭症的发病率非常高,而且是所有神经发育疾病中遗传因素较高的一种。
目前,人类对于自闭症的发病机理知之甚少。自闭症患者被确诊的时候往往在 3 岁以后,这时患者的大脑发育已经基本结束。
这时就无法知道自闭症患者的大脑在发育时到底发生了什么。不过,越来越多的动物模型证据表明,早期自闭症患者的大脑发育受到了影响。
目前已知的自闭症相关基因超过 1000 个,该团队从中选取 36 个基因。它们具有高度的致病性,并且和基因转录表达存在相关性。
随后,他们使用 CHOOSE 系统建立了一个 CRISPR 库,在干细胞里敲除这些基因(每一个细胞里只有 1 个基因被敲除)。之后,使用这些干细胞在体外培养类脑器官,分化成为大脑皮层组织。
四个月后,这些组织中已经分化出既复杂、又成熟的细胞类型(共有 16 种)。随后,他们对组织加以处理并进行单细胞测序。
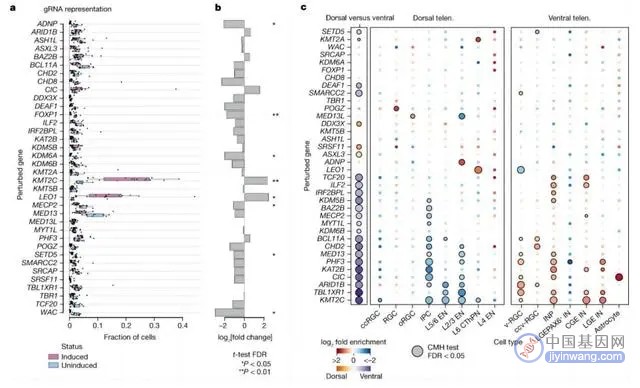
通过数据分析,他们发现了很多有意思的现象。首先,他们发现所有致病基因,并不都是通过同一种方式来影响大脑发育。
比如,当不同基因被敲除以后,可以影响不同的细胞类型。这说明自闭症的致病原因并不是只有一种。
同时,他们还发现在自闭症基因被敲除后,有些细胞类型更加容易受到影响。比如,当有多个基因被敲除后,一种中间神经干细胞(IPC,Intermediate progenitor cells)的百分比都会被降低。
这种 IPC 在人脑中具有重要作用,它可以分化成大脑皮层中不同层的兴奋神经元,并且可以参与到人脑皮层的扩大之中。
这也可能在间接地提示,这种人脑特有的发育过程也参与了人类脑疾病比如自闭症的发生。
之后,课题组利用单细胞测序的优势,进行了转录组的深入分析,描绘了每一个自闭症基因被敲除之后转录组发生的变化,并生成了强大的表型数据库,能够帮助人们更好地研究这些致病基因。
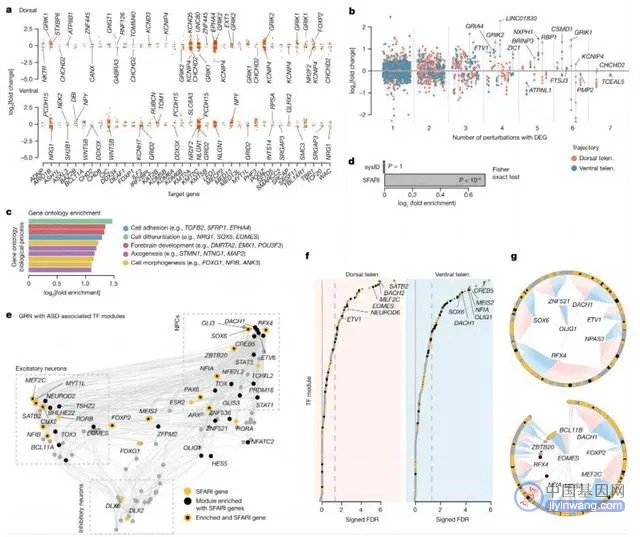
最后,他们又整合了单细胞染色质开放和转录组的情况,基于此开展了针对基因调控网络的分析,借此发现了一些和自闭症基因相关的调控网络的变化情况。
众所周知,人的大脑是最复杂的器官之一。那么,为什么李冲等人选择了这样一个课题?
(来源:Nature)
02
填补大脑研究的空白之一
的确,无论是研究人脑发育、研究人脑功能、还是研究脑相关疾病都是高难度的课题。其中一个重要原因就是对于发育中的人脑,人们始终无法在分子水平和细胞水平进行观察和分析。
目前,人们对于人脑发育的认知,大部分是从各种模式生物例如小鼠的研究中发现的。
但是,人脑发育有着一定的特殊性。很多时候在动物模型中的发现,不能很好地诠释人脑发育的现象,尤其是在研究人脑疾病的致病和发病机理方面。
为了克服这些困难,李冲的博士后导师——奥地利科学院分子生物科技研究所教授尤根·A. 诺布里克(Jürgen A. Knoblich)和团队,曾在 10 年前首次利用多功能干细胞在体外进行诱导,借此分化并培养出一种三维类人脑器官。
这些类脑器官对于研究人脑有着巨大优势,它们能很好地模拟早期人大脑的发育的过程,不仅呈现出体内发育的结构性特征,更能高保真地分化出很多细胞类型。
由此可见,类器官是一种可被用于模拟人脑发育的模型。而且,通过利用不同的分化因子,这些类脑器官可以特定地进行分化,从而诱导形成不同的脑区,以用于研究不同神经元的功能。
更重要地是,当把这一模型分化在不同脑细胞中,即可模拟整个体内发育的动态过程。这样一来,就能全面系统地在不同发育时期开展观察分析。
由于类器官在组织和细胞上都比较复杂,因此需要高分辨的研究手段。而且类器官往往需要长时间的体外培养(2-6 个月甚至更长),这给学界带来了巨大挑战。
目前,类器官已被广泛用于遗传型脑疾病的研究。但是,目前已知的致病基因有成百上千种,如果对每一致病基因逐个研究,不仅耗时耗力更重要的是限制了人们对于治病基因以及大脑发育的理解。
所以,迫切需要一种高通量、高分辨的定量方法,来加速我们对于基因在大脑发育中的功能研究,而本次工作则极大地填补了这一空白。
(来源:Nature)
03
培养一批类器官,耗时长达 4 个月
“不积跬步,无以至千里。”做科研更是如此,研究伊始对于开展长期体外培养、并在器官组织水平上进行高通量单细胞筛选分析,课题组其实并没有太多经验。
而类器官经过长期的分化,导致细胞类型比较多样。因此这个课题是一个非常复杂、非常精细的高风险项目。每一步都必须稳打稳扎,才能确保获得高质量的数据。
为此,他们在可行性上进行了大量的摸索优化,通过提高基因的编辑效率,优化类器官水平上的遗传学设计,以便保证基因原件在最终分化的组织中,能够有效地表达并且能被监测到。
同时,还需要针对实验设计进行反复推敲,确保最终有足够的样本进行有统计学意义的分析。此外,直到类器官发育到 4 个月的时候才能进行样本分析,所以很多验证性实验非常耗时。
对于如何选择要靶向的基因,该团队也进行了反复思考。由于他们对脑疾病的机理非常感兴趣,所以选择以自闭症为研究对象,着重研究一批高致病性致病基因。
因为这些基因的致病性在此前的大规模人群测序研究中已经得到确认,只是具体的致病机制不详。
由于不少实验都非常耗时,很多时候需要提前半年或提前更长时间进行规划,提前预约实验仪器设备、以及联系相关辅助人员。
当这些类脑器官发育 4 个月以后,他们开始对组织进行单细胞测序。在初步分析结果中,他们看到了一些很有意思的表型,比如很多基因的编辑会导致细胞命运的变化。
李冲等人对此感到特别兴奋。为了能够更加系统性地进行定量,他们和瑞士苏黎世联邦理工学院的同行开展合作。
通过这一合作,他们不仅在细胞类型组成上进行了全面的比较,也利用单细胞转录组的强大信息探索了下游信号通路变化。
同时,合作者也开始将他们刚刚开发的基因调控网络分析方法用到实验分析之中,借此得到了和自闭症相关的调控网络。
在利用生物信息学进行定量分析时,他们发现了一个有意思的现象:三个 BAF complex 的组成蛋白的敲除导致了相似的表型变化。
期间,他们一直思考在筛查中发现的现象,是不是也存在于真实世界之中?换句话说,本次研究能否为某些疾病的发生提供一些机制上的支持。
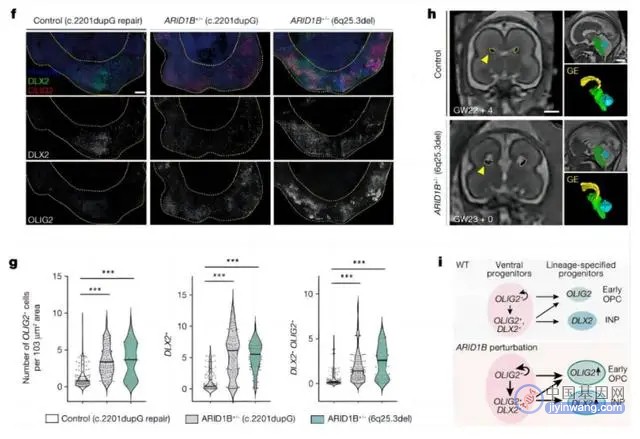
幸运的是,课题组找到了两位存在 ARID1B 突变(位于 6 号染色体的基因)的患者。于是,他们从患者身上提取体细胞,并重编程成为干细胞,再发育成为类脑器官。
后来,他们在这两名患者细胞发育的类脑器官里都看到了相似的表型,这让整个研究团队都非常兴奋。
因为他们在 CHOOSE 系统中筛查到表型和在病人样本中看到的是一致的,这再次印证了本次系统对于研究人类致病基因功能的可用性。
(来源:Nature)
最终,相关论文以《单细胞脑类器官筛查识别自闭症的发育缺陷》(Single-cell brain organoid screening identifies developmental defects in autism)为题发在 Nature[1]。

图 | 相关论文(来源:Nature)
李冲是共同第一作者兼共同通讯作者,奥地利科学院分子生物科技研究所教授尤尔根·A. 诺布里克(Jürgen A. Knoblich)和瑞士苏黎世联邦理工学院教授芭芭拉·特鲁特莱因(Barbara Treutlein)担任共同通讯作者。
参考资料:
1.Li, C., Fleck, J.S., Martins-Costa, C.et al. Single-cell brain organoid screening identifies developmental defects in autism. Nature 621, 373–380 (2023). https://doi.org/10.1038/s41586-023-06473-y
声明:本站所有文章资源内容,如无特殊说明或标注,均为采集网络资源。如若本站内容侵犯了原著者的合法权益,可联系本站删除。