Nat Commu:从人类基因测序直接预测表型:从头设计的全新体系
近年来高通量基因测序技术蓬勃发展使得个人基因测序数据爆炸式增加。然而目前基因分析手段仍然以相关性分析为主,系统性预测基因型对表型的影响仍然面临重要挑战,从基因型到表型解读亟须方法上的新突破。
2023年2月17日,英国剑桥医学研究协会分子生物学实验室(Medical Research Council Laboratory of Molecular Biology,MRC-LMB)的Julian Gough 课题组(第一作者:路畅)在Nature Communications 上发表研究论文Hypothesis-free phenotype prediction within a genetics-first framework【1】,描述了一个从头设计的全新分析体系Nomaly。不同于当前普遍应用的全基因组扫描强相关性位点的分析方法(比如GWAS),Nomaly通过整合分子生物学、生物信息学的研究结果和合适工具,从个人的基因测序结果对表型异常(可观测或可检测的特征和表型,包含疾病和组织层面的表型)进行直接预测。该分析体系包括从人类基因测序直接预测表型,以及对预测结果进行验证和评判两个主要模块。

以DNA测序结果出发,对组织层面的表型进行直接预测是人类基因密码解读的终极目标。然而直接预测的可行性以及可操作性仍不可知。此项工作花费十余年,创新性地从头设计了预测方法,对个人基因组中的变化进行系统性地整体分析,整合了多层次基因信息流(DNA突变 -> 氨基酸突变 -> 蛋白结构功能影响),整合了多个大型表型数据库注解信息(数万级别的表型和基因已知关系知识),系统性地对表型异常进行量化打分从而获得组织层面的表型预测。Nomaly体系应用到了三个独立数据集上,验证了这种ab initio的表型预测可以达到统计显著,实现对组织或疾病层面表型预测并加以潜在基因解释。Nomaly‘预测+解释’双重能力是相关性分析难以实现的。
文章中的首要数据集是由全世界范围内的已经拥有自己基因测序结果的志愿者组成:志愿者自愿上传他们自己购买的DNA测序结果,基于这些DNA数据进行预测,并基于此产生了关于表型的调查问卷请他们回答。为了建立对来源不同、DNA测序方法不同的上传数据的自动质量控制和处理管线,研究者对近七千完全公开的个人基因数据库(OpenSNP)进行分析,并将研究得到的预处理方法和结果公开【2】。另外,研究人员还在英国最大的患有发育疾病的孩子的数据集(Deciphering Developmental Disease, DDD)【3】上对预测成功率的显著性进行了测试。最后,对参与了全能干细胞项目(Human Induced Pluripotent Stem Cell Initiative, HipSci)【4】的人利用该法进行了细胞层面的表型预测,并通过细胞实验对其中一个与有丝分裂异常的预测结果进行了验证。
该体系中的预测方法以蛋白质的基本结构及功能单位 —— 蛋白结构域为核心建立:利用了经过数十年发展的蛋白结构域数据库(SUPERFAMILY database)【5】,利用了蛋白结构域的隐马尔可夫模型来对基因位点变化导致的氨基酸突变进行打分,并通过蛋白结构域语义注解(功能+表型)数据库(dcGO)【6】将人类基因组中的位点对应至上万种表型。预测体系通过对基因变化导致的功能变化做出差异程度打分,基于差异程度大则可能是相应表型异常的假设,对表型异常进行预测(outlier prediction)。
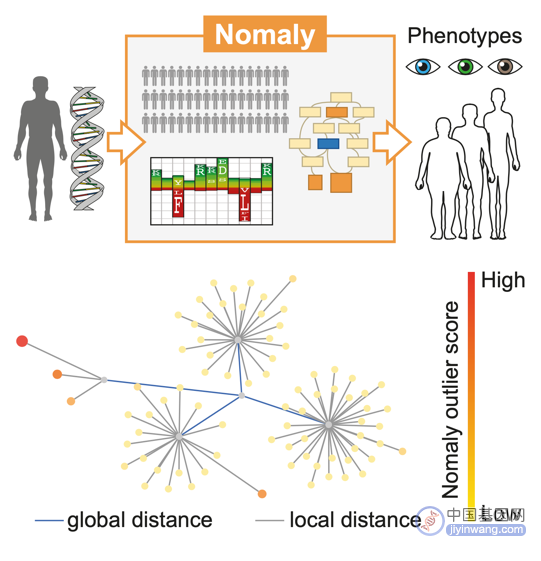
图注 – 预测方法(Nomaly)简介:以个人DNA测序结果为输入,将其与上千人组成的数据库比较,预测由DNA变化导致的异常表型。
这种通过“从头预测+验证预测显著性”从而获得具有生物意义致病基因解释的体系是基因分析领域的重要进展。在DNA测序方法爆发式发展、数百万人都有了全基因组测序数据的今天,测序结果的分析方法革新呼之欲出,以在这些不断增长的数据资源中更有效地发掘信息、获得更多的具有医疗价值的发现。进一步发展该体系,以及与其他方法进行结合,还将推进个人化精准医疗的研究。
原文链接:
https://doi.org/10.1038/s41467-023-36634-6
参考文献
1. Hypothesis-free phenotype prediction within a genetics-first framework. Lu, C., Zaucha, J., Gam, R., Fang, H., Smithers, B., Oates, ME., Bernabe-Rubio, M., Williams, J., Zelenka, N., Pandurangan, AP., Tandon, H., Shihab, H., Kalaivani, R., Sung, M., Sardar, AJ., Tzovoras, BG., Danovi, D., Gough, J. Nature Communications 14, 919 (2023).
2. Lu, C., Tzovaras, BG. & Gough, J. A survey of direct-to-consumer genotype data, and quality control tool (GenomePrep) for research. Comput Struct Biotechnol J 19, 3747–3754 (2021). (第一作者同时为通讯作者)
3. The DDD Study. Large-scale discovery of novel genetic causes of developmental disorders. Nature 519, 223–228 (2015).
4. Kilpinen, H. et al. Common genetic variation drives molecular heterogeneity in human iPSCs. Nature 546, 370–375 (2017).
5. de Lima Morais, D. A. et al. SUPERFAMILY 1.75 including a domain-centric gene ontology method. Nucleic Acids Res 39, D427–D434 (2011).
6. Fang, H. & Gough, J. dcGO: database of domain-centric ontologies on functions, phenotypes, diseases and more. Nucleic Acids Res 41, D536–D544 (2013).
声明:本站所有文章资源内容,如无特殊说明或标注,均为采集网络资源。如若本站内容侵犯了原著者的合法权益,可联系本站删除。