LncBook:人类长链非编码RNA(lncRNA)“百科全书”

随着对基因组认识的加深,有着“多技能”傍身的长链非编码RNA(Long non-coding RNA,lncRNA)被越来越多的科研人员和研究领域关注。很多小伙伴想着手lncRNA的研究工作,却在五花八门的lncRNA“神器”面前手足无措。
“想筛选在癌症中有潜在功能的lncRNA基因进行实验研究,有没有这样的数据库?”
“想获取小鼠中人类lncRNA的同源信息,哪个平台有详细注释?”
“获得了一系列lncRNA,如何筛选其中更可靠的对象做实验?”
“筛选了感兴趣的lncRNA,计划开展分子机制研究,如何能获取与它互作的miRNA和蛋白?”
“有没有一个库能把所有信息都涵盖呀”。
当然有啦!“一本”LncBook帮你解决lncRNA分析路上的各种困难。
LncBook(https://ngdc.cncb.ac.cn/lncbook/)是一个综合性人类lncRNA数据库,新发布的2.0版本包含了95243个lncRNA基因及323,950个转录本的高质量集合,并从多组学层面对这些lncRNA基因进行了全面注释,包括进化保守性特征、多生物学场景下的表达谱和DNA甲基化谱、疾病/性状相关变异、lncRNA-蛋白质相互作用、lncRNA-miRNA相互作用、小蛋白等,提供友好的检索、浏览、可视化、分析和下载服务(图1)。此外,部署了多个在线分析工具,包括ID转换工具、转录本编码潜能预测、基于BLAST的序列检索、根据基因组位置分类,帮助用户更高效地开展在线分析。
2.0版本的数据库文章以:LncBook 2.0: integrating human long non-coding RNAs with multi-omics annotations 为题于2022年11月在 Nucleic Acid Research 期刊在线发表。
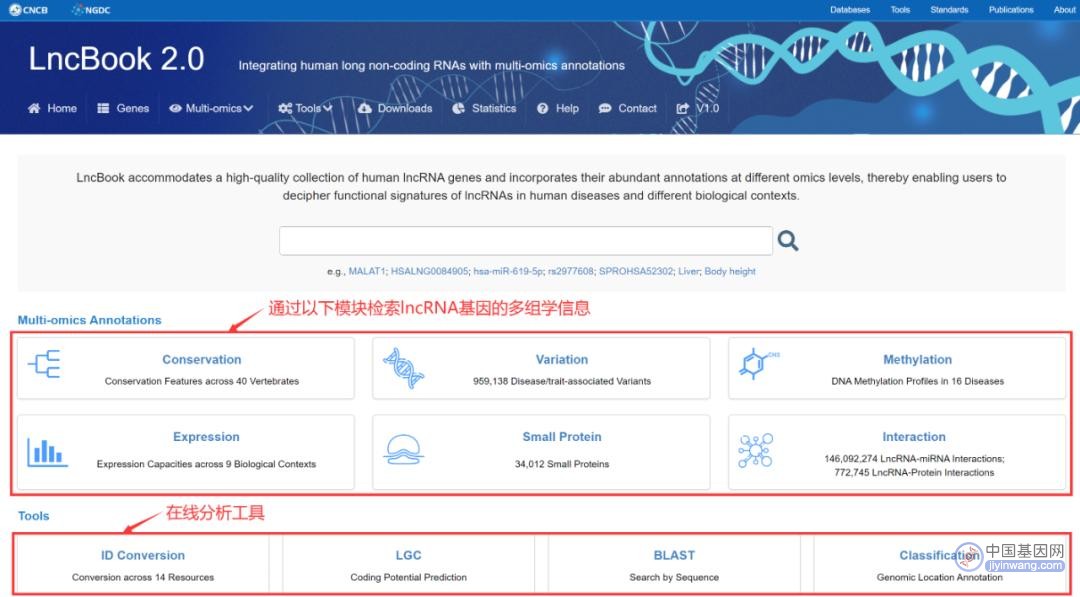
图1:LncBook主页面
了解了LncBook的“神通广大”,接下来,我们具体来看一下到底怎样使用它吧!
LncBook数据库的使用
一、获得全面的人类lncRNA参考数据集
LncBook先后整合了GENCODE、NONCODE、CHESS、LNCipedia、FANTOM-CAT、BIGTranscriptome等10余个数据库的lncRNA数据,通过严格的质控审编,提供全面高质量的人类lncRNA集合和参考文件。GTF和FASTA文件可在“Downloads”模块免费获取。此外,数据库还整合了lncRNA及GENCODE中的各类基因,形成更为全面的参考数据集,帮助用户更好地对人类基因组进行分析(图2)。更为方便的是,利用ID转换工具,可获取整合的任意库中该lncRNA对应的ID。
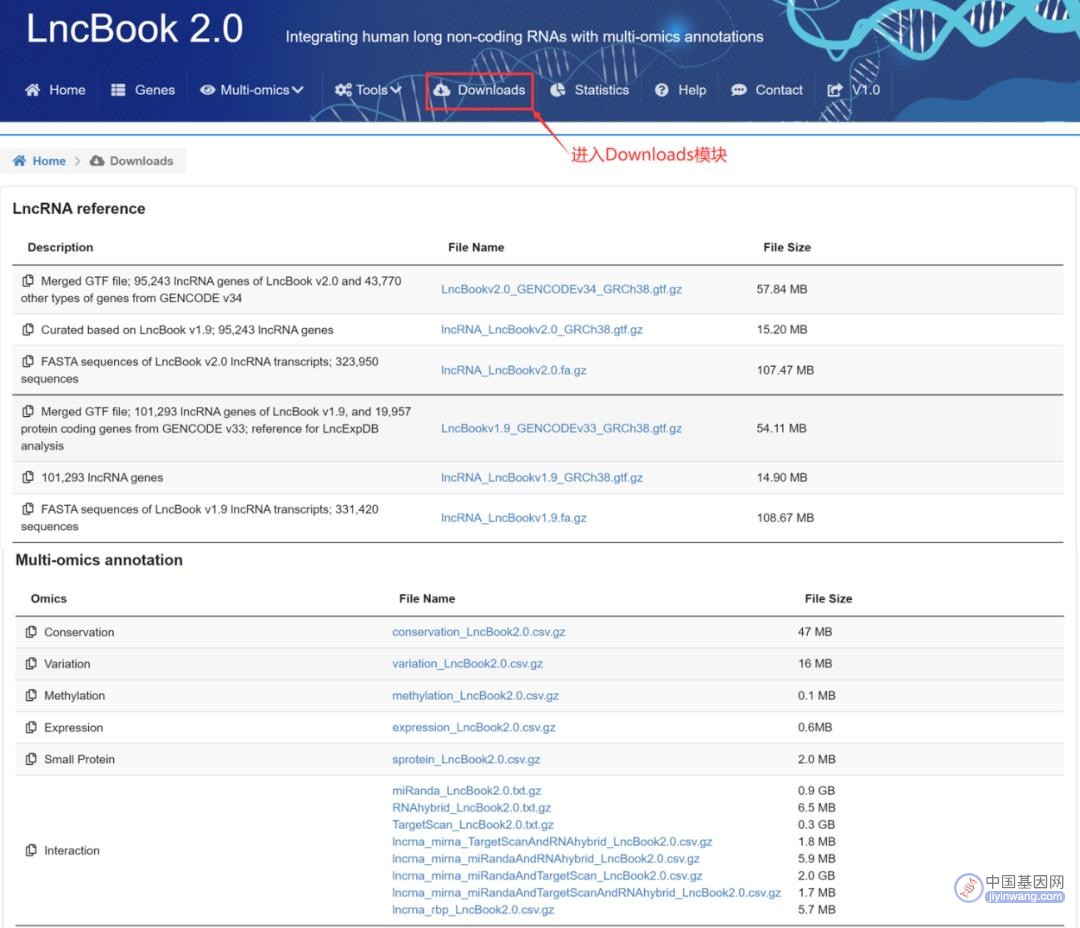
图2:人类lncRNA参考数据集下载
二、筛选潜在的功能性lncRNA基因
点击进入“Genes”模块。“Genes”模块从序列保守性、基因表达、DNA甲基化修饰、基因组变异、lncRNA-蛋白互作和小蛋白表达方面提供了高质量的分子特征关联分析。例如通过如下过滤条件筛选lncRNA基因:
高度保守:在“Conservation(age)”处过滤,如基因年龄为17;
具备高表达能力:“Expression Capacity”选择则“HC”;
表达或甲基化呈特征变化:“FeaturedExpression”/“Featured Methylation”(yes);
具有疾病或性状相关变异:“Variation(disease/trait-association)”(yes);
与蛋白质有互作:“Interaction”(yes);
编码小蛋白:“Small Protein”(yes)。
筛选结果如图3所示,可通过“Download”一键下载基因列表。
点击GeneID可全面了解单个基因的细节信息~
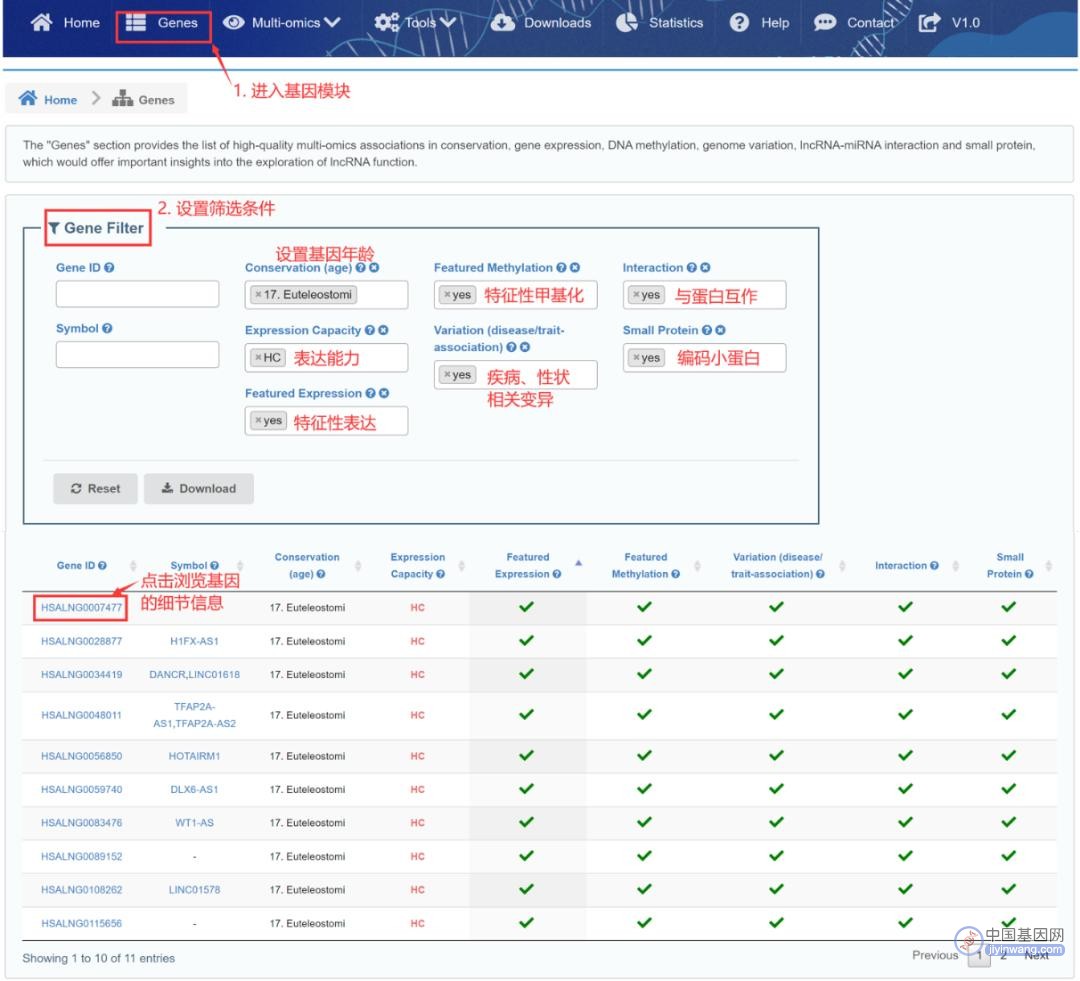
图3:功能性lncRNA基因筛选
三、获取人类lncRNA基因在各物种中的同源信息
“Conservation”模块提供了人类lncRNA基因在40种脊椎动物间的保守性特征(包括基因年龄、出现基因的物种个数),以及在每个物种中的比对细节(包括比对物种及对应物种基因组组装、保守程度、同源基因等信息)。用户可以在该界面进行如下分析:
①获得某一人类lncRNA基因在不同物种中同源性的蛋白编码或者非编码基因(这里以MALAT1为例)(图4):
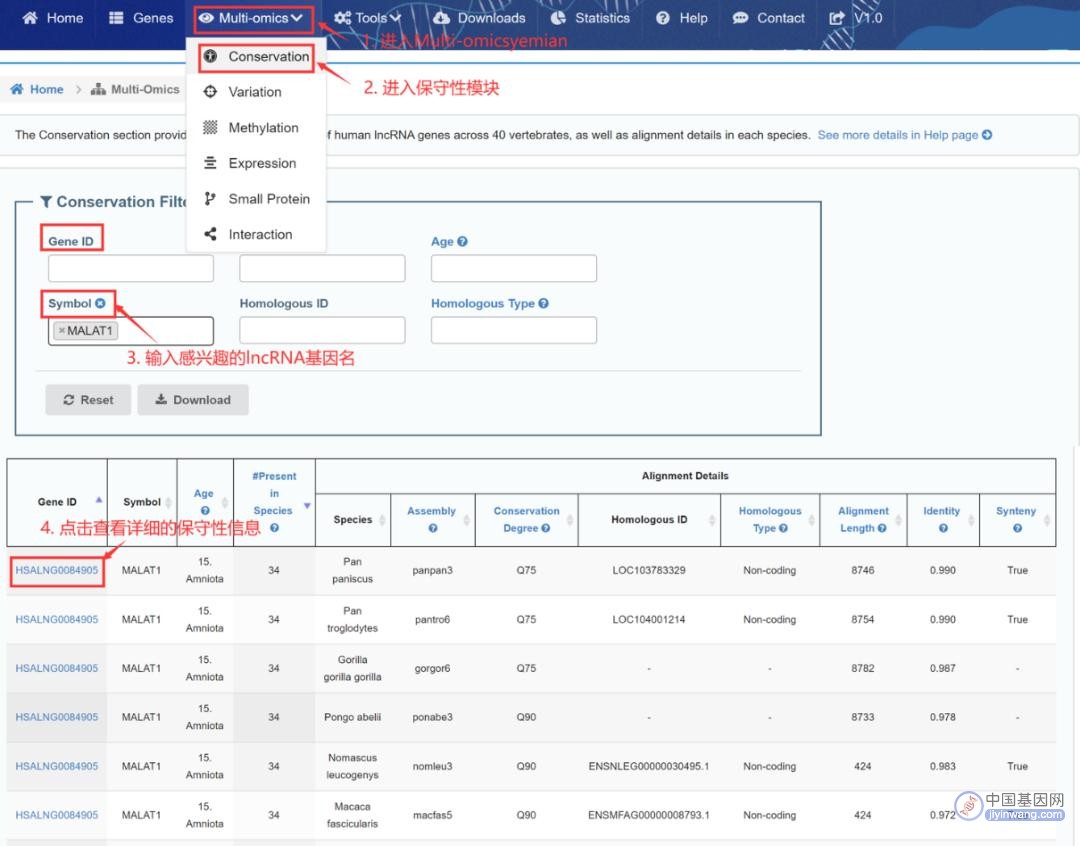
图4:查询MALAT1基因的同源基因
页面跳转后,我们能看到图形化展示的lncRNA基因进化树,以及详细的同源基因信息和序列比对信息,而且都是可下载的哦(图5)!其中Q90保守程度最高,黄色字体代表该物种中有同源的蛋白编码基因,蓝色表示有同源的非编码RNA基因。
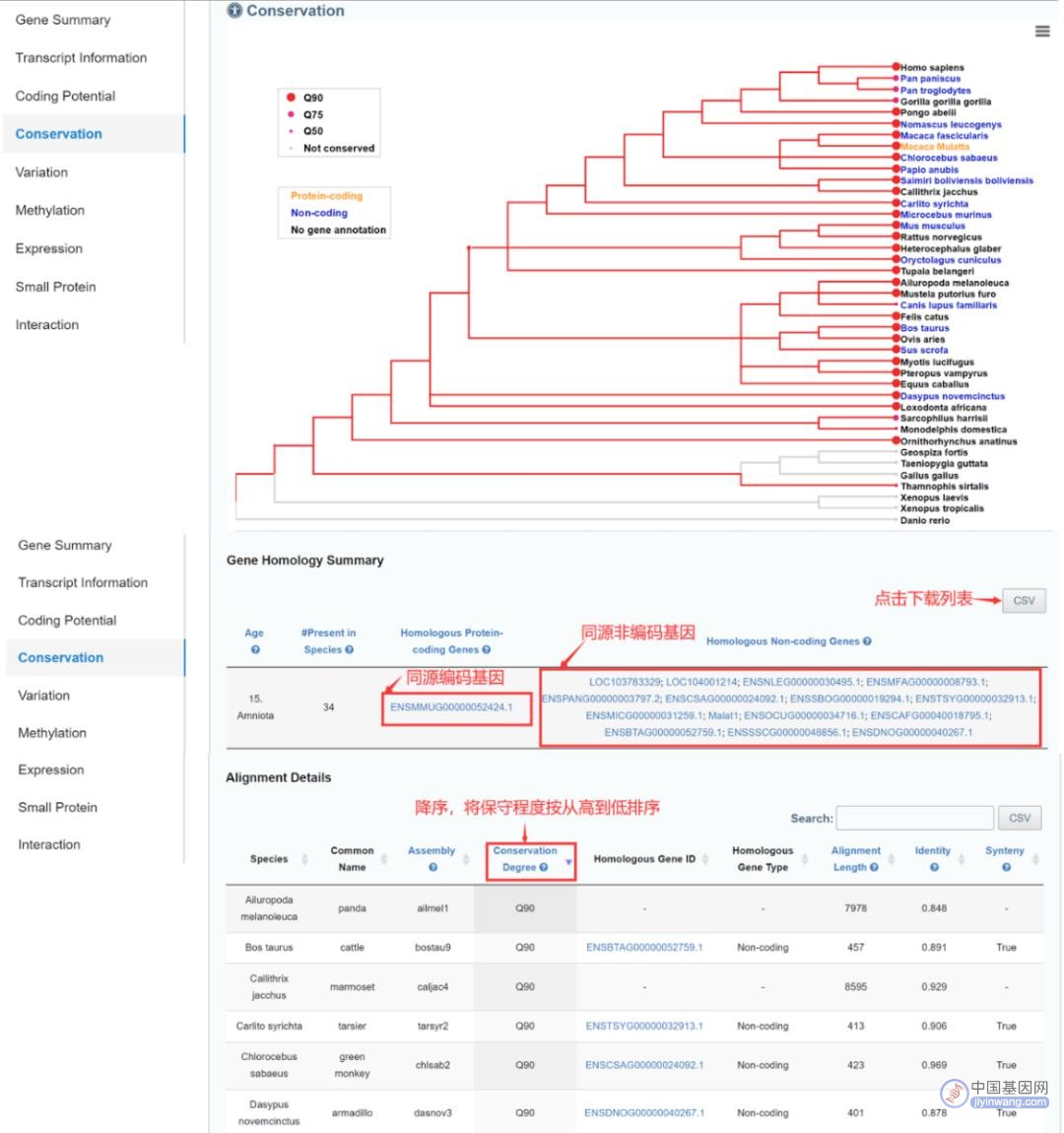
图5 MALAT1基因的保守性信息
②搜索最保守或者人类特异的lncRNA基因(图6):
lncRNA的基因年龄为同源序列最早出现的时间,从最新到最古老依次为:"Homo" (humanspecific)、"Hominini"、"Homininae"、"Hominidae"、"Hominoidea"、"Catarrhini"、"Simiiformes"、"Haplorrhini"、"Primates"、"Euarchontoglires"、"Boreoeutheria"、"Eutheria"、"Theria"、"Mammalia"、"Amniota"、"Tetrapoda"、"Euteleostomi"。
因此我们可以通过对基因年龄进行筛选以找到最保守(“Age”→“17.Euteleostomi”)或者人类特异(“Age”→“1.Homo”)的lncRNA基因。
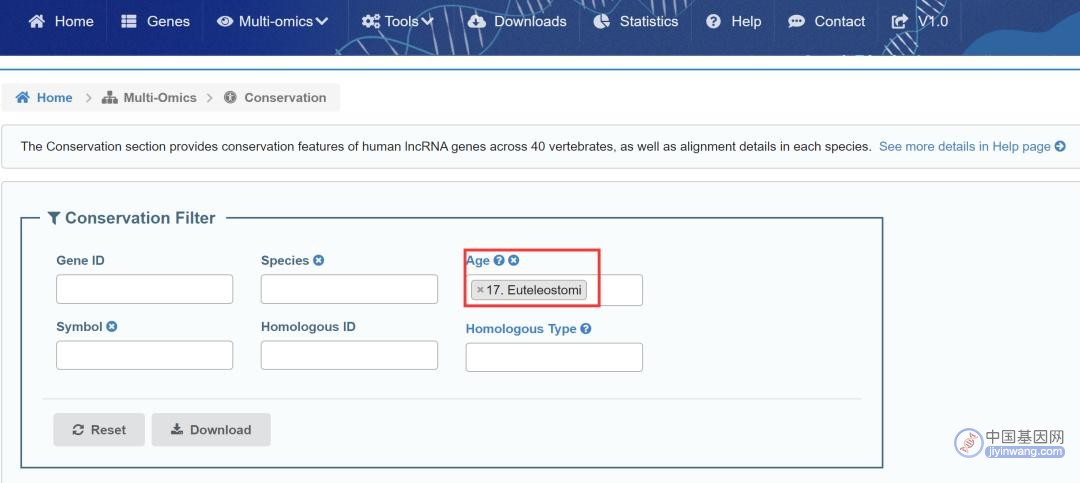
图6:搜索在40个脊椎动物中保守的人类lncRNA基因
四、查看lncRNA基因变异对功能的影响
“Variation”模块提供了lncRNA变异与疾病、性状的关联注释。数据库从COSMIC、ClinVar和GWAS Catalog中收集了疾病/性状相关的变异和关联的疾病/性状信息,并将其映射到lncRNA位点上。在“Variation”模块中用户可以:
①根据基因ID、dbSNPID,查看lncRNA基因变异类型,以及变异关联的疾病或性状(图7)。
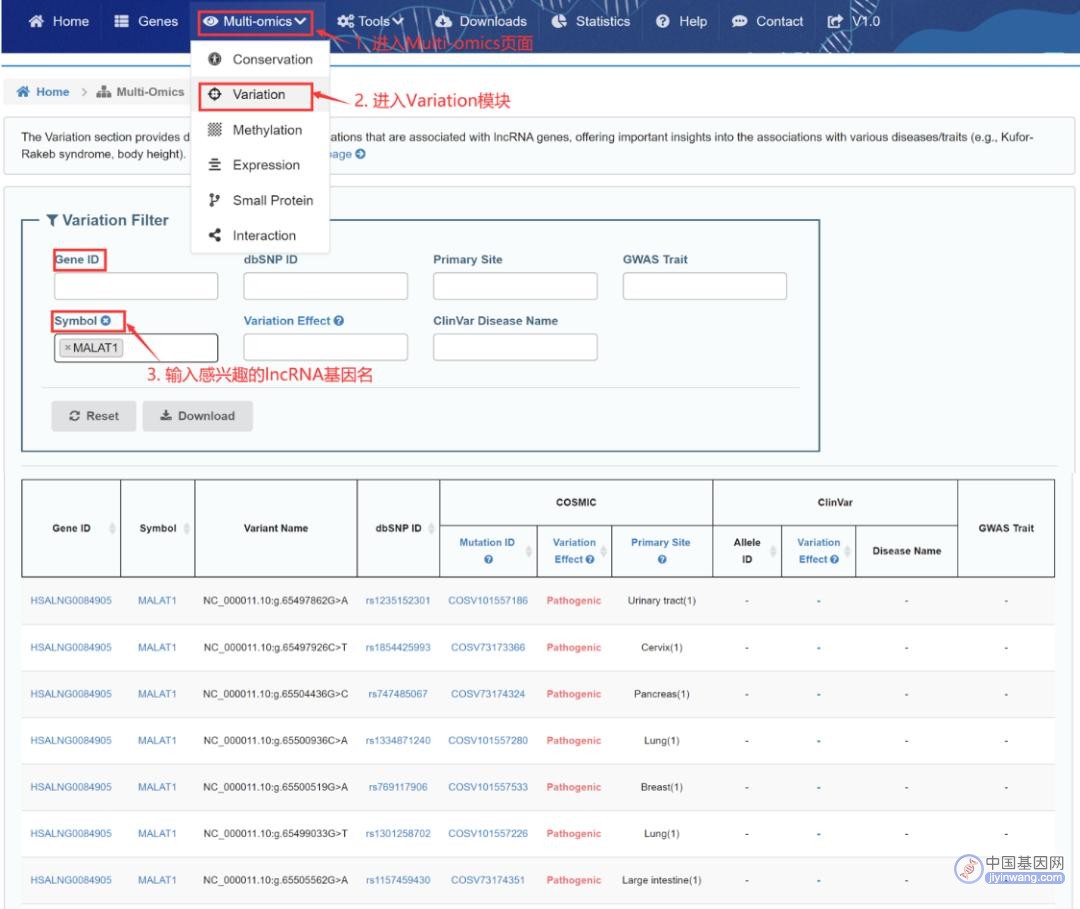
图7:MALAT基因变异注释
②根据已知的功能影响、疾病名称或性状,搜索相关lncRNA及其变异类型,并下载感兴趣条目(图8)。
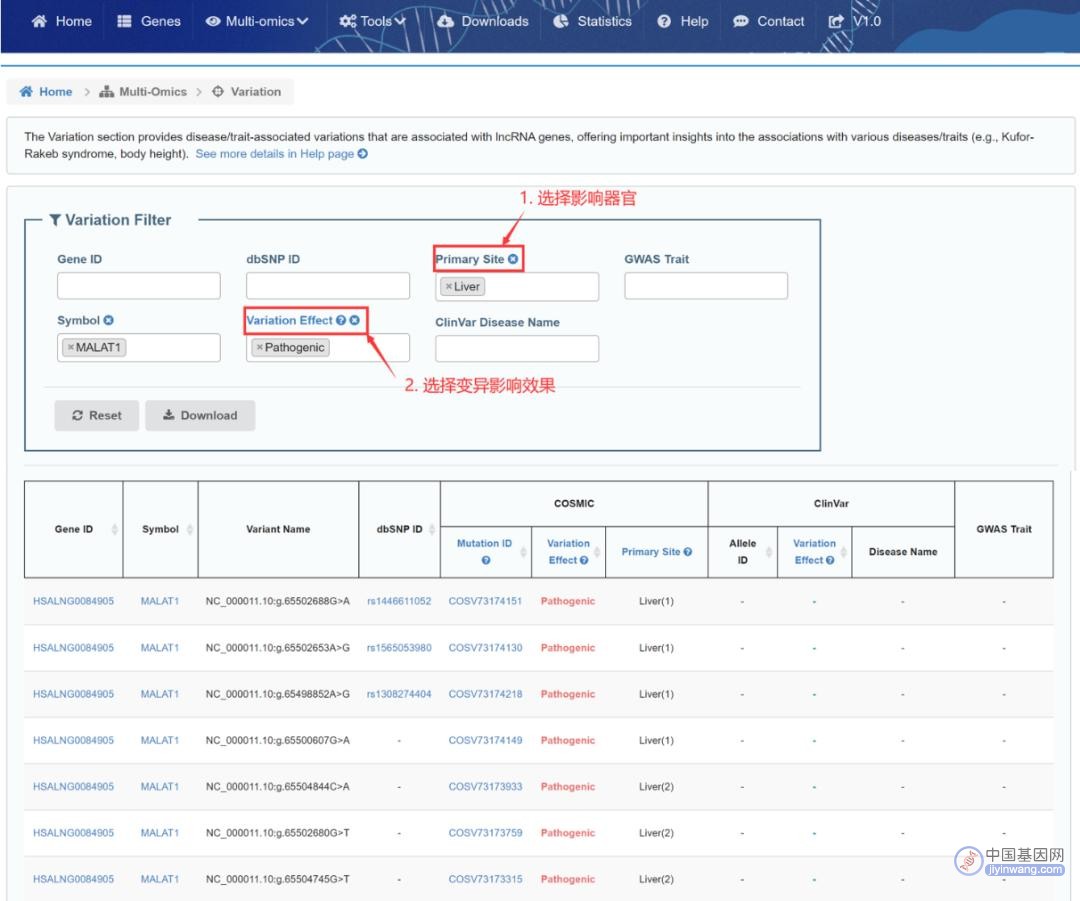
图8:基于变异影响筛选lncRNA
五、筛选在疾病中表现出特异甲基化修饰的lncRNA基因
“Methylation”模块提供了在至少一种疾病中,启动子区或者body区有高/低甲基化变化的lncRNA基因。在这里用户能够:
①浏览感兴趣的lncRNA基因在16种疾病(目前包括14种癌症和2种精神类疾病)中的特异性甲基化情况:“Gene ID”/“Symbol”处输入基因名。
②筛选在特定疾病中差异甲基化的lncRNA基因:在特定疾病的下拉菜单中选择“hyper”(高甲基化)/“hypo”(低甲基化)(图9)。
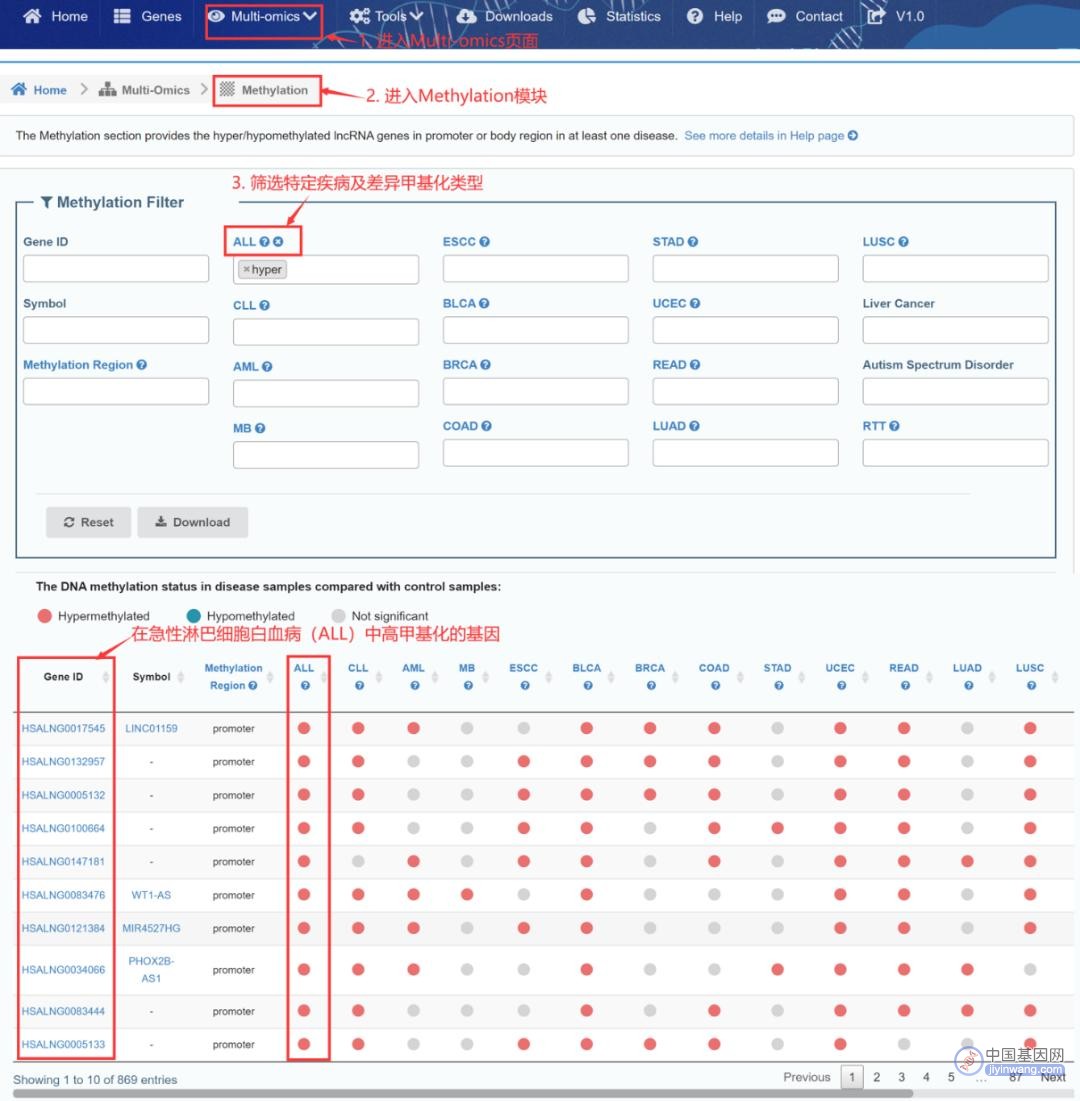
图9:搜索在ALL中表现出高甲基化修饰的lncRNA基因
六、查询在不同生物学场景下存在特征表达的lncRNA基因
“Expression”模块评估了9种生物学场景下lncRNA基因的表达能力以及337个生物学条件下基因的表达水平,并鉴定了特征表达的基因。以上内容整合自LncExpDB数据库(https://ngdc.cncb.ac.cn/lncexpdb/),想要了解各生物学场景下lncRNA的表达谱系统分析,特征lncRNA基因(管家基因/组织特异性基因、差异表达基因、节律基因、动态表达基因、亚细胞区室富集基因)的鉴定,以及lncRNA相互作用靶基因相关内容可以关注LncExpDB哦!
用户不仅可以选择在一种或多种环境下具有高表达能力的lncRNA基因,还可以获得至少在一个生物学场景中特异表达/一致表达/差异表达/动态表达/周期性表达的lncRNA基因,此类基因被定义为特征性表达基因(标示为★)。筛选结果均可下载(图10)!
注:在至少一种条件下lncRNA基因表达值高于整个转录组(包括lncRNA基因和蛋白编码基因)表达值上四分位数的基因被定义为高表达能力,即HC(标注为红色)。低表达能力lncRNA基因系指在所有条件中表达值低于整个转录组表达值下四分位数的lncRNA基因,即LC(标注为蓝色)。其余lncRNA基因被定义为中间表达能力基因,即MC(标注为橙色)。在特定生物学场景的所有生物学条件下,最大表达值小于1.0 TPM的基因被定义为无表达基因,即NE(标注为灰色)。
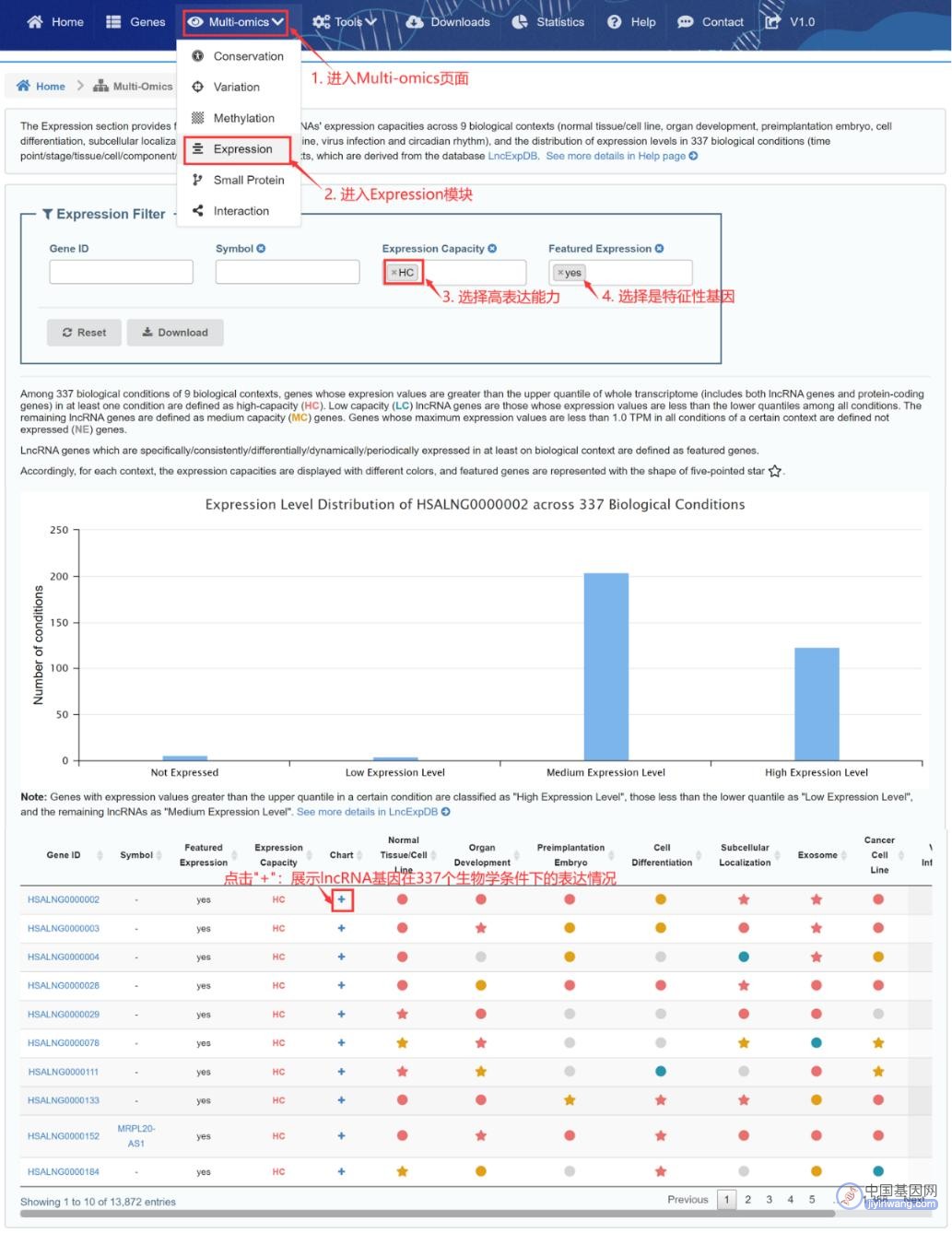
图10:查询不同生物学场景下具有高表达能力且具有特征表达的lncRNA基因
七、获取lncRNA编码的小蛋白
“Small Protein”模块整合了SmProt中由Ribo-seq和质谱实验证实的小蛋白(Small protein),通过BEDtools将其比对到lncRNA上,并提取完全且唯一落在lncRNA转录本上的小蛋白。共记涵盖了5,743个lncRNA基因编码的34,012个小蛋白。
用户可通过基因ID、小蛋白ID或者实验证据搜索感兴趣的条目(这里以MALAT1基因为例,图11),点击下载按钮就可以进行下载啦~
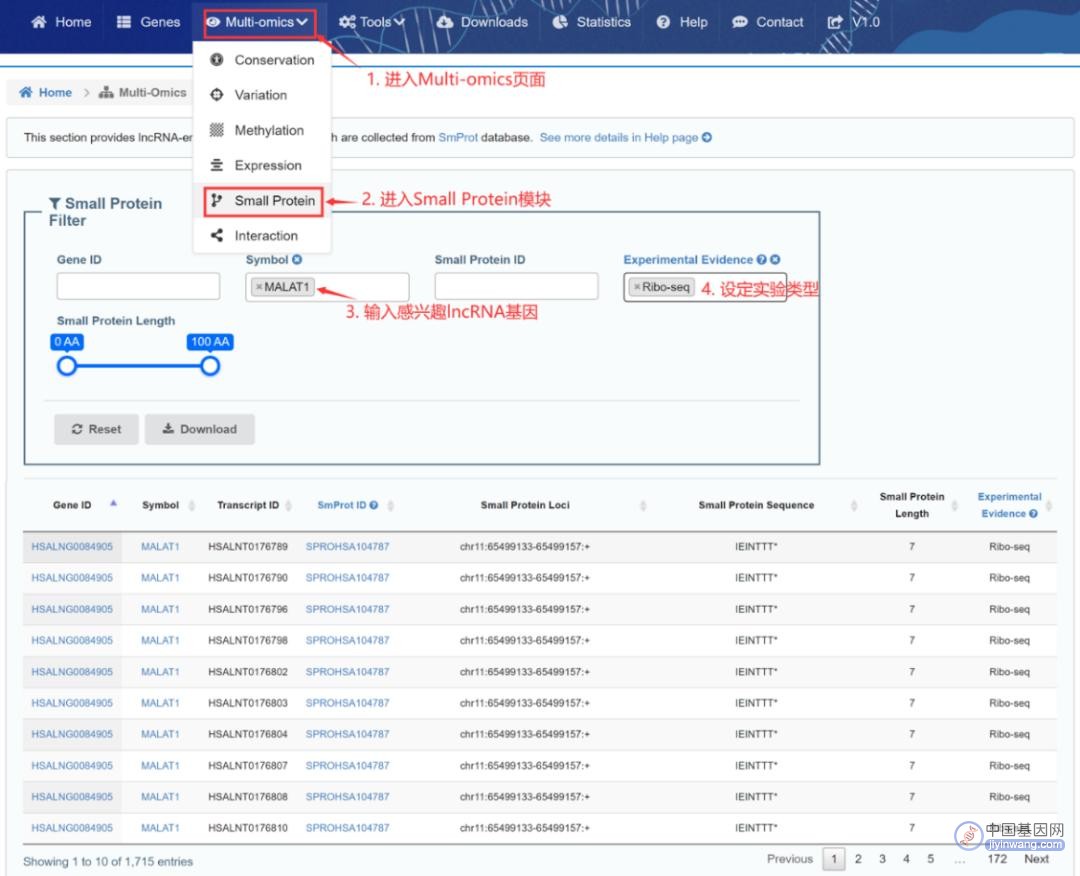
图11:搜索MALAT1经Ribo-seq实验验证的编码小蛋白信息
八、查询lncRNA互作miRNA和蛋白质
“Interaction”模块使用三种工具(miRanda, TargetScan和RNAhybrid)对lncRNA-miRNA相互作用进行预测。在至少两个工具中得到验证的关系对得以被保留,对全部lncRNA预测了146,092,274个lncRNA-miRNA互作。注意,只有一种软件支持的互作关系不在页面展示,可从“Downloads”模块获取。lncRNA-miRNA互作的查询步骤如图12:
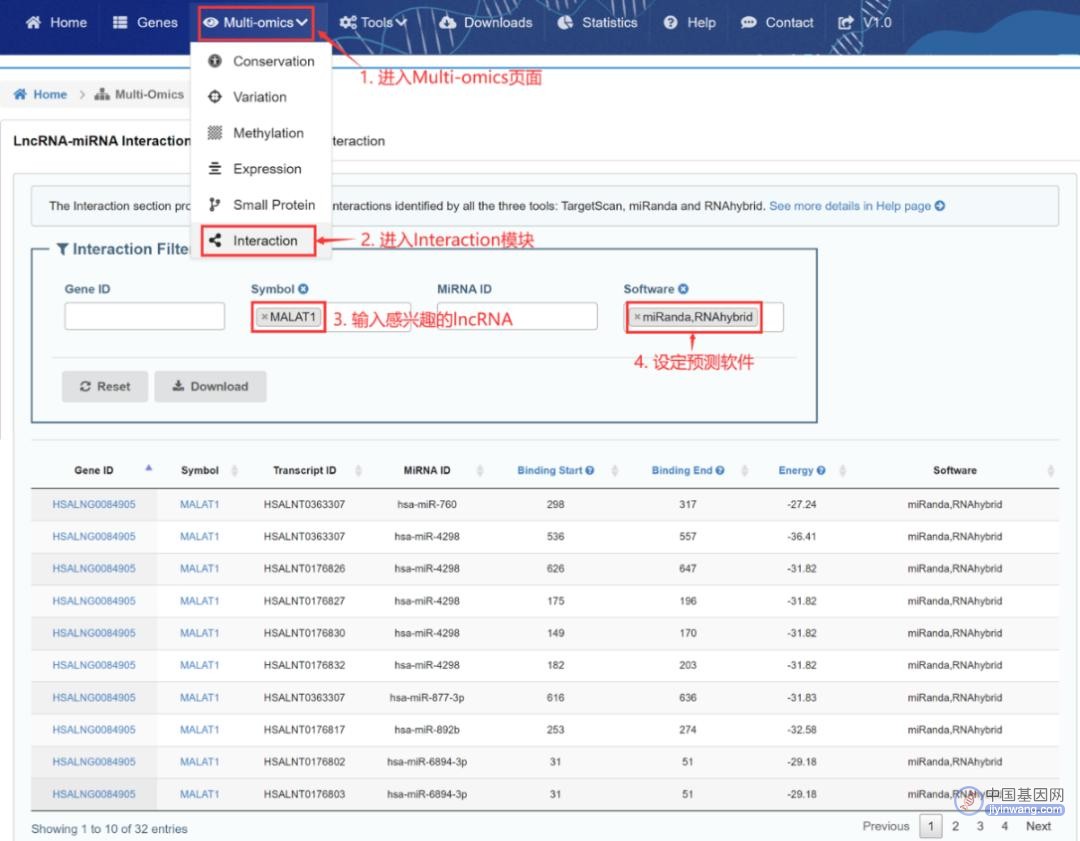
图12:检索经miRanda和RNAhybrid预测的与MALAT1互作的miRNA
数据库基于ENCODE收集的HepG2和K562细胞系中150个RNA结合蛋白(RNA Binding Protein,RBP)的848,077个RBP结合位点,通过BEDtools将RBP结合位点映射到lncRNA上,并保留了那些完全唯一落在lncRNA转录本上的RBP结合位点。共计获得了2022个lncRNA的772,745个lncRNA-蛋白质相互作用。lncRNA-蛋白质互作的查询步骤如图13:
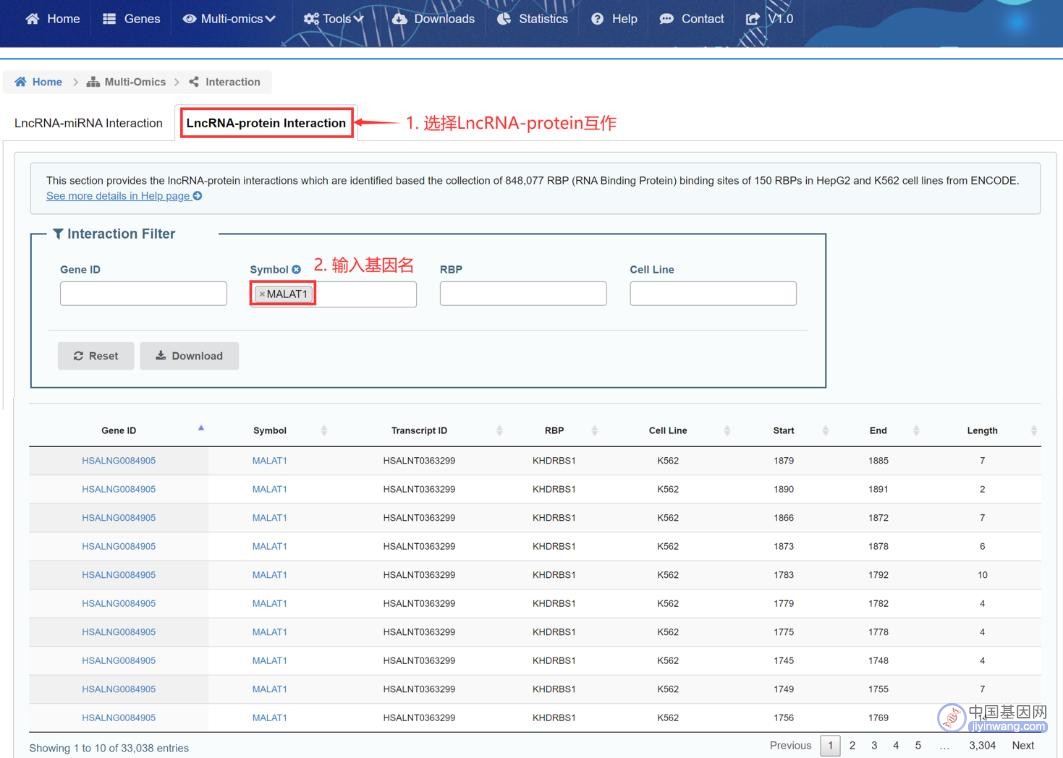
图13 检索与MALAT1互作的蛋白质
九、支撑多种检索功能
在Home界面,用户可通过检索Gene Symbol ID、lncRNA ID、miRNA、dbSNP ID、Small Protein ID、GWAS特性、变异影响、物种等信息,浏览感兴趣的内容(图14)。
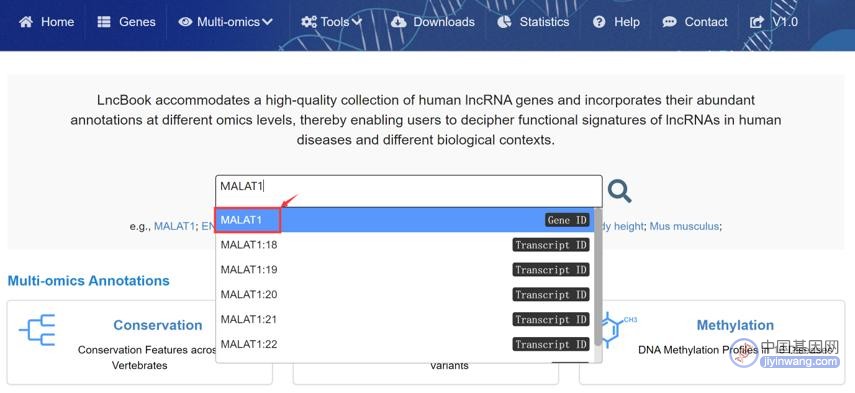
图14:快速检索MALAT1基因信息
在基因页面,以lncRNA基因MALAT1的检索为例,我们能查看到基因的详细信息,包括lncRNA基因的基本信息总结(Gene Summary)、转录本信息(Transcript Information)、lncRNA转录本的编码潜能(Coding Potential)以及多组学信息(图15)。页面展示的分析结果都可以一键获取哦~
值得注意的是,单击基因ID可以跳转到LncExpDB页面,用户可以查看不同生物背景下的lncRNA基因的表达谱。通过点击GeneSymbol,用户还可以获取lncRNA在LncRNAWiki中的文献报道信息,包括疾病关联、药物、靶基因、调控因子、实验样本、生物学功能、CRISPR实验以及文献。
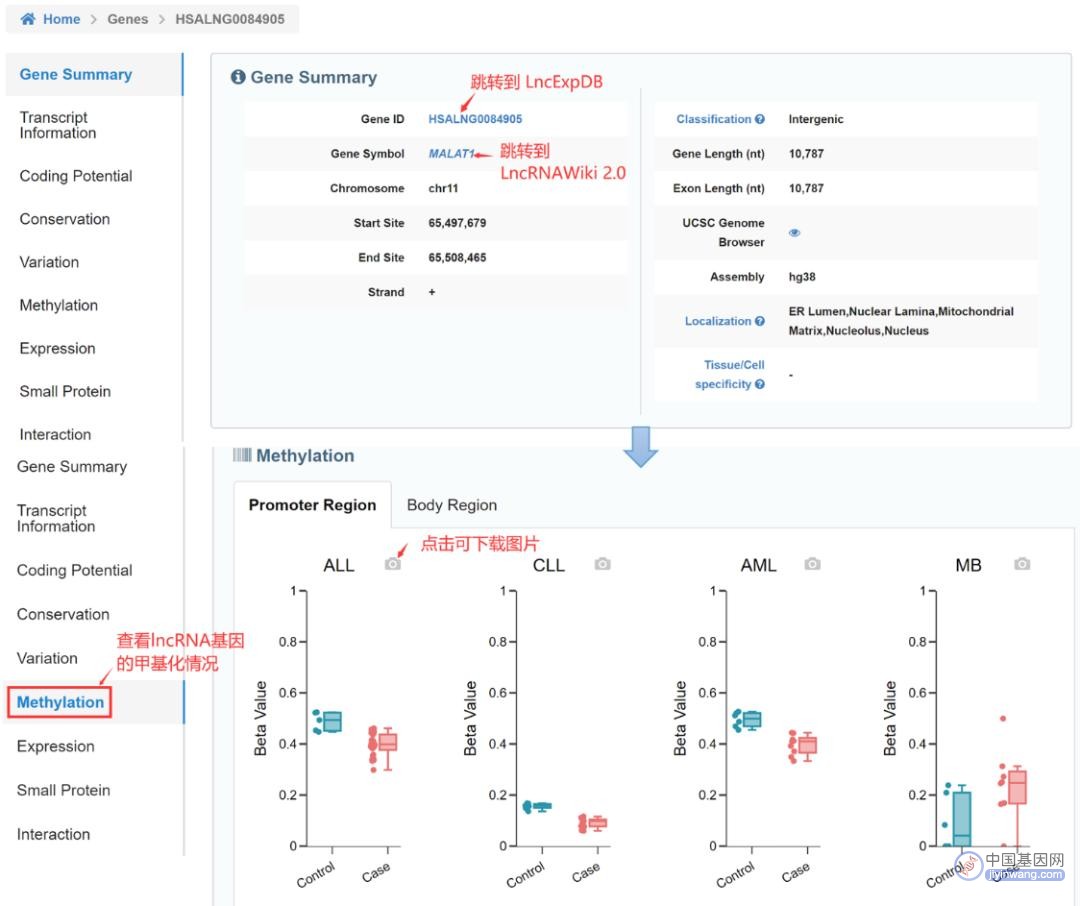
图15:MALAT1基因细节信息
十、在线分析工具的使用
ID Conversion:上传包含基因ID的文件或输入基因ID即可获得不同数据库(包括LncBook、NONCODE、GENCODE、LNCipedia、BIGtranscriptome、CHESS、RefLnc、MiTranscriptome、FANTOM、HGNC、Entrez、UCSC、RefSeq以及lncRNAdb)中该基因相应的ID。
LGC:输入Fasta文件以进行转录本编码潜能预测。
BLAST:上传Fasta文件以查找相似的转录本。
Classification:该工具支持对用户输入的GTF文件自动进行转录本分类。
以上便是LncBook 2.0 的基本使用步骤啦,想要了解更多内容欢迎点击数据库链接(https://ngdc.cncb.ac.cn/lncbook/)进行浏览。
参考文献:
1.LncBook 2.0: integrating human long non-coding RNAs with multi-omics annotations. Nucleic Acids Res 2022. Doi: 10.1093/nar/gkac999. [PMID=36330950], [OLB-PM=36330950]
2.LncBook: a curated knowledgebase of human long non-coding RNAs. Nucleic Acids Res 2019. Doi: 10.1093/nar/gky960. [PMID=30329098], [OLB-PM= 30329098]
声明:本站所有文章资源内容,如无特殊说明或标注,均为采集网络资源。如若本站内容侵犯了原著者的合法权益,可联系本站删除。