研究破解埃博拉病毒基因组从头起始复制的分子机制
9月12日,中国科学院微生物研究所施一、齐建勋、高福院士团队,在《自然》(Nature)上,发表了题为Molecular mechanism of de novo replication by the Ebola virus polymerase的研究文章,首次报道了nsNSV聚合酶识别3’-leader RNA的作用模式,揭示了EBOV聚合酶发挥从头起始复制活性的分子机制,促进了对nsNSV聚合酶合成RNA过程的理解,并为开发针对nsNSV复制过程的抗病毒药物提供了新靶点。
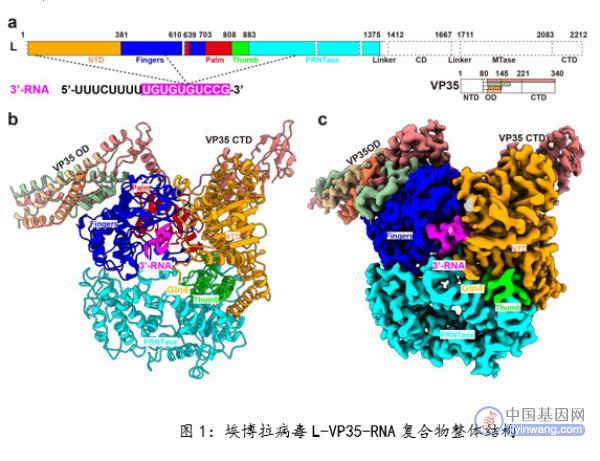
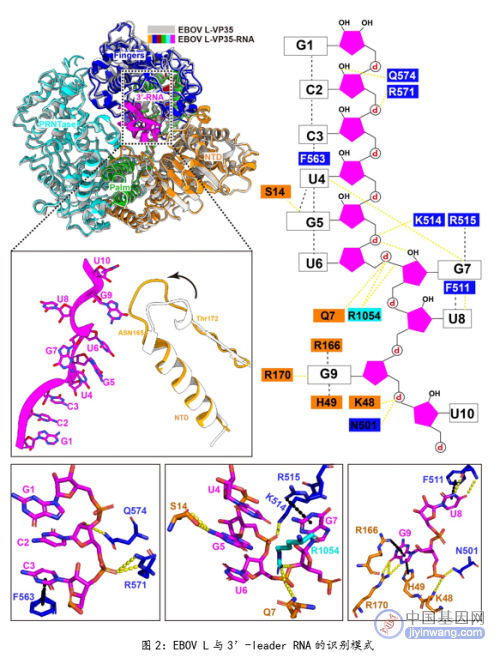
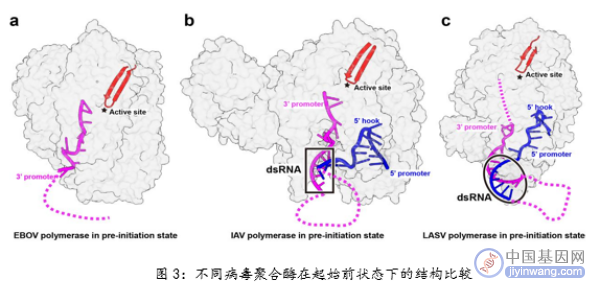
研究人员利用冷冻电镜技术解析了EBOV L-VP35-RNA复合物的高分辨率结构,发现了3’-leader RNA以独特的弯曲构象结合在模板进入通道中,通过RNA内部碱基与碱基之间的相互作用,以及RNA与聚合酶蛋白N末端结构域(NTD)、手指结构域(Fingers)和拇指结构域(Thumb)相互作用,使蛋白/核酸复合物稳定地处于起始前构象。研究还发现,G7碱基方向发生了翻转,并与U4碱基形成氢键相互作用,暗示这一特殊构象在病毒聚合酶从头起始复制过程中发挥着重要作用。
进一步,研究分别进行了RNA和关键结合氨基酸位点的突变实验。将G7突变为其他碱基时,不管在体外水平还是复制子水平,病毒聚合酶完全失去了从头起始复制的能力。而突变与核酸结合的关键氨基酸时,这些位点的突变对引物延伸活性的影响较小,而对从头起始复制活性的影响颇大。这表明3’-leader RNA与L蛋白独特的结合构象对病毒从头起始复制至关重要。
负链RNA病毒在宿主细胞中以核糖核酸复合物(RNP)为基本单元进行基因组的转录与复制过程。对于分节段的负链RNA病毒,其基因组的3’和5’端会部分互补配对形成“假环”结构,需要同时结合基因组的5’和3’末端,才能将聚合酶稳定在起始前构象。而对于不分节段的负链RNA病毒,其RNP复合物的3’和5’端分别位于相反的两端,只需要通过其3’末端的特殊弯曲结构,就能将聚合酶稳定在起始前构象。因此,该研究从分子水平说明了不分节段的负链RNA病毒聚合酶进化出了一种不同的复制起始机制来适应其RNP结构。
研究工作得到国家重点研发计划、中国科学院战略性先导科技专项、国家自然科学基金和中国科学院青年创新促进会等的支持。
论文链接:https://www.nature.com/articles/s41586-023-06608-1
声明:本站所有文章资源内容,如无特殊说明或标注,均为采集网络资源。如若本站内容侵犯了原著者的合法权益,可联系本站删除。