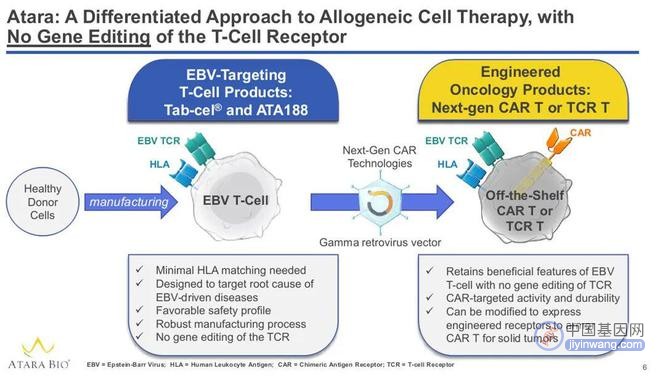
《自然》重磅:张锋团队首次在真核生物中找到类Cas蛋白,有望带来较CRISPR更精准的下一代基因编辑工具
张锋团队又发论文啦!

今日,《自然》杂志刊登张锋团队新研究成果,研究者首次在真核生物中找到受RNA引导的核酸内切酶Fanzor(Fz),并可组装能对人类基因组进行编辑的类CRISPR/Cas系统,经初步改造编辑活性可达18.4%。该蛋白的真核起源和较小体积,都预示着它可能具有比目前CRISPR/Cas更广阔的应用场景。

论文题图
毫不夸张地说,CRISPR/Cas的出现为生物学发展带来了巨大的变革,起源于原核生物的CRISPR也让人好奇,是否在真核生物中也存在类似的系统。
2021年,张锋团队在《科学》发文,他们发现了一种类CRISPR系统OMEGA(obligate mobile element–guided activity)。OMEGA系统由转座子末端转录的非编码RNA(ωRNA)和内切酶组成,其中3种转座子编码蛋白IscB、IsrB、TnpB是天然存在的RNA引导的核酸酶,且IscB和TnpB分别为Cas9和Cas12的可能祖先。
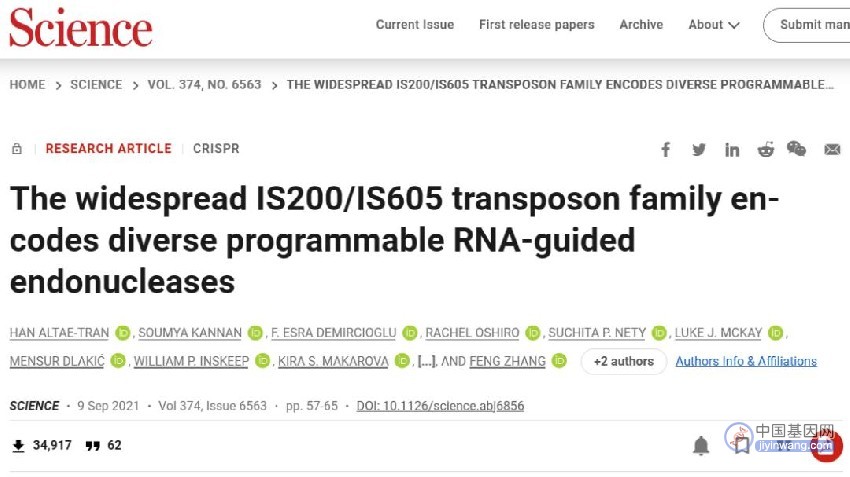
而早在2013年,Fanzor蛋白就被报道为一种真核TnpB-IS200/IS605样蛋白,这不由得让人怀疑,Fanzor就是那个我们还未知的真核生物中的Cas。
经公开遗传数据库搜索,研究者发现Fanzor蛋白广泛存在于真菌、原生生物、节肢动物、软体动物、巨病毒等物种,可分为Fz1和Fz2两种不同的独立起源,并发现了细菌TnpB向真核生物水平转移并进化为Fanzor的痕迹。
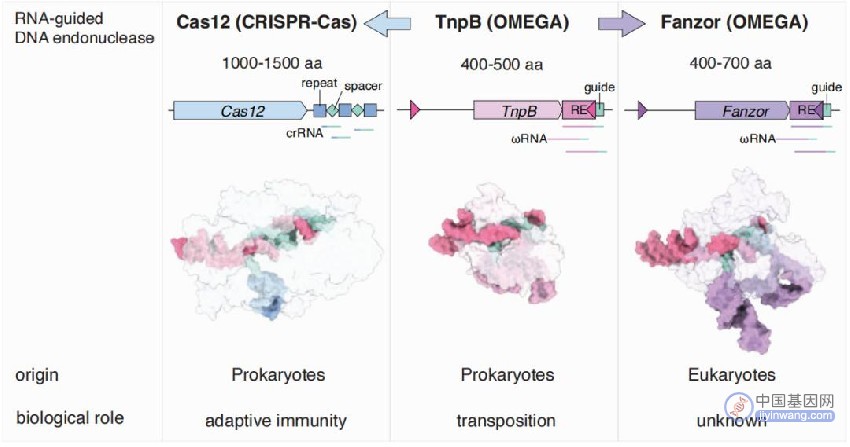
与TnpB和Cas12的结构对比可以看出,Fanzor结构与它们非常相似,这无疑说明Fanzor可能具有类似的功能。
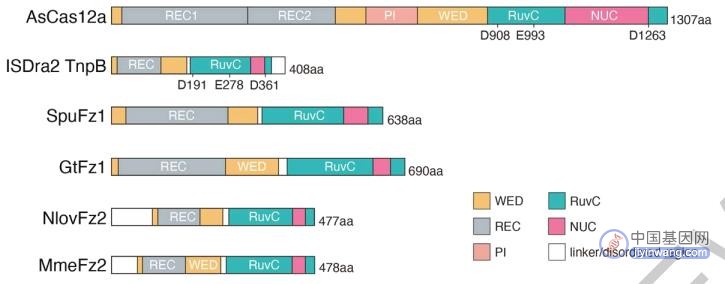
Cas12、TnpB、Fanzor的结构对比
研究者猜测,Fanzor可能以ωRNA 3端侧翼序列为向导RNA,在目标DNA序列执行切割功能。为此,他们构建了Fz OMEGA系统,并与质粒文库匹配进行切割实验。
实验结果可见,不同Fanzor蛋白具有特定的切割模式,并具有针对双链DNA(dsDNA)的特异性。
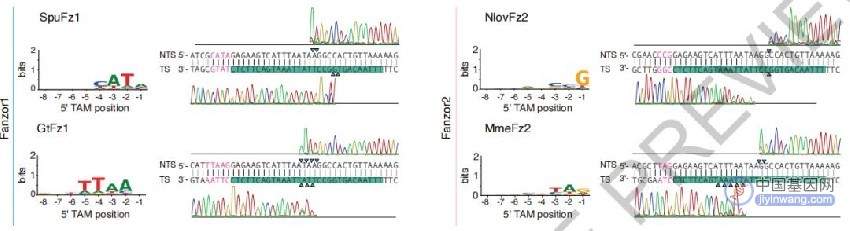
不同Fanzor蛋白具有特定的切割模式
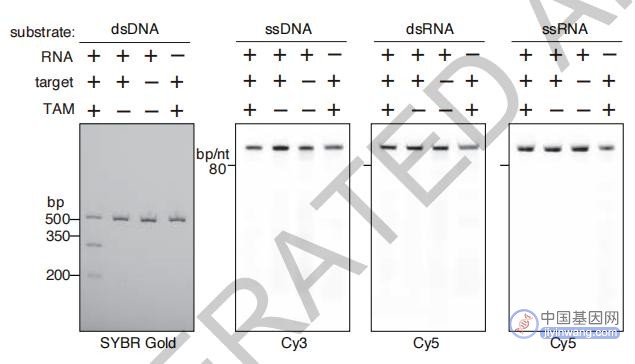
Fanzor表现出ωRNA引导的、TAM和靶序列依赖的dsDNA切割
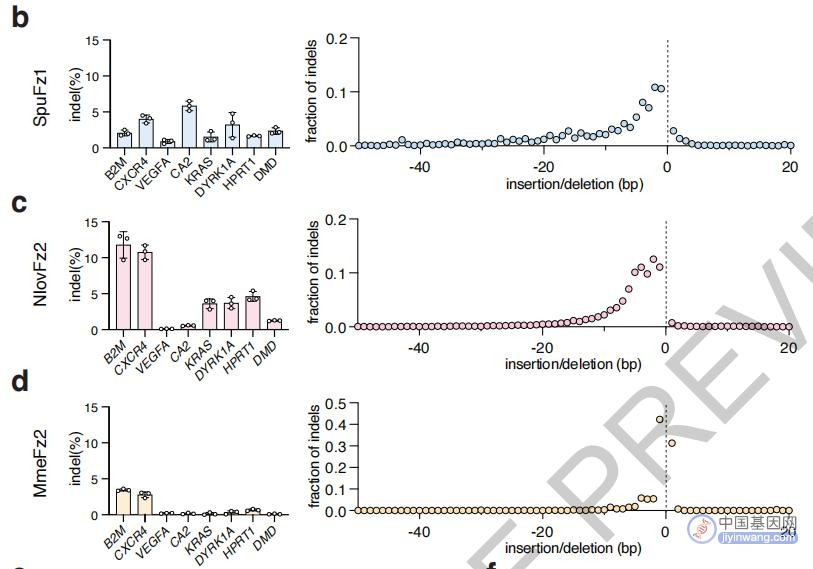
研究者在人类细胞中测试了Fz OMEGA的编辑效率,针对8个不同基因位点,4个Fanzor同源物中有3个表现出了可测量的编辑活性,效率最高达11.8%,总体水平与AsCas2f1相当。
编辑效率最高达11.8%
为提升编辑效率,研究者还尝试了修饰ωRNA和向Fanzor中引入突变。
多方尝试之下,可将编辑效率最高提升至18.4%。
不同修饰ωRNA(上)和Fanzor突变(下)后的编辑效率
研究者还通过冷冻电镜技术分析了SpuFz1的结构,在2.7Å下可见典型的双球形结构,REC和WED结构域识别包含TAM的DNA双链,NUC和RuvC结构域则形成了类似Cas的沟槽,容纳ωRNA与DNA形成的异源双链。
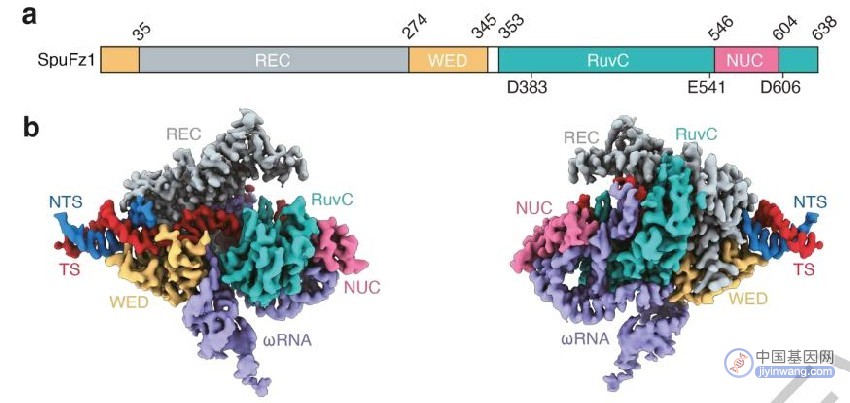
Fanzor结构
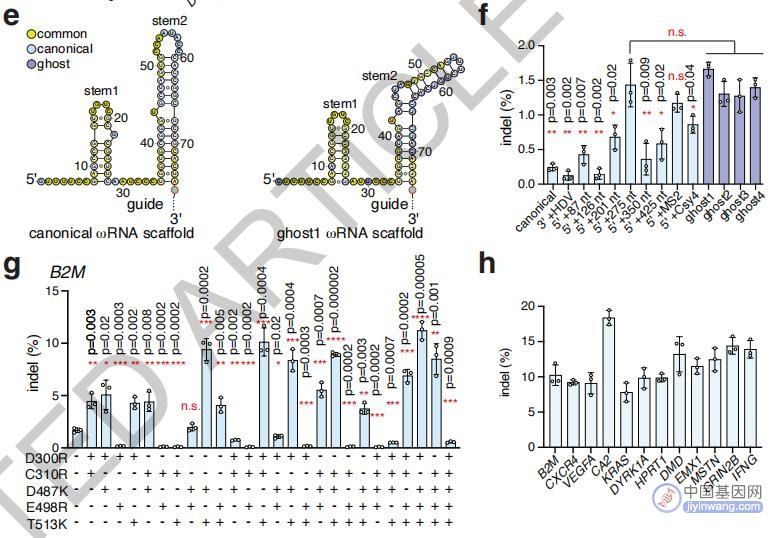
最后,研究者还对SpuFz1的天然ωRNA结构进行了分析,确定其中tem2的区域是功能所不需的,去除后ωRNA总长为96nt,可令结构更紧凑、便于应用。
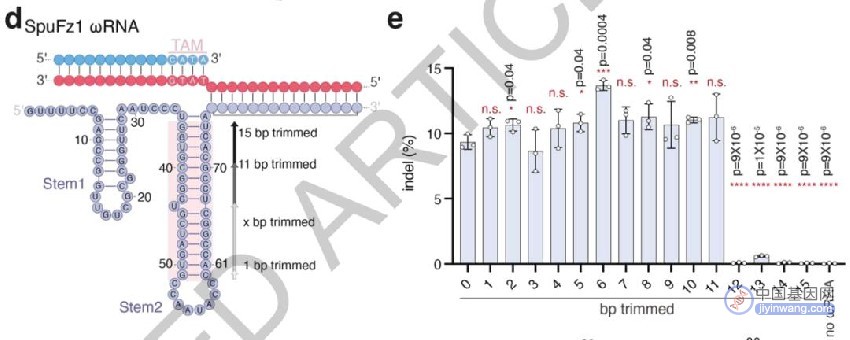
ωRNA结构中tem2不影响活性
不过,目前为止,研究者们还没有搞清楚Fanzor蛋白的生理功能,仅猜测与转座有关。Fanzor的真核生物起源和它相较Cas12等更小的大小,使得它有潜力成为新一代的基因编辑手段,但是它的天然功能使其可能面对在生物体内活性低、作用严重受控等问题。研究者认为,这可以通过基因工程改造来优化。
参考资料:
[1]Saito, M., Xu, P., Faure, G. et al. Fanzor is a eukaryotic programmable RNA-guided endonuclease. Nature (2023). https://doi.org/10.1038/s41586-023-06356-2
[2]https://www.broadinstitute.org/news/researchers-uncover-new-CRISPR-like-system-in-animals-that-can-edit-the-human-genome
声明:本站所有文章资源内容,如无特殊说明或标注,均为采集网络资源。如若本站内容侵犯了原著者的合法权益,可联系本站删除。