Nature速递:通过网络推断和计算机模拟基因扰动解剖细胞特性

关键词:基因调控网络,多模态数据融合,复杂网络

论文题目:Dissecting cell identity via network inference and in silico gene perturbation
论文来源:Nature
论文链接:https://www.nature.com/articles/s41586-022-05688-9
细胞身份受基因表达复杂调控的支配,表现为基因调控网络。但以前用于分析基因调控网络的方法主要集中在静态网络结构上,因此确定静态基因调控网络如何在动态生物过程中控制细胞身份仍然是一个挑战。需要可扩展和可解释的方法来理解基因调控机制如何与观察到的复杂单细胞表型相关。
这项最新发表于 Nature 的工作,使用从单细胞多组学数据推断的基因调控网络来执行硅转录因子扰动,仅使用未扰动的野生型数据模拟细胞身份的后续变化。研究将 CellOracle,这种基于机器学习的方法应用于已建立的范式——小鼠和人类造血,以及斑马鱼胚胎形成——并且正确地模拟了由于转录因子扰动而发生的表型变化。
通过在发育中的斑马鱼中系统的硅转录因子扰动,研究模拟并实验验证了以前未报道的由 noto(一种已建立的脊索调节因子)损失引起的表型。此外还鉴定了一个轴向中胚层调节因子 lhx1a。综上,这些结果表明 CellOracle 可以用于分析转录因子对细胞身份的调控,并可以为发育和分化机制带来启发。并且,这项研究表明,基于自定义网络模型的仿真和矢量可视化实现了动态 TF 函数的可解释、可扩展和广泛适用的分析。
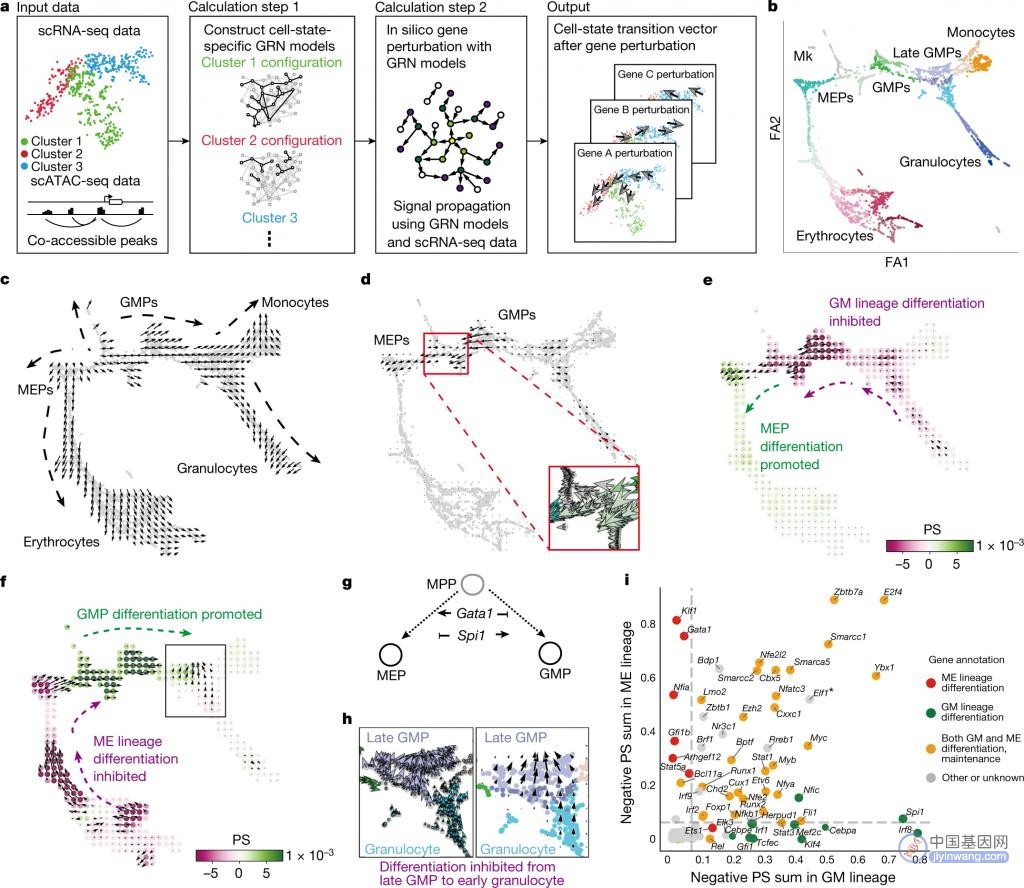
图 1. CellOracle 及其在造血系统中的应用概览。
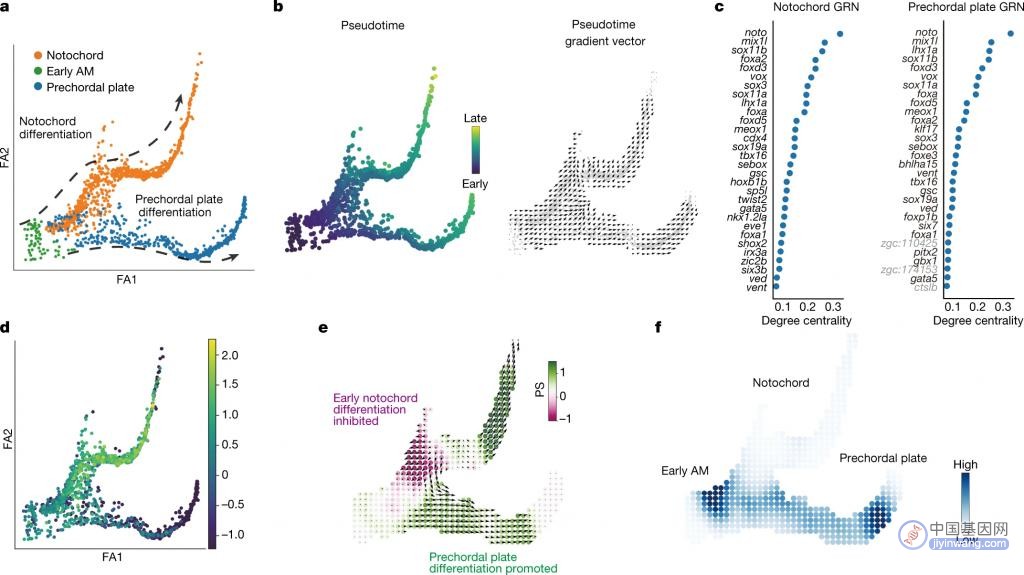
图 2. CellOracle KO 模拟与斑马鱼胚胎形成数据。
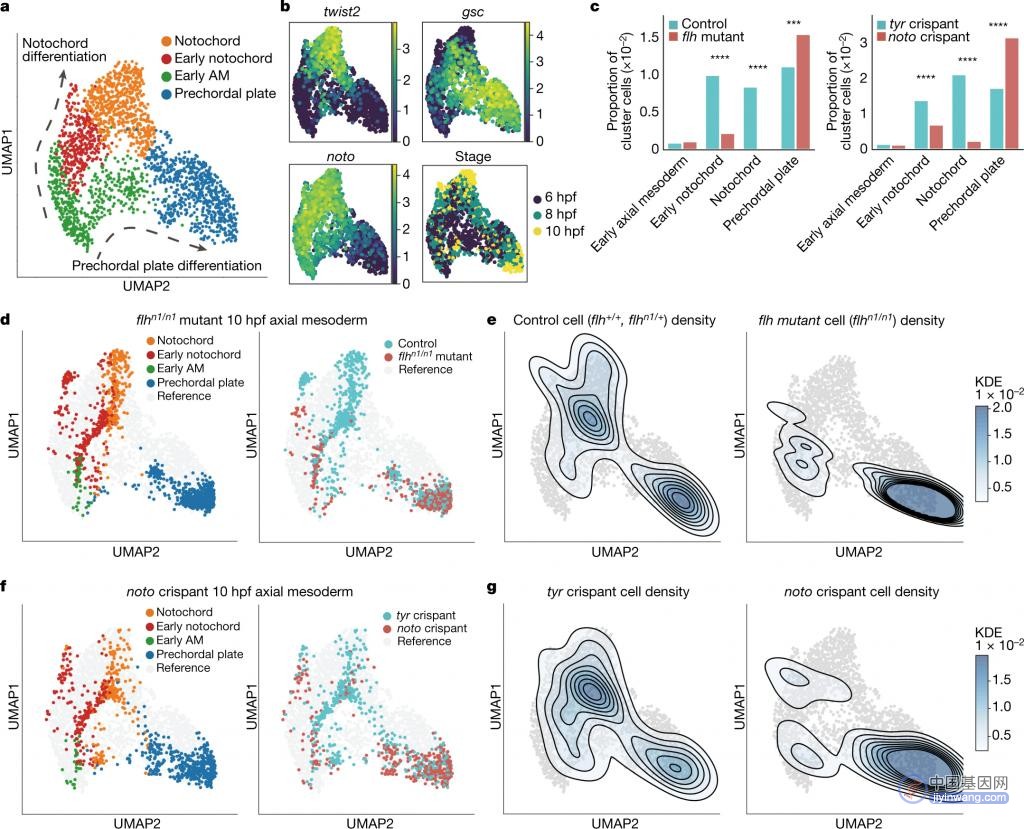
图 3. 斑马鱼 noto LOF 预测的实验验证。
声明:本站所有文章资源内容,如无特殊说明或标注,均为采集网络资源。如若本站内容侵犯了原著者的合法权益,可联系本站删除。