Immunity:瑞典研究团队揭示大量新的TCR等位基因变异
T细胞包含表达CD8+或CD4+ (αβ) T细胞受体 (TCR) 的细胞,可分别识别由MHC I类或II类分子呈递的肽,以及表达γδ TCR的细胞,可识别非肽磷酸化抗原 (pAg) 和类异戊二烯生物合成途径的中间体。αβ T细胞功能的主要调节因素在于个体的MHC背景,迄今为止已知有超过16000种I类和6000种II类等位基因变异【1】,γδ T细胞能利用可变基因TRGV4和TRGV9,依赖于CDR3(负责抗原识别的互补决定区)与靶标结合,还能通过对BTNL家族异二聚体的依赖性反应以固有方式发挥额外功能。与免疫球蛋白基因座类似,TCR基因座包含可变(V)、多变(D)和连接(J)基因,这些基因在T细胞发育过程中重组以产生具有不同结合潜力的功能性TCR。除了功能性基因外,基因座还包含许多无功能的假基因或缺乏功能重组序列的开放阅读框 (ORF)【2, 3】,它们与功能性基因的序列高度相似,这为短读长测序技术对变异等位基因的鉴定工作带来了困难【4】。
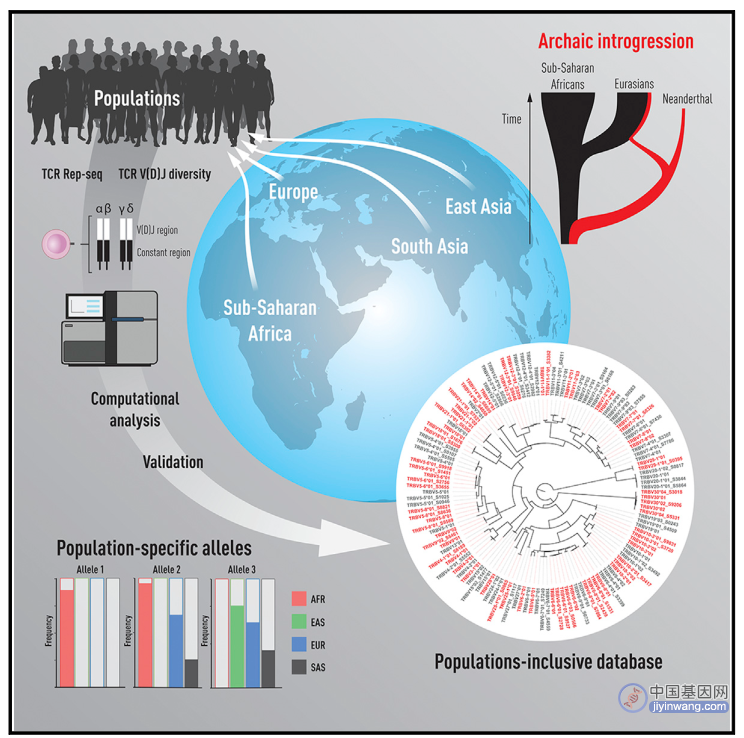
近日,来自瑞典卡罗林斯卡学院的Gunilla B. Karlsson Hedestam团队在Immunity杂志上发表了一篇题为Archaic humans have contributed to large-scale variation in modern human T cell receptor genes 的文章,他们对来自四个人群(非洲、东亚、南亚和欧洲)的45个供体表达的TCRα、β、γ和δ基因进行详细分析,揭示了175个以前未报告的TCR V和J等位基因。其中大部分包含编码变化,并且在人群中以不同的频率出现。重要的是,他们还确定了三个源自尼安德特人的渗入TCR区域,包括一个高度分化的TRGV4变体,它介导改变的嗜乳脂蛋白样分子3 (BTLN3) 配体反应性,并且在所有现代欧亚人群中很常见。这些结果表明个体和群体中TCR基因的显着变异,为将等位基因变异纳入人类生物学中TCR功能研究提供了强大动力。

为了捕获TCR基因中的人类等位基因变异,作者收集了45名来自撒哈拉以南非洲、东亚、南亚或欧洲人群的个体的外周血单核细胞(PBMC)样本,使用Illumina MiSeq 2x300循环系统进行测序后利用IgDiscover生成完整的TRA、TRB、TRG 和TRD基因型,发现许多之前未报告的人类TCR V和J基因中高等位基因变异,并使用如靶向PCR和Sanger测序等方式对这些变异进行确认。对这些样本的TCR基因分型揭示了不同样本来源个体间的显著差异,比如确定了32个仅存在于非洲人群中的等位基因,以及仅存在于欧洲人群中的1个TRAV等位基因和3个TRBV等位基因等。
智人的免疫系统是在居住的特定环境下进化而来的,在大约6万年前,当他们从非洲迁移到欧亚大陆,与尼安德特人相遇并结合。通过与尼安德特人TCR基因序列的比较发现现代人类和古人类共享许多TCR等位基因,基因渗入分析显示尼安德特人的基因渗入都发生在非洲以外的现代人群中,渗入的TRGV4*02_S0072等位基因显示出与类人猿TRGV4等位基因的相似性,与已知人类TRGV4*02 相比仅差8 nt。于是作者想知道渗入的TRGV4*02_S0072的γδ TCR是否在功能上有所不同。已知γδ TCR与BTNL3–BTNL8的相互作用由结合到BTNL3的Vγ4的HV4和CDR2 loops介导的,将含有TRGV4*02或TRGV4*02_S0072的γδTCR的BTNL3反应性进行比较发现两种TCR在细胞表面上表达相似,在用抗CD3抗体刺激后对TCR交联反应同样敏感,然而,在表达BTNL3–BTNL8靶细胞的共培养实验中,与TRGV4*02 TCR相比, TRGV4*02_S0072 TCR下调显著降低,NFAT-GFP或NF-κB-CFP报告活性也显著降低。
接下来,作者进一步对倭黑猩猩、黑猩猩、大猩猩和恒河猴可用的TCR基因进行比较分析,分别鉴定出193、190、221和265个TCR等位基因,反映了TCR 基因座的整体基因组覆盖程度。变异主要位于CDR中,FRs(骨架区)在所有检查的灵长类物种之间高度保守,作者在文章中展示了这四种灵长类物种的TCR等位基因变异以及与每个TCR基因最接近的人类等位基因,这些数据为灵长类动物未来的TCR分析提供了有用资源。
总之,这项工作揭示了大量先前未报到的TCR VDJ等位基因变异,了解人类TCR遗传变异将有助于深入了解这种变异的功能作用,有助于定义迄今为止可能被忽视的TCR生物学的关键组成部分。
原文链接:
https://doi.org/10.1016/j.immuni.2023.01.026
参考文献
1. Robinson, J., Halliwell, J.A., Hayhurst, J.D., Flicek, P., Parham, P., and Marsh, S.G. (2015). The IPD and IMGT/HLA database: allele variant databases. Nucleic Acids Res. 43, D423–D431. https://doi.org/10.1093/nar/gku1161.
2. Cooper, M.D., and Alder, M.N. (2006). The evolution of adaptive immune systems. Cell 124, 815–822. https://doi.org/10.1016/j.cell.2006.02.001.
3. Dean, J., Emerson, R.O., Vignali, M., Sherwood, A.M., Rieder, M.J., Carlson, C.S., and Robins, H.S. (2015). Annotation of pseudogenic gene segments by massively parallel sequencing of rearranged lymphocyte receptor loci. Genome Med. 7, 123. https://doi.org/10.1186/s13073-015-0238-z.
4. Collins, A.M., Peres, A., Corcoran, M.M., Watson, C.T., Yaari, G., Lees, W.D., and Ohlin, M. (2021). Commentary on Population matched (pm) germline allelic variants of immunoglobulin (IG) loci: relevance in infectious diseases and vaccination studies in human populations. Genes Immun. 22, 335–338. https://doi.org/10.1038/s41435-021-00152-6.
声明:本站所有文章资源内容,如无特殊说明或标注,均为采集网络资源。如若本站内容侵犯了原著者的合法权益,可联系本站删除。