Nature揭示人类基因组非编码序列翻译调控新机制,为开发新型肿瘤免疫疗法提供基础
上世纪五十年代,沃森与克里克共同发现了 DNA 的双螺旋结构,使遗传研究深入到分子层面,让人类更加清晰地认识到遗传信息的构成和传递途径。
基因是生物体内控制遗传信息的物质基础,但 DNA 并不全是基因,研究发现人类基因组中的绝大部分 DNA 序列都不编码蛋白质,被称为“非编码序列”,其拥有什么功能以及如何进化成为能够编码蛋白质的基因等问题尚不明确。
近期,哥伦比亚大学吴雪兵课题组通过大量研究揭示了人类细胞降解所有类型非编码翻译产生的蛋白的统一机制,发现 BAG6 系统特异性识别非编码蛋白上 C 端疏水区进而导致蛋白降解。目前,这项研究成果已经以“Noncoding translation mitigation”为题发表在 Nature 上。

(来源:Nature)
“尽管一般认为非编码序列存储了复杂的基因调控的信息,但整体而言对于非编码序列的作用所知甚少。此次,我们的新研究揭示了非编码序列翻译调控的新机制,为理解非编码翻译在肿瘤、衰老及神经退行性疾病中的调控和开发新型肿瘤免疫疗法提供了理论基础。”哥伦比亚大学吴雪兵告诉生辉。
2007 年,吴雪兵本科毕业于清华大学自动化系,随后他留校从事生物信息学研究,采用网络生物学的方法大规模预测人类疾病相关的基因,就读期间他曾获得包括特等奖学金、学术新秀和优秀研究生等在内的多个奖项与称号。2009 年,在清华大学取得硕士学位后,吴雪兵赴美国麻省理工学院攻读计算系统生物学博士学位,师从诺贝尔奖得主 Phillip Sharp 教授,而他的研究领域也从纯计算转向了基于实验与计算相结合的方法探索分子生物学。
2014 年,吴雪兵博士毕业后在 Whitehead 生物医学研究所 David Bartel 教授实验室从事博士后研究,采用高通量组学探索 RNA 折叠对基因表达进行调控的新机制。2018 年底,吴雪兵在哥伦比亚大学创立了实验室,围绕 RNA 相关的基因调控机制及其在人类疾病和治疗方法中的应用等方面开展研究。截至目前,他已在 Cell、Nature、Science 等期刊发表论文 20 余篇。

▲图|哥伦比亚大学吴雪兵教授(来源:受访者)
01
揭示非编码序列翻译调控新机制
基因组是生命的蓝图,其包含了创造一个生命体所需的所有信息,而这些信息全都编码在由 A、C、G、T 四种碱基组成的 DNA 序列中。
谈及开展这项研究的初衷,吴雪兵坦言,他对人类基因组的非编码序列一直保持着浓厚的兴趣。“这些序列中只有很少一部分,大约只有 2% 是编码蛋白质的序列,也就是传统意义上的基因,而剩下的 98% 都被认为是非编码序列。”吴雪兵说道,“越是高等的生物,非编码序列在基因组中所占的比例越高。”他指出。
近 20 年来,DNA测序技术的发展使人类对于非编码序列有了很多新的认识。“其中有一个重要发现,这些非编码的序列跟编码的基因一样,也会被转录成 RNA,然后翻译成蛋白,尤其是在肿瘤以及阿尔茨海默病等疾病中。”他说道。
以肿瘤为例,绝大部分肿瘤的特异性抗原都是由非编码序列产生而来。“这些抗原是免疫系统识别和清除肿瘤细胞的关键,肿瘤细胞中这些非编码蛋白是如何产生和如何被处理成抗原是目前的热点问题。”他说道,“从更大层面上来看,为什么细胞内会产生这些非编码蛋白,它们有何种功能、如何被降解掉,以及其中一部分如何进化成有功能的新的蛋白基因等等,这些都是有待进一步探索的重要问题。”
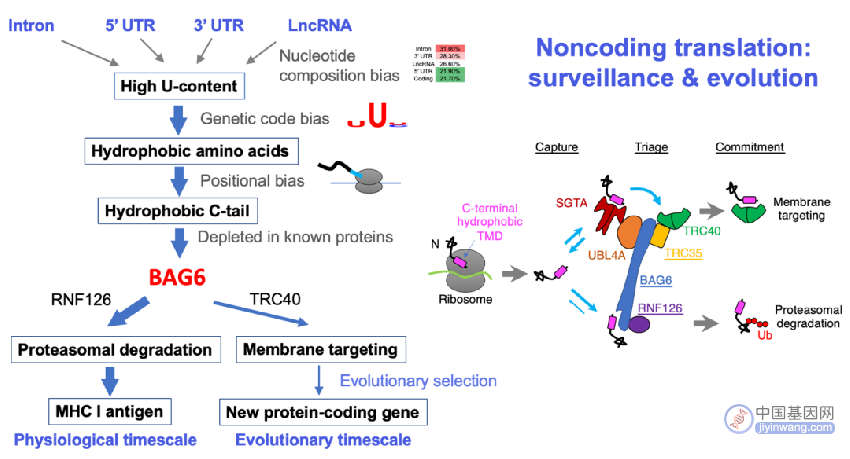
▲图|非编码序列翻译调控机制示意(来源:受访者)
先前研究表明,绝大部分非编码序列都被转录成为 RNA,包括长链非编码 RNA(lncRNA)、非翻译区(3'UTR 和 5'UTR)以及内含子(intron)。
在这项研究中,吴雪兵团队发现 lncRNA、intron、3'UTR、5'UTR 这些非编码序列翻译产生新蛋白的 C 端都是由非编码序列翻译而来并包含 C 端疏水区。
“通过大规模并行分析所有类型的上万个人源非编码序列,以及数百万个随机序列,我们发现,大部分非编码序列产生的蛋白都被细胞降解掉了。”吴雪兵说道。
具体而言,大部分未经过进化的非编码序列和随机序列都包含 C 端疏水区,同时 C 端疏水区导致蛋白被蛋白酶体降解。
为了进一步揭示降解机制,“我们通过分析 DNA 和蛋白序列的特征,以及系统性地敲除每一个基因,发现 BAG6 膜蛋白质量监控系统能够特异性地识别非编码蛋白上的 C 端疏水区,进而导致蛋白降解。”吴雪兵介绍道。
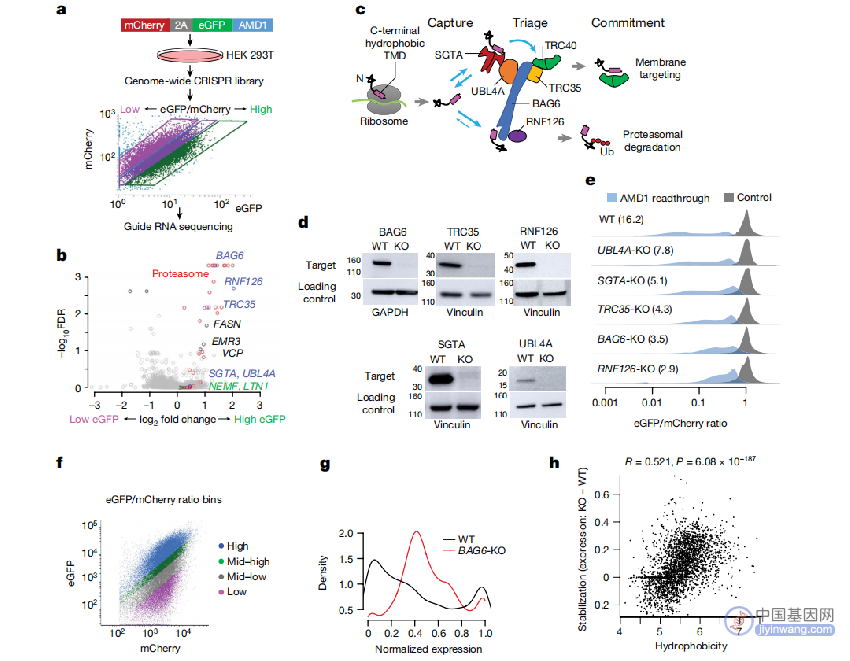
▲图|BAG6 介导非编码翻译产物的蛋白酶体降解(来源:Nature)
总的来说,这项研究揭示了人类基因组中编码序列和非编码序列的本质区别,以及在进化过程中两者能够相互转化的机制。“同时也为进一步探索非编码翻译在肿瘤、衰老和神经退行性疾病中的调控作用,以及新型肿瘤免疫疗法的开发打下了基础。”吴雪兵指出。
谈及下一步的研究动向,吴雪兵表示将继续探索非编码翻译在疾病中的调控和作用机制。“其中一个重点是围绕肿瘤,比如肿瘤细胞为什么有更多的非编码蛋白,以及这些非编码蛋白如何被处理成抗原。”他介绍说,“我们最新发现的这个机制可能被肿瘤细胞用于抑制抗癌基因,另外也可能会被肿瘤细胞抑制,从而达到不被免疫细胞识别和攻击的目的。因此,进一步深入研究有望为开发新的肿瘤药物和治疗方法提供基础。”他补充道。
02
目前正开发优化基于 RNA 的 CRISPR 技术
2003 年,吴雪兵考入清华大学自动化系。“这一年有个里程碑事件,即国际人类基因组计划宣布完成,虽然这与‘自动化’工程专业貌似毫不相关,但是当时系里的多位资深教授都在大力推动信息论、系统论和计算方法在生命科学尤其是基因组学中的应用。”他说道。
也正是从那时起,吴雪兵深受感染并开始自己学习分子生物学领域的知识。如今,他已经在哥伦比亚大学建立实验室,研究方向主要围绕“非编码序列”和“基于 RNA 的 CRISPR 技术开发”。

(来源:Nature)

(来源:Cell)
围绕“非编码序列”,早在 2013 年,吴雪兵和团队曾在 Nature 和 Cell 上各发表了一篇论文,分别阐述了非编码序列转录调控的机制和这一机制在新基因进化上的作用。
“简单来说,绝大部分非编码 DNA 序列都被转录成 RNA,这些 RNA 中的大部分都是基因组的“噪音”,因而细胞需要防止非编码序列的转录或者将其产物降解。”吴雪兵介绍道。
“我们的研究表明,编码和非编码序列在 A、C、G、T 四种碱基的组成上存在显著区别,这些区别被 RNA 剪接和多腺苷化相关的过程特异性地识别,进而抑制非编码转录。”他说道,“而在进化过程中,转录过程本身会导致 DNA 的突变,这些突变有一定的倾向性,通常会使被转录的非编码序列越来越像编码序列,最终可能产生新的编码功能蛋白的基因。”他指出。

(来源:Science)
围绕“CRISPR 基因编辑技术”,吴雪兵与 CRISPR 基因编辑先驱张锋教授曾有过多次合作,并参与了他们研究组在 CRISPR 方面的第一个工作,即 2013 年首次在人类细胞中实现 CRISPR 基因编辑。

(来源:Nature Biotechnology)
“之后在我领衔的一项研究中,我们首次系统性地研究了 CRISPR/Cas9 在整个基因组中的特异性。一个非常意外的发现是 Cas9 可以特异性地结合上千个 DNA 位点,但这些脱靶的结合并不导致 DNA 突变。”他指出,“此外,我们还发现了 gRNA 序列本身对 Cas9 特异性的巨大影响。总之,这些结果有助于降低 CRISPR/Cas9 系统的脱靶效应和提高这一系统的特异性。”他补充说。
关于 CRISPR 基因编辑系统,吴雪兵也表达了自己的观点,“目前大部分 CRISPR 技术是针对基因进行编辑,即直接改变 DNA。虽然基于包括碱基编辑(Base editing)和先导编辑(Prime editing)在内的 DNA 编辑技术理论上能一次性地纠正基因突变,进而一劳永逸地根治很多遗传疾病,然而这些技术目前存在一些局限性。”他指出,比如编辑效率和递送系统等的限制问题。
除了存在局限性,在吴雪兵看来,目前的基因编辑技术还面临两个根本性的难题。首先是安全性。“DNA 上的改变是永久性的,并且由于脱靶效应导致的突变可能会演变成肿瘤等疾病,相较之下,基于 RNA 的 CRISPR 技术则不会存在这个问题。”他说道。
其次是适应症。“其实很多疾病是不可逆的,即不能通过简单的纠正基因突变来进行治疗。比如基因突变导致器官和组织异常发育而产生的疾病。而针对这些疾病有望通过改变相关的下游基因表达来进行治疗,因此,基于 RNA 的 CRISPR 技术在这个方面也具有优势。”他解释说。
现阶段,吴雪兵实验室正通过综合实验和计算方法探索哺乳动物 mRNA 的调控机制,还在探索基于 RNA 的 CRISPR 技术靶向治疗疾病的治疗潜力,包括肿瘤、代谢疾病和心血管疾病等。谈及技术产业转化,吴雪兵表示,“我们目前还在进一步优化我们 RNA 和 CRISPR 相关的技术,其中一些具有较好的产业转化潜力,未来随着技术研究的不断成熟,我们可能会适时进行产业转化的探索和尝试。”他总结道。
参考资料:
1.https://doi.org/10.1038/s41586-023-05946-4
2.https://doi.org/10.1038/nature12349
3.https://doi.org/10.1016/j.cell.2013.10.048
4.https://www.science.org/doi/10.1126/science.1231143
5.https://doi.org/10.1038/nbt.2889
6.https://www.cuimc.columbia.edu/news/illuminating-tumor-cells-dark-proteins
7.https://xuebingwu.github.io/
8.https://datascience.columbia.edu/people/xuebing-wu/
声明:本站所有文章资源内容,如无特殊说明或标注,均为采集网络资源。如若本站内容侵犯了原著者的合法权益,可联系本站删除。