CRISPR基因编辑面临“血统难题”:非洲人遗传多样性导致脱靶效应

CRSIPR基因编辑技术已经诞生了10年时间,在这10年里,CRISPR基因编辑在全世界科学家的努力下达到了快速发展,已成为最简单高效的基因编辑工具,在基因功能研究、药物靶点筛选、遗传疾病治疗、癌症研究、作物育种等领域取得了突破性成就。
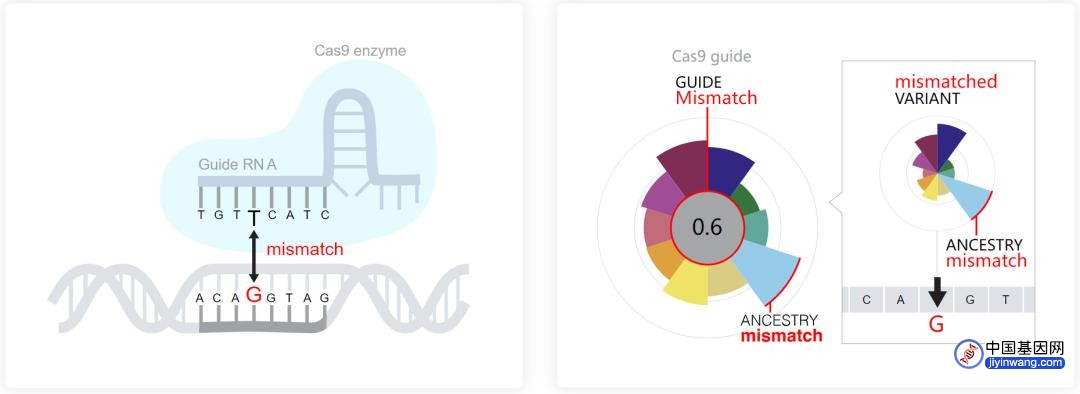
使用CRISPR技术进行基因组编辑时,首先需要设计一个向导RNA(gRNA)来识别和靶向我们需要编辑的目标基因组位点。在gRNA引导下,Cas酶到达正确的基因组位点并对DNA进行切割和编辑。如果gRNA与目标基因组位点匹配度不够,CRISPR系统可能就无法完成所需的编辑。
如今,已有多项基于CRISPR技术的基因疗法应用于人类临床试验,然而,实验小鼠通常是近亲繁殖而来、基因相同,但人类的基因组多样性较高,因血统不同而有所差异。
这些血统差异,意味着CRISPR并不总会像预期的那样编辑一些基因组位点,特别是在非洲血统人群中,因为他们的基因组可能与那些用来设计向导RNA(gRNA)的参考基因组不同。
近日,美国Broad研究所的研究人员在预印本平台 bioRxiv 发表了题为:Germline variation contributes to false negatives in CRISPR-based experiments with varying burden across ancestries 的研究论文。
该研究表明,由于在CRISPR基因编辑的gRNA设计过程中使用的参考基因组通常没有考虑血统问题,导致在癌症基因研究中可能会错过非洲血统人群的重要癌症靶点基因。该研究还开发了一款在线工具,帮助评估血统问题对标准CRISPR-gRNA的影响。

在设计向导RNA(gRNA)时,通常是基于从少数人基因组中测序编译的参考基因组,而这并不能代表人类基因组的多样性。例如,非洲血统的人比欧洲人和亚洲人的基因更具多样性,这是因为人类的祖先是在相对较近的时间里走出非洲大陆并传播到世界各地。
在这项研究中,研究团队借助“癌症依赖性图谱”(Cancer Dependency Map)来探索CRISPR基因编辑的“血统难题”。癌症依赖性图谱是英国Wellcome Sanger研究所和美国Broad研究所合作的大型研究计划,利用CRISPR基因编辑技术敲除1000株人类癌细胞中的约1.8万个基因,从中寻找与癌症生长或存活有关的基因,从而发现癌症治疗靶点。
这项研究发现,在这些癌细胞系中,CRISPR无法敲除其中2%-5%的基因,这种情况在非洲血统人群来源的细胞系中更常见,比其他群体来源的细胞系要多出20%左右。这意味着在这项“癌症依赖性图谱”计划,将会错过一些非洲血统人群的癌症治疗靶点。
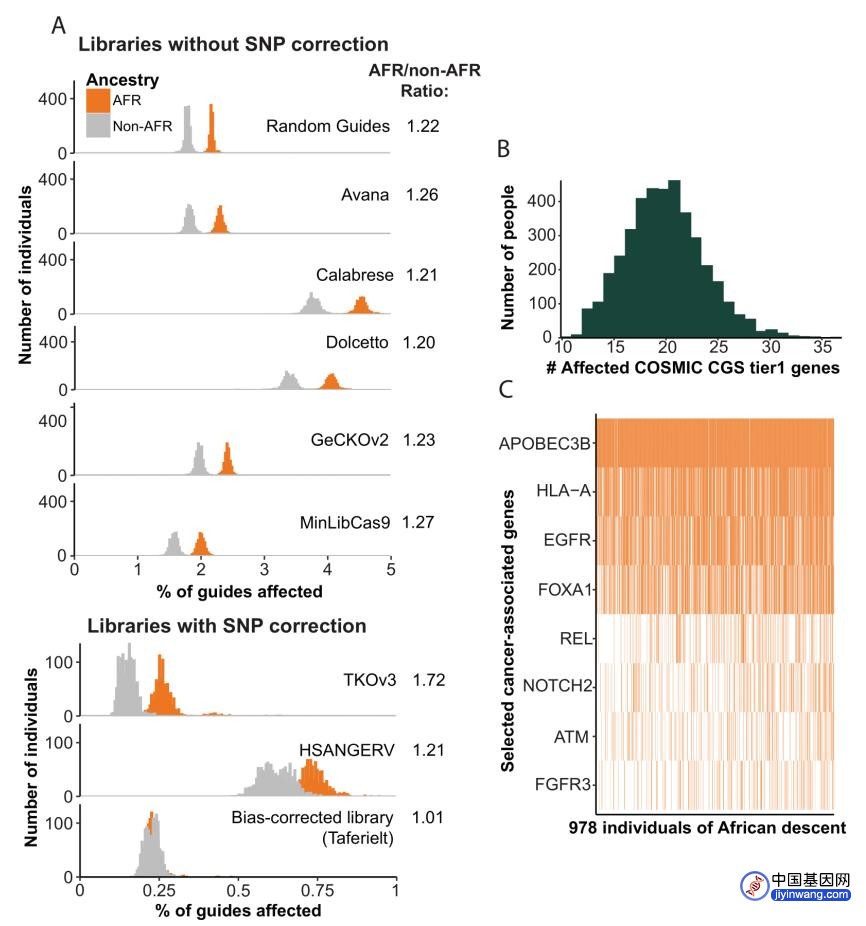
除此之外,还有一些研究显示,当时用CRISPR基因编辑治疗镰状细胞病(一种主要在非洲血统人群中出现的遗传疾病)时,根据参考基因组设计的gRNA会出现不匹配目标基因组位点的情况,从而导致在错误的位点切割基因组DNA,这种偏离目标基因组位点的编辑,可能会导致包括癌症在内的潜在风险。
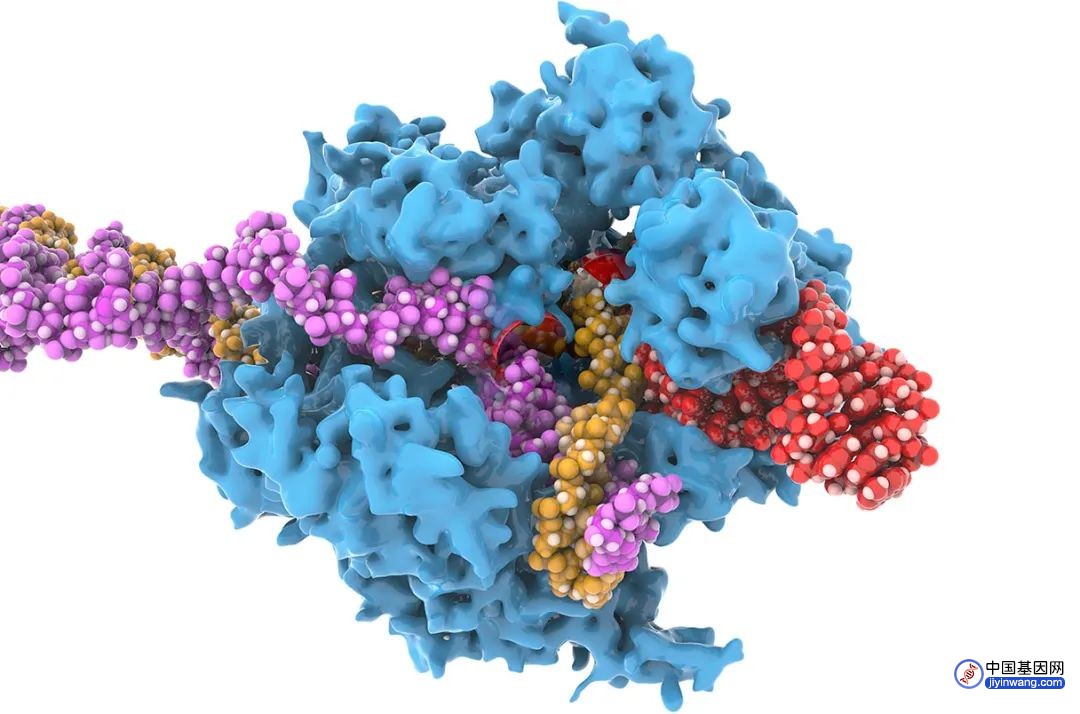
以CRSIPR-Cas9为例,由于人类基因组因血统不同而有所差异,gRNA(红色)有时与目标DNA序列(黄色)不匹配,从而导致Cas9酶(蓝色)无法完成所需切割与编辑。
基于这项研究,研究团队建立了一个免费的在线工具——Ancestrygarden.org,用于将设计的gRNA与从不同人群中获得的数万个基因组进行对比,从而评估血统问题对标准CRISPR-gRNA的影响。
这项研究能够促进我们重新思考如何在实验室中使用CRISPR技术,该研究开发的在线工具也将帮助更好地探索和理解人类基因组变异。
论文链接:
https://www.biorxiv.org/content/10.1101/2022.11.18.517155v1
声明:本站所有文章资源内容,如无特殊说明或标注,均为采集网络资源。如若本站内容侵犯了原著者的合法权益,可联系本站删除。