Science:绘制了史上最全面的哺乳动物DNA甲基化网络
比较表观基因组学是一个新兴起的研究领域,它将表观遗传标志与物种的系统发育关系相结合,以探索不同物种的特征,如最大寿命。以往的研究已经探索了DNA甲基化模式在调控序列中反映物种间进化关系的能力,但存在测序深度不足以及每个物种样本数量有限的限制。最近的研究虽然在基因启动子处比较了多种动物物种的甲基化数据,但并未专门针对保守的CpG位点展开详细研究。因此,深入了解DNA甲基化与最大寿命和系统发育之间的关系,仍是当下亟须解决的难题。

近日,来自加州大学洛杉矶分校大卫格芬医学院的Steve Horvath教授团队在国际顶刊《Science》上在线发表了一篇题为“DNA methylation networks underlying mammalian traits”的研究型文章(图1)。
为了探究DNA甲基化(DNAm)与最大寿命和系统发育之间的关系,研究人员开展了一项大规模的跨物种分析。通过在许多物种间进行比较研究,他们成功识别出了最大寿命和其他特征与表观遗传学之间的相关性,发现了DNA甲基化配置的分歧与遗传学在进化过程中紧密平行,DNA甲基化受到进化压力和选择的影响。这项研究为进化生物学、发育生物学和衰老研究等多个领域提供了丰富的公开数据资源,有助于深入理解物种特性和DNAm在其中的作用。

图1
这项研究采用了高测序深度的测量平台,生成了包括348个哺乳动物物种、来自70种组织类型的15,456个DNA样本数据集,以探究DNA甲基化与物种系统发育之间的关系(图2)。通过构建表观遗传树,研究人员发现在高度保守的DNA区域CpG位点的甲基化水平可以反映传统的系统发育树,这一结果经过了多项敏感性分析验证。此外,研究人员还使用了Blomberg的统计方法识别出一组在甲基化与系统发育树之间具有显著关联的CpG位点,特别是富集于上游间基因区域。
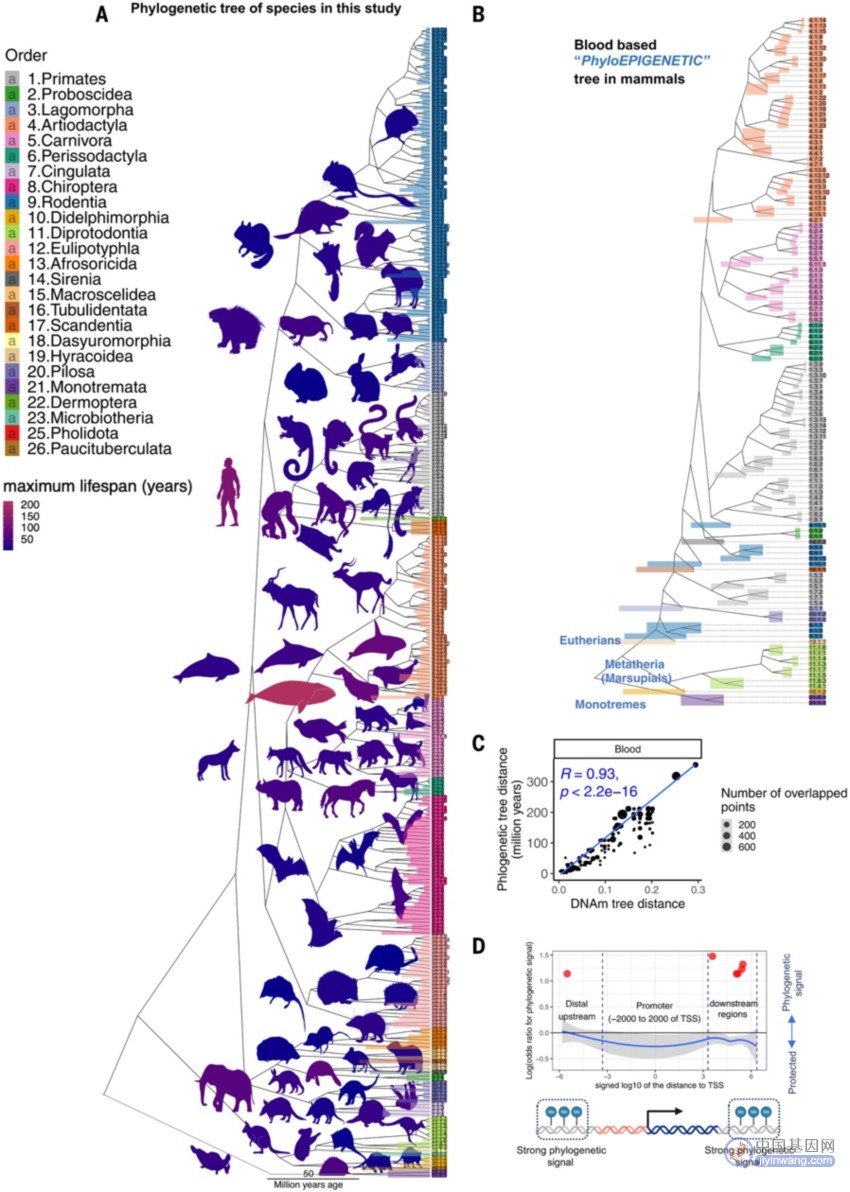
图2
研究人员使用WGCNA方法,将具有相似甲基化模式的CpG位点组成共甲基化模块,并用“模块特征基因”来总结这些模块的甲基化特征。这些数据分析分为两个阶段:首先,研究人员建立了一个共甲基化网络(Net1),使用了来自174个物种的11,099个DNA样本的数据。然后,他们还创建了一个独立的验证数据集,包括240个物种的4357个样本,验证了这些共甲基化模块的存在性。这些模块的保持统计数据显示了高度的一致性,证实了这些模块的可靠性。通过不同颜色来标记这些模块,最小的模块含有33个CpG位点,而最大的模块则包含1864个CpG位点。
利用高深度的测序平台,研究人员分析了来自348个哺乳动物物种的15,456个DNA样本的DNAm剖面,揭示了保守CpG位点上的甲基化与物种进化的关系(图3)。通过建立物种特异的phyloepigenetic树,验证了DNAm与传统系统发育树的高一致性。通过加权相关网络分析,他们鉴定了55个高度相关的甲基化模块,其中30个与物种特征(如分类目、组织类型、最大寿命、成年体重等)相关。这些模块的特征基因被发现与发育、免疫、代谢、生殖、干细胞、应激响应、衰老和信号通路等多种生物过程有关。
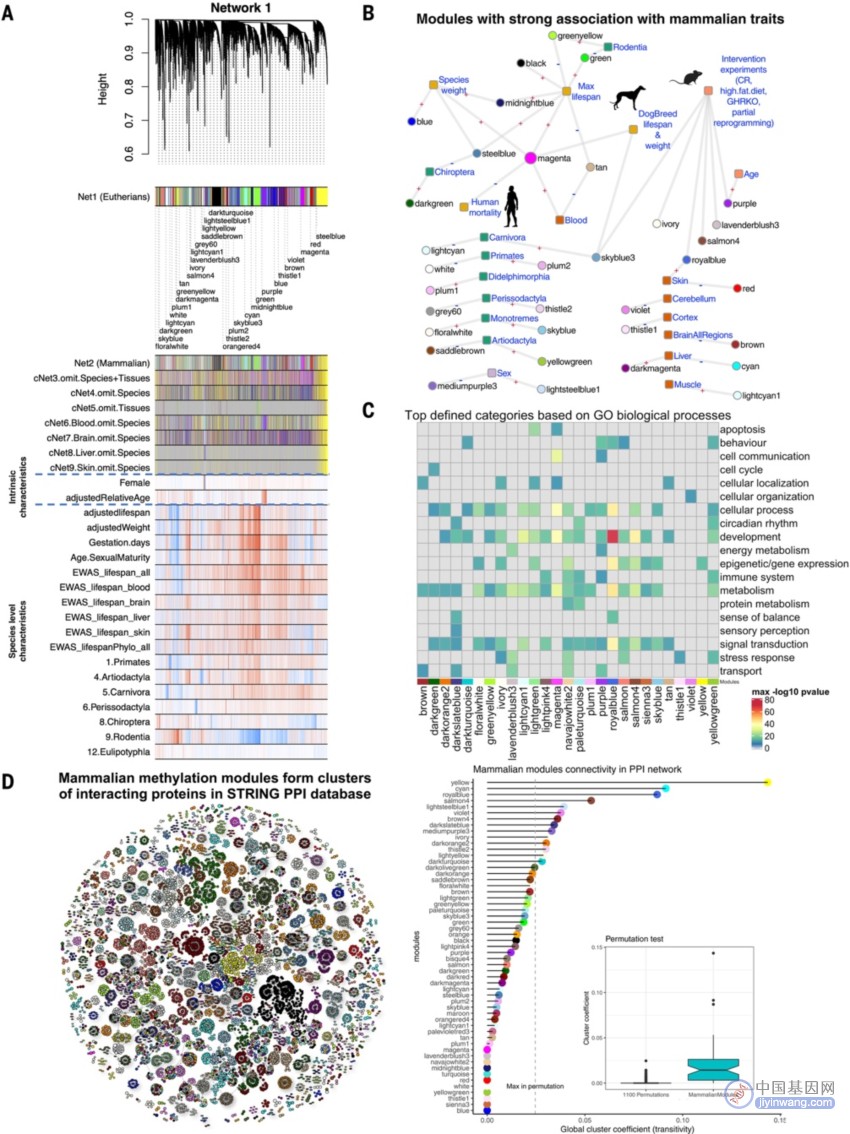
图3
随后,研究人员通过大规模的跨物种DNA甲基化(DNAm)分析,克服了测序深度和样本数量的限制。他们发现保守的CpG位点的甲基化水平在物种间存在密切的关联,构建了一种称为“phyloepigenetic tree”的系统发育关系。利用无监督分析方法,将具有相似甲基化动态的CpGs聚类成模块,这些模块与物种特征如分类、组织类型、性别、寿命等相关。其中,一些模块与最大寿命、平均成年体重等特征关联紧密。
随后,研究人员构建了一个紫色模块(RelativeAge+模块),该模块显示出与相对年龄之间的强正相关关系(图4)。为了排除物种和组织类型的混杂效应,他们还构建了共识网络,发现RelativeAge+模块在不同的共识网络中得到保留,表明其在不同物种和组织中具有保守性。该模块在调控区域富集,强调了胚胎干细胞调控、轴突束缚、血管生成和与糖尿病相关的途径。此外,研究人员使用基因型组织表达数据确定了一些调控因子,如OCT4、SHH、ASCL1、SOX2和NEUROG2蛋白,它们与RelativeAge+模块相关。
通过调查生长激素受体敲除、热量限制和高脂饮食等干预对小鼠寿命的影响,研究人员发现了与寿命相关的表观遗传学模块。其中,紫色模块(RelativeAge+)在侏儒小鼠和热量限制后显示出显著降低,而高脂饮食后略微增加。此外,通过对小鼠进行Yamanaka因子短暂表达实验,发现其中一些模块对于年龄的干预也表现出显著效应,尤其是与皮肤和肾脏中的细胞年轻化相关的模块。然而,最大寿命相关的模块与影响小鼠寿命的干预措施之间呈现出互斥关系,表明这两类模块之间存在差异。
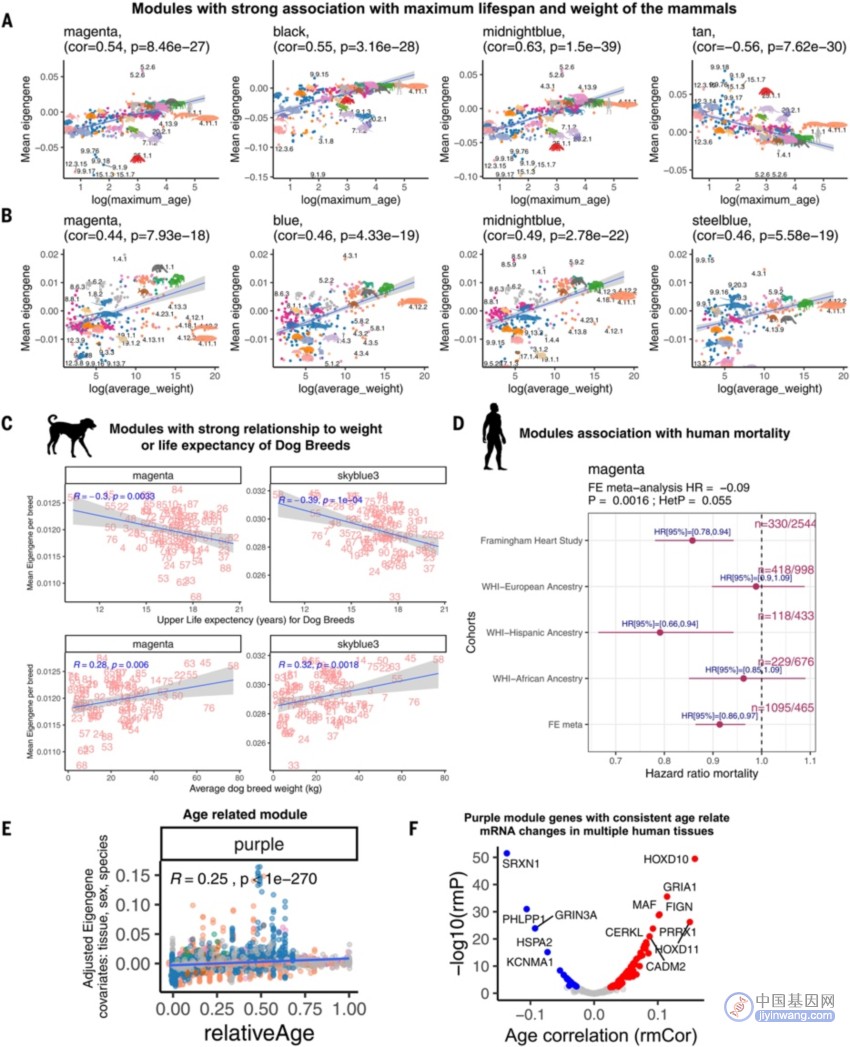
图4
接着,研究人员使用大规模的数据集,采用多种建模方法,识别了与最大寿命相关的甲基化位点,发现了一些显著的甲基化位点与最大寿命呈正相关或负相关,这些位点位于特定基因附近。他们发现与生物体最大寿命相关的甲基化位点并不随着哺乳动物组织的年龄而改变。即使限制在某个年龄组的动物中进行寿命的表观基因组关联分析(如仅限年轻动物),其结果也与全年龄动物的分析高度一致。研究人员鉴定了与寿命相关的甲基化位点附近的1006个基因,其中16个基因的表达水平在不同的人类组织中与年龄高度显著相关(图5)。这些寿命相关基因在动物器官形态发生中起关键作用,并在小鼠的肋骨数量增加中发挥作用,其中包括了HOXL亚类的家族基因,同时还与小鼠的异常生存有关。
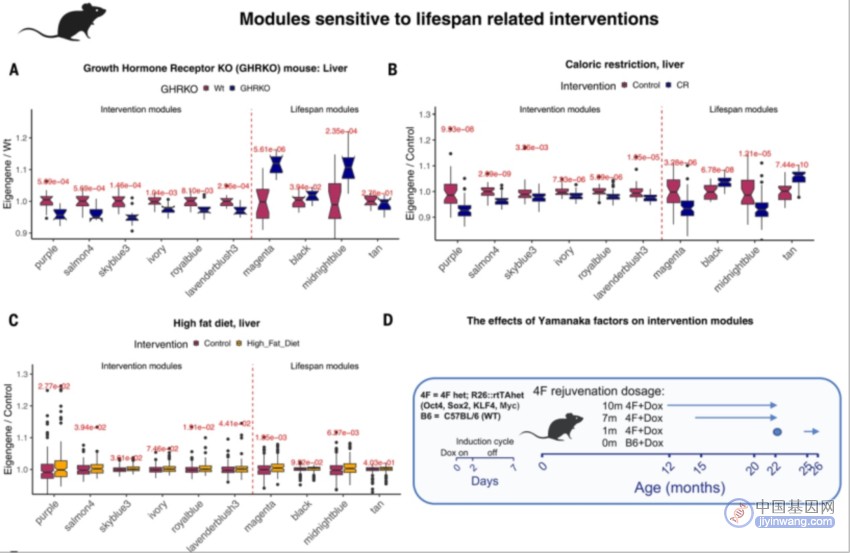
图5
研究人员还发现多个关键的甲基化位点,如TLE4、HOXA4和GATA3等,与最大寿命呈显著相关。这些关联在不同组织和年龄段中保持一致。这些与寿命相关的位点在基因组中有着特定的功能富集,涉及到生物体器官形态发生、RNA合成、小鼠肋骨数量增加等生物过程。他们还揭示了一些潜在的上游调控因子,如干细胞因子SOX2-OCT4-NANOG,以及与发育相关的因子如SHH、POU4F2和ASCL1。此外,研究人员还在鼠模型中发现,多能干细胞因子的瞬时表达会影响与寿命相关的基因的表达。
此外,研究人员发现与寿命相关的CpG位点主要分布在基因的启动子区域,这些位点的甲基化水平与寿命呈正相关(图6)。研究人员还发现某些共甲基化模块与寿命密切相关,这些模块在长寿物种中甲基化水平较低。进一步的染色质状态分析揭示了这些寿命相关CpG位点与染色质状态的关联,强调了其在基因表达调控中的重要性。
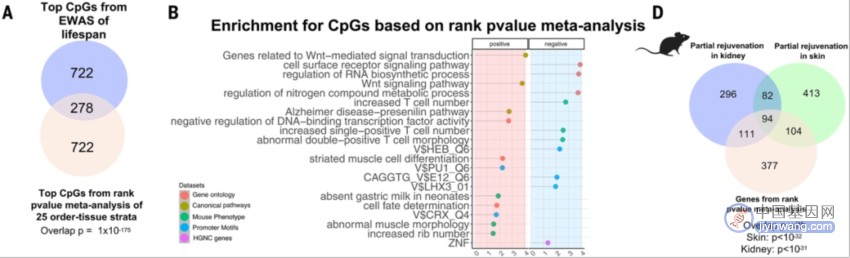
图6
另外,研究人员还通过数据分析和模型构建,探讨了多种因素对最大寿命的影响,包括基因表达、生物特征和环境干预等。他们发现一些共甲基化模块与动物的寿命和生存策略密切相关,揭示了这些模块在哺乳动物中的生命历程中扮演着关键角色。此外,通过染色质分析,研究还发现某些染色质状态与寿命相关的CpG位点之间存在显著的关联,为进一步探讨基因表达调控和寿命之间的关系提供了新的视角。这些研究结果为深入探究寿命的分子机制以及延长寿命的策略提供了重要的理论支持。
总结
这项研究通过分析跨物种DNA甲基化数据集,揭示了哺乳动物的甲基化模式。通过构建哺乳动物系统发育树,发现DNA甲基化的差异与进化中的遗传变化密切相关。一些CpG位点在长寿物种中具有显著的系统发育信号,这与启动子区域相对较低的信号形成对比。这些发现有望提供关于基因调控和生命寿命之间关系的新见解,拓展了我们对哺乳动物甲基化模式及其与生命寿命的关联的理解。
声明:本站所有文章资源内容,如无特殊说明或标注,均为采集网络资源。如若本站内容侵犯了原著者的合法权益,可联系本站删除。