首个Y染色体完整基因测序结果问世!填补人类基因组最后的空白
人类Y染色体中高度重复序列的普遍存在阻碍了它的完整组装,并导致它在基因组分析中被系统性地遗漏。
2023年8月23日,美国杰克逊基因组医学实验室Charles Lee团队在Nature 在线发表题为“Assembly of 43 human Y chromosomes reveals extensive complexity and variation”的研究论文,该研究揭示了跨越182900年人类进化的43条Y染色体的从头组装,并报告了大小和结构的相当大的多样性。
男性特异性常染区有一半发生大反转,其复发率比其他染色体高出两倍以上。与这些反转相关的扩增序列显示出不同的突变率,这些突变率与序列上下文相关,并且一些扩增基因显示出与谱系特异性假基因的获取和清除协同进化的证据。人类基因组中最大的异染色质区域Yq12由交替重复序列组成,在数量、大小和分布上表现出广泛的变化,但保持1:1的拷贝数比。最后,数据表明,重组假常染色体区域1与X和Y染色体的非重组部分之间的边界距离目前建立的边界500 kb。来自多个个体的完全序列解析的Y染色体的可用性为识别与特定Y染色体变异的新特征关联以及深入了解人类基因组复杂区域的进化和功能提供了独特的机会。

研究人员结合PacBio HiFi和ONT长读测序数据,组装了43个男性个体的Y染色体,代表了来自1000基因组计划的5个大陆群体。虽然GRCh38(主要是R1b-L20单倍群)和T2T Y组合代表欧洲Y谱系,但一半的Y染色体构成非洲谱系,并包括大多数根深蒂固的人类Y谱系。因此,这个新组装的43个Y染色体数据集提供了一个更全面的遗传变异视图,在核苷酸水平上,跨越18万年的人类Y染色体进化。
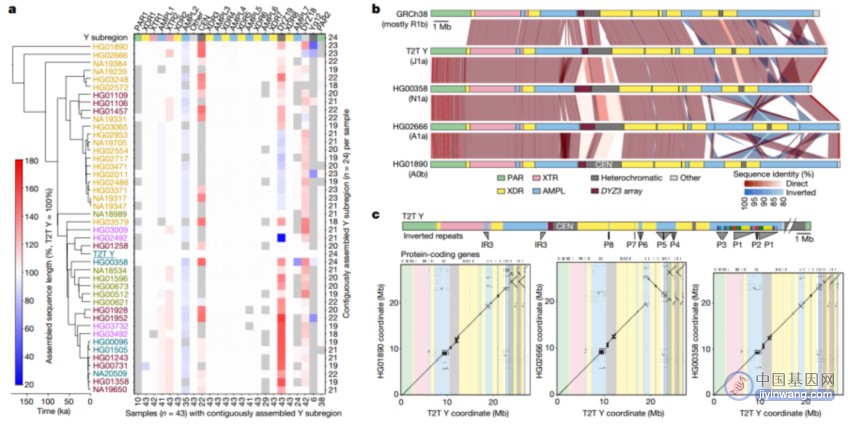
Y染色体的大小和结构变异(图源自Nature)
无独有偶,在同一时间(2023年8月23日),美国国立卫生研究院Adam M. Phillippy团队在Nature 在线发表题为“The complete sequence of a human Y chromosome”的研究论文,该研究揭示了来自HG002基因组的人类Y染色体的62,460,029个碱基对的完整序列(T2T-Y)。该序列纠正了GRCh38-Y中的多个错误,并在参考序列中增加了超过3000万个碱基对的序列,显示了基因家族TSPY、DAZ和RBMY的完整扩增结构;另外41个蛋白质编码基因,主要来自TSPY家族;以及在异色Yq12区域中人类卫星1和3块的交替图案。研究人员将T2T-Y与先前的CHM13基因组组合结合起来,绘制了可用的群体变异、临床变异和功能基因组学数据,为24条人类染色体提供了完整而全面的参考序列。

该文分析了一条完整Y染色体新组装的假常染色体区域(PARs)、扩增序列和回文序列、着丝粒卫星和q臂异染色质的组成。作者对T2T-Y进行了注释,并将其与之前的T2T-CHM13组合在一起,形成了一个新的、完整的人类所有染色体参考序列,T2T-CHM13+Y。为了使用这个新的参考序列,研究人员从ClinVar、GWAS、dbSNP和gnomAD中提取了可用的变异数据集。此外,还召回了1000基因组计划(1KGP)27和西蒙斯基因组多样性小组(SGDP)28数据中的变异,以及ENCODE数据中的表观遗传谱。这些实验表明,当使用T2T-Y作为参考时,XY个体的可映射性和变体调用得到了改善。
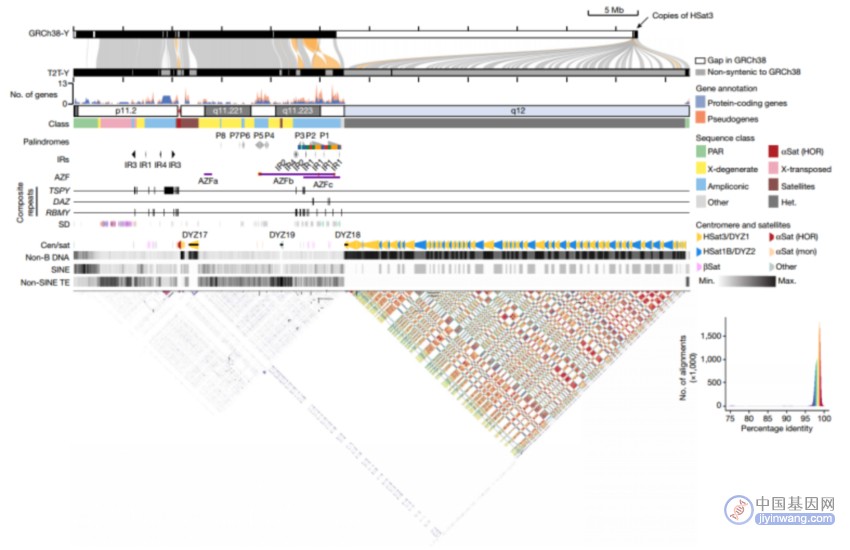
完整Y染色体的结构(图源自Nature)
综上,两篇文章共同揭示了Y染色体的组装和整体结构。这些不同的、完整的人类基因组的组装,以及类似质量的非人类灵长类动物的组装,将为人类变异和进化提供一个无与伦比的视角。这有助于人们全面了解人类遗传变异,也为将Y染色体序列与特定的人类特征联系起来以及更彻底地研究人类进化提供了起点。
文章来源“iNature”
声明:本站所有文章资源内容,如无特殊说明或标注,均为采集网络资源。如若本站内容侵犯了原著者的合法权益,可联系本站删除。