科学家发现9个中国人群自闭症候选基因并揭示高功能孤独症的遗传特性
孤独症谱系障碍(Autism spectrum disorder),又称自闭症,是一类神经系统发育疾病,主要临床特征为社交障碍和重复刻板行为,在中国发病率约为0.29%【1】。发育迟缓或智力障碍(ID/DD)是孤独症最常见的共病【2】。研究者通常将没有ID/DD症状的孤独症患者称为高功能孤独症,而与发育迟滞或智力障碍共病的称为低功能孤独症。目前针对中国孤独症人群开展的大规模全基因组与全外显子遗传研究相对较少,东亚其他国家也没有大规模孤独症的遗传学研究。

2023年7月7日中国科学院脑科学与智能技术卓越创新中心/上海交通大学医学院松江研究院仇子龙研究员团队和上海交通大学医学院附属新华医院李斐主任团队合作在期刊Biological Psychiatry杂志上发表题为Discovery and validation of novel genes in a large Chinese ASD cohort 的研究论文,首次报道了中国孤独症人群的大规模全外显子遗传学研究。

上海交大医学院附属新华医院教育部上海市环境与儿童健康重点实验室及发育行为儿保科李斐主任数年来收集了上千个孤独症病例,团队对患者进行了细致的行为学评估与智商发育商检测,对高功能与低功能孤独症有详细的分类。基于这些重要的临床数据,研究团队针对新华医院收集的772个中国孤独症核心家系进行了全外显子组测序研究,并结合团队之前报道的上海精神卫生中心369个中国孤独症核心家系数据【3】,共鉴定了22个高置信度的孤独症候选基因(FDR<0.1),其中包含9个未在欧美孤独症遗传学研究中发现的新孤独症候选基因(图A,红色字体为新候选基因)。本研究还将这些发现与目前已经发表最大的孤独症新发突变数据库整合分析后【4】,27个基因获得了全外显子组的显著性(FDR<0.1,图B),说明中国孤独症易感基因与欧美人群中的发现有较大不同。这一结果进一步丰富了人类孤独症候选基因列表,并证明在不同遗传背景的人群中进行孤独症遗传学研究对于全面理解孤独症遗传学基础的重要意义。
研究团队还与北京师范大学/中科院生物物理所王晓群教授团队合作,结合人类胚胎大脑皮层单细胞测序图谱【5, 6】,发现在高功能孤独症人群中发生新发突变的基因在一类人类特异的神经前体中丰富表达,此类细胞可能进一步发育为皮层深层神经元。而在低功能孤独症中发生新发突变的基因富集于另一类神经前体细胞中(图D)。这一结果证明了两类患者在遗传学及致病模式上存在潜在差异。
最后,研究者结合小鼠遗传学及行为学研究,验证了一个新孤独症候选基因-SLC35G1对小鼠社交行为的重要功能。相比于野生型小鼠,Slc35g1杂合子小鼠表现出孤独症样表型。
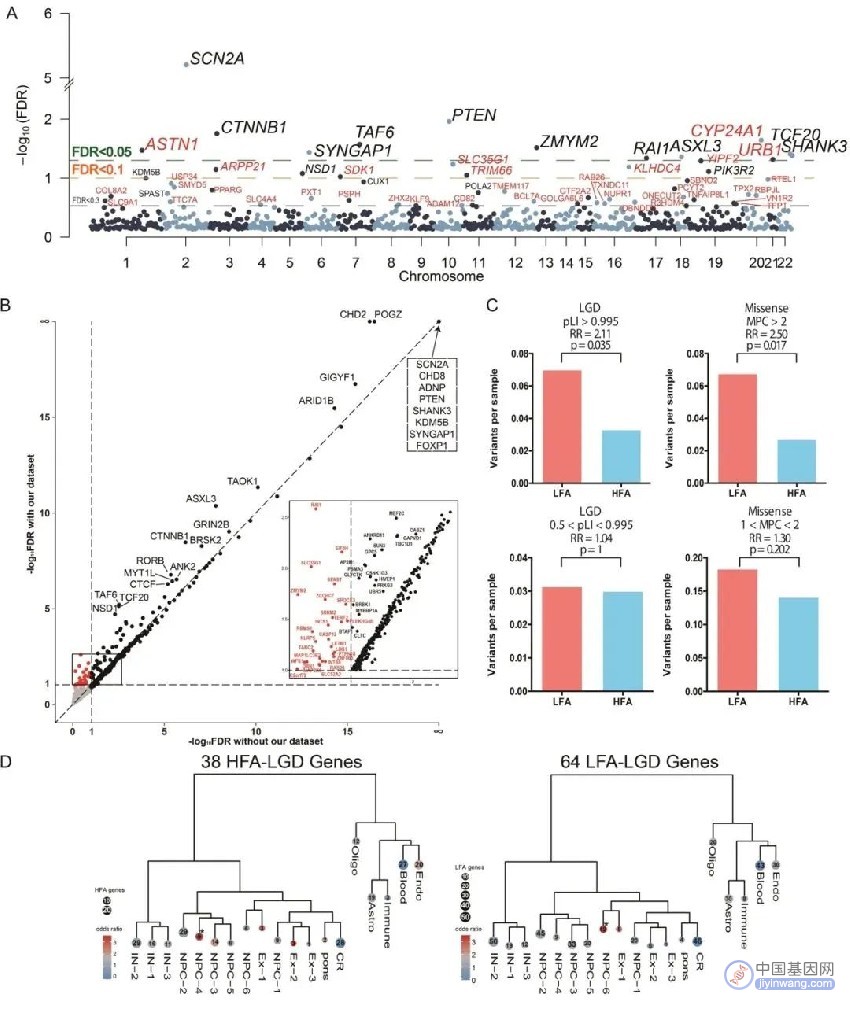
图A,用TADA模型分析了1141例孤独症新发突变的曼哈顿图。图B,数据库结合前后FDR。图C,两组孤独症人群突变统计。图D,人类胚胎大脑皮层单细胞测序图谱中主要细胞类型的分级聚类的树状图。
本研究是仇子龙与李斐合作团队在孤独症遗传学研究领域的又一成果,为全面理解孤独症遗传学结构,阐释高功能孤独症特性,进一步改善干预孤独症核心表型方案提供了新的思路。值得一提的是,为了整合中国孤独症研究的基础与临床力量,李斐与仇子龙研究员在2023年4月8日发起成立了“长三角孤独症研究中心”,本工作的主要作者及相关文章的主要作者杜亚松教授均为长三角孤独症研究中心成员,期待长三角孤独症研究中心继续整合各大医院的临床资源,做出有特色的孤独症研究,促进孤独症的临床诊治。
中国科学院脑科学与智能技术卓越创新中心/上海交通大学医学院松江研究院仇子龙研究员和上海交通大学医学院附属新华医院李斐主任为该论文共同通讯作者。中国科学院脑科学与智能技术卓越创新中心王锦程博士,上海交通大学医学院附属新华医院俞珏华研究员,中国科学院生物物理研究所博士生王梦迪,上海交通大学医学院附属新华医院章玲丽青年研究员,上海交通大学医学院附属新华医院杨侃青年研究员为论文共同第一作者。上海交通大学医学院附属新华医院杜秀娟博士,温州医科大学吴金雨研究员,北京师范大学王晓群教授共同参与了此研究。
原文链接:
https://doi.org/10.1016/j.biopsych.2023.06.025
参考文献
1 Zhou, H. et al. Prevalence of Autism Spectrum Disorder in China: A Nationwide Multi-center Population-based Study Among Children Aged 6 to 12 Years. Neurosci Bull 36, 961-971, doi:10.1007/s12264-020-00530-6 (2020).
2 Mottron, L. A radical change in our autism research strategy is needed: Back to prototypes. Autism Res 14, 2213-2220, doi:10.1002/aur.2494 (2021).
3 Yuan, B. et al. Identification of de novo mutations in the Chinese ASD cohort via whole-exome sequencing unveils brain regions implicated in autism. Neurosci Bull doi: 10.1007/s12264-023-01037-6. (2023).
4 Zhou, X. et al. Integrating de novo and inherited variants in 42,607 autism cases identifies mutations in new moderate-risk genes. Nat Genet 54, 1305-1319, doi:10.1038/s41588-022-01148-2 (2022).
5 Fan, X. et al. Spatial transcriptomic survey of human embryonic cerebral cortex by single-cell RNA-seq analysis. Cell Res 28, 730-745 (2018).
6 Fan, X. et al. Single-cell transcriptome analysis reveals cell lineage specification in temporal-spatial patterns in human cortical development. Sci Adv 6, eaaz2978 (2020).
声明:本站所有文章资源内容,如无特殊说明或标注,均为采集网络资源。如若本站内容侵犯了原著者的合法权益,可联系本站删除。