巨型病毒的基因组保护措施:由无接头的核小体紧密包装
一些古细菌组蛋白是具有两个组蛋白折叠结构域的双联体,被认为是早于真核生物核小体进化的形式,虽然它们与真核核小体的包装类似,但与真核组蛋白的序列差异过大,无法识别与组蛋白双联体特定结构域的对应关系。2007年对巨型马赛病毒的鉴定及测序让人们找到它们在真核组蛋白中的配对同源物,马赛病毒中的Hβ-Hα与真核H2B和H2A同源,Hδ-Hγ与H4和H3同源【1, 2】。生化重组和冷冻电镜的高分辨率结构解析显示马赛病毒核小体和真核细胞核小体惊人的相似之处,与包裹147 bp DNA的八聚体真核核小体不同,四聚体马赛病毒核小体仅包裹121 bp的DNA。由于马赛病毒双联体组装成未被DNA完全包裹的核小体,因此有人提出它们本质上是不稳定的,这可能是为了促进感染早期阶段的表达或基因调控【3】。然而,这些研究是使用核小体定位序列和交联进行的,并且没有分离天然形式的病毒染色质,重组的马赛病毒核小体在多大程度上代表它们在病毒粒子中的构象仍然是有争议的。
2022年11月11日,来自弗雷德·哈金森癌症研究中心的Steven Henikoff团队和纽约大学格罗斯曼医学院的Karim-Jean Armache团队在Molecular Cell杂志上合作发表了一篇题为A giant virus genome is densely packaged by stable nucleosomes within virions 的文章,通过透化马赛病毒病毒粒子并进行全基因组核酸酶消化、化学切割和质谱分析,他们发现马赛病毒染色质的组织与真核生物的根本不同之处。马赛病毒核小体完全保护病毒粒子内的DNA,因为它是紧密相邻的121 bp DNA包裹核心,没有接头DNA或沿基因相移。此外,他们还观察到在核小体定位序列的多拷贝串联重复上重组的核小体是紧密排列的,而不是采取带有基因标点的“串珠”形式,代表了马赛病毒组蛋白包装DNA的一种独特模式。

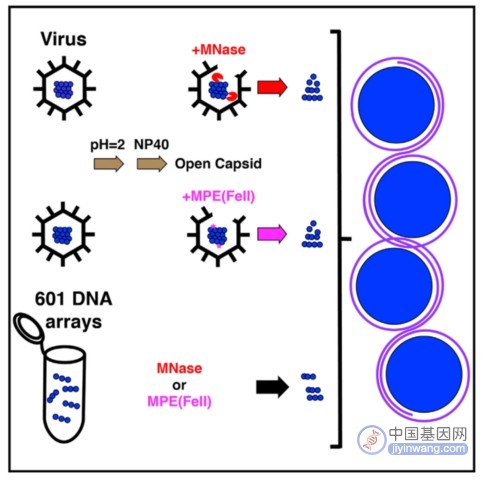
作者首先参考Schrad等人的方案【4】在低pH条件下透化并在中性缓冲液中进行微球菌核酸酶(MNase)消化后获得高质量染色质,为了确定染色质的蛋白质组成,作者离心病毒悬浮液,然后从沉淀、上清液和洗涤液中提取总蛋白质,经过SDS-PAGE/银染、胰蛋白酶消化和质谱等步骤鉴定了Hβ-Hα和Hδ-Hγ的存在。紧接着,作者对病毒片段进行了双末端DNA测序,并使用Bowtie2将它们与 GenBank T19基因组组装进行比对。MNase释放的马赛病毒单核小体DNA片段比果蝇的小,并且核小体之间的间距要小得多,相较于真核核小体包裹147 bp DNA,马赛病毒的包装更为紧密,由没有接头的121 bp 核小体彼此相邻组成,且足以抵抗小分子DNA切割试剂MPE(FeII) 的插入和切割。
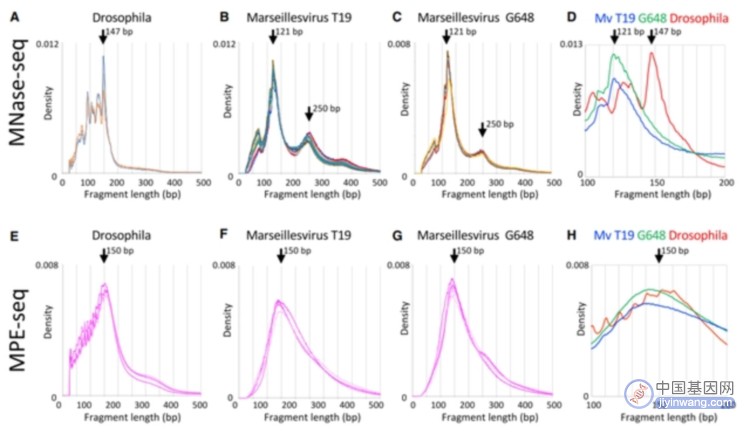
图1. MNase和MPE消化的马赛病毒染色质产生紧密排列的核小体大小的DNA片段
接下来,作者对齐了所有191个在其5’末端为R600 bp的ORF(开放阅读框)并对基因体上每个核苷酸位置进行平均。在果蝇中,MPE生成的片段对101-200-bp片段显示相同的5’对齐的平移定相模式,证实使用MPE和MNase生成的图谱与对照真核核小体大致一致,相比之下,马赛病毒T19和G648 101-200 bp片段在以相同比例绘制时几乎没有5’相位。此外,与果蝇 ORF 3’端的染色质也不同,T19和G648染色质均在3’端的上游显示出MNase抗性的平均峰值。
之前工作【5】中在单个601序列上重组马赛病毒核小体未能显示出完整的包装是否是由于缺乏相邻的核小体呢?因此,作者对已以60或300 μg/mL浓度组装到3拷贝601阵列和12拷贝601阵列上的天然和交联重组马赛病毒染色质进行MPE和MNase消化。在 DNA 提取之后,准备测序文库并进行了双末端测序,并将来自天然或交联的3拷贝和 12 拷贝染色质消化物的片段与 3 拷贝 601 阵列进行比对。此外,作者还在包含7拷贝Widom 601序列和12 bp接头的质粒DNA上重建了马赛病毒和爪蟾核小体。整体结果表明马赛病毒核小体遵循与真核核小体相同的核小体包裹规则,但双组蛋白已经进化为包裹更少的DNA以促进密集的核小体包装,而无需中间的接头。
总之,这项工作证明了巨型马赛病毒的双重组蛋白将121 bp的DNA紧紧包裹在病毒粒子中且缺乏接头序列,这种独特的染色质形式表明病毒粒子内的保护适应性。有趣的是,最近人们对于真核细胞核小体其实是由病毒工厂进化而来的说法很感兴趣【6】,如果真的是这样,紧密排列的病毒粒子如何转变为一个功能完备的病毒工厂的机制可能为真核生物进化的早期阶段提供线索。
原文链接:https://doi.org/10.1016/j.molcel.2022.10.020
参考文献
1. Boyer, M., Yutin, N., Pagnier, I., Barrassi, L., Fournous, G., Espinosa, L., Robert, C., Azza, S., Sun, S., Rossmann, M.G., et al. (2009). Giant Marseillevirus highlights the role of amoebae as a melting pot in emergence of chimeric microorganisms. Proc. Natl. Acad. Sci. USA 106, 21848–21853.
2. Erives, A.J. (2017). Phylogenetic analysis of the core histone doublet and DNA topo II genes of Marseilleviridae: evidence of proto-eukaryotic provenance. Epigenetics Chromatin 10, 55.
3. Liu, Y., and Krupovic, M. (2022). Genome chromatinization in giant doublestranded DNA viruses. Trends Biochem. Sci. 47, 3–5.
4. Schrad, J.R., Abraha˜ o, J.S., Cortines, J.R., and Parent, K.N. (2020). Structural and proteomic characterization of the initiation of giant virus infection. Cell 181,1046–1061.e6.
5. Valencia-Sa´ nchez, M.I., Abini-Agbomson, S., Wang, M., Lee, R., Vasilyev, N., Zhang, J., De Ioannes, P., La Scola, B., Talbert, P., Henikoff, S., et al. (2021). The structure of a virus-encoded nucleosome. Nat. Struct. Mol. Biol. 28, 413–417.
6. Talbert, P.B., Armache, K.J., and Henikoff, S. (2022). Viral histones: pickpocket’s prize or primordial progenitor? Epigenetics Chromatin 15, 21.
声明:本站所有文章资源内容,如无特殊说明或标注,均为采集网络资源。如若本站内容侵犯了原著者的合法权益,可联系本站删除。