Nature:薛愿超团队揭示基因组重复序列Alu调控转录新机制
人类基因组含有大量的重复序列和转录调控元件,而重复序列过去常被认为是“无用”DNA(Junk DNA)。增强子作为调控基因转录的重要元件,往往需要通过远距离染色质环化与目标启动子进行相互作用,从而决定基因的时空表达特异性。这一过程不仅参与了细胞命运的决定,还在多种疾病的发生发展中扮演着重要角色。增强子能够精确地选择将要激活的目标启动子,以调节靶基因的转录激活水平。然而,增强子和启动子配对选择的特异性是如何实现的一直是未解之谜。
2023年7月12日,中国科学院生物物理研究所薛愿超团队在 Nature 期刊发表了题为:Complementary Alu sequences mediate enhancer–promoter selectivity 的研究论文。
该研究发现,增强子(enhancer)和启动子(promoter)非编码RNA中反向互补的Alu序列,可通过碱基配对决定增强子-启动子的配对选择特异性。

薛愿超团队利用实验室之前开发的RNA原位构象测序技术RIC-seq(Nature,2020),系统捕获了增强子RNA和启动子来源的非编码RNA之间的相互作用,并构建了高分辨率增强子-启动子RNA互作(EPRI)图谱,该图谱共涵盖了7种人源细胞系中178,434个高可信的增强子-启动子互作对。相较于传统的染色质构象捕获技术如Hi-C、HiChIP、ChIA-PET等,RIC-seq技术在解析增强子-启动子互作位点的详细序列特征方面具有更高的分辨率。
基于EPRI图谱,研究人员发现相互作用的增强子和启动子RNA片段处富集Alu重复序列。Alu序列是哺乳动物以及人类基因组中广泛分布的一类重复序列,它们在基因转录调控中的作用仍不清楚。研究人员利用CRISPR-Cas9系统分别敲除增强子-启动子互作对中的Alu元件,发现靶标基因的转录水平显著下调,定量染色体构象捕获实验(3C-qPCR)结果也表明配对的增强子和启动子之间的空间邻近接触显著减少。反过来,在启动子区人为敲入Alu元件或利用CRISPR-dCasRx系统将Alu RNA序列靶向递送到不受调控的启动子RNA,都能够使原本不受特定增强子调控的基因被激活,并且该启动子与增强子之间的染色质空间接触也显著增加。这些结果表明增强子和启动子RNA中的Alu序列可通过碱基互补配对形成RNA双链体,进而决定了增强子-启动子的配对选择特异性。这一发现为研究基因组中重复序列的功能指明了新方向。
此外,为了进一步探索EPRI的功能以及对疾病发生发展的影响,研究人员将非编码区风险变异位点映射到增强子-启动子互作图谱,发现有超过53万个突变落在启动子和增强子RNA相互作用位点,提示这些突变可能破坏EPRI导致基因异常表达,从而引发疾病。因此,该研究团队又在EPRI图谱的基础上构建了“突变-功能”图谱,系统地注释了非编码突变(尤其是Alu元件的删除和插入)影响的靶标基因以及可能的致病机制,为理解疾病发展的潜在机制提供了宝贵的资源。
研究团队进一步发现,位于Alu元件中的突变会影响数千个蛋白编码基因的表达,从而对细胞功能产生重要影响。如癌基因PTK2增强子中的多态性Alu元件删除能够显著降低细胞增殖和侵袭能力,这为Alu元件突变与癌症易感性之间的关联研究提供了重要线索。
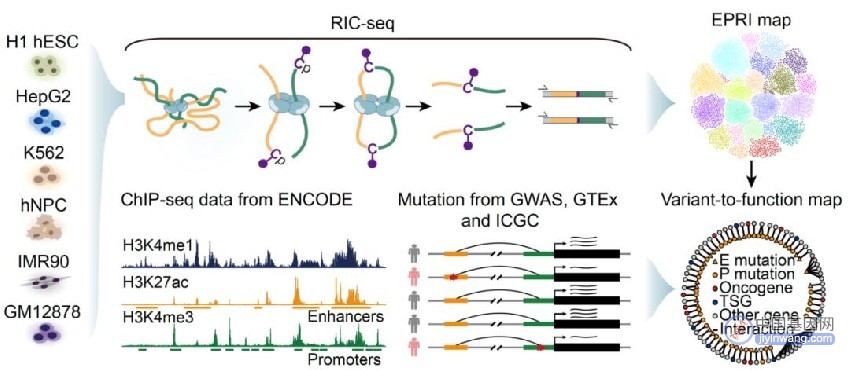
“增强子-启动子互作图谱”以及“突变-功能图谱”构建
该研究工作在新的层面上揭示了增强子-启动子配对选择特异性的原则,并系统的将非编码风险变异与其分子功能联系起来,为我们深入理解基因表达调控的分子机制以及疾病发生发展提供了新的视角。
论文链接:https://www.nature.com/articles/s41586-023-06323-x
声明:本站所有文章资源内容,如无特殊说明或标注,均为采集网络资源。如若本站内容侵犯了原著者的合法权益,可联系本站删除。