小RNA和核蛋白Hfq通过结合新生RNA调控基因表达
一些小的核糖核蛋白(small ribonucleoproteins,sRNPs)在转录过程中作为分子伴侣与目标RNAs组装在一起,促进了RNA的化学修饰和加工,以及调控了目标RNA的表达【1】。一些RNA介导的酶,比如CRISPR-Cas,能够靶向成熟的RNAs【2】。而另外一些sRNPs则能够作用于未成熟的转录产物,它们通过引导RNA与未成熟转录RNA相互作用时,首先要与RNA的延伸和折叠相协调。
sRNPs若要与目标RNA结合必须要改变目标RNA的结构或者在其折叠之前与之结合,否则RNA的二级结构将对两者的结合产生阻碍【3,4】。而sRNPs与目标RNA的结合会受到转录速度以及RNA在转录过程中发生折叠的可能性的支配【5】。随着转录的延伸,新生RNA链会形成各种二级或者三级结构,一些sRNPs的结合位点将被这些高级结构掩盖【6】。最近一些研究表明,转录过程中新生RNA的一些可变折叠对于RNA稳定结构的形成,以及sRNPs的结合有着重要作用,但具体分子机制还不清楚。
2023年4月27日,来自美国约翰霍普金斯大学的Sarah A. Woodson和Margaret L. Rodgers团队在Molecular Cell发表了文章Small RNAs and Hfq capture unfolded RNA target sites during transcription,他们在细菌Hfq·DsrA·rpoS系统中,对sRNPs在sRNA引导下与新生RNA的结合位点的识别机制进行了详细探索,阐明了影响这一生物学过程的关键因素。

在大肠杆菌中,Hfq是一个核蛋白,DsrA是一段小RNA(sRNA)。在细菌的指数增殖期,rpoS基因的5’ UTR区域能够折叠为一个抑制性二级结构,并掩盖掉核糖体结合位点(ribosome binding site, RBS),进而抑制基因的表达。而DsrA能够与rpoS基因的5’ UTR区域结合,解开抑制性二级结构,释放RBS位点,促进基因表达。分子伴侣Hfq能够促进DsrA与rpoS 5’ UTR的结合,并形成一个三聚体复合物。
为了研究sRNA与新生mRNAs的结合,研究人员采用了单分子共定位共转录组装平台(single-molecule co-localization co-transcriptional assembly,smCoCoA)对Hfq·DsrA介导的rpoS基因表达过程进行了实时检测。研究人员首先选用rpoS基因的全长5’ UTR区域进行检测,并在5’ UTR的末端添加了Cy3荧光标记,当大肠杆菌中的RNAP接近Cy3荧光基团时(1-3 nm之内),Cy3发出的荧光会有大幅度增强,出现PIFE(protein-induced fluorescence enhancement)现象(图1)。
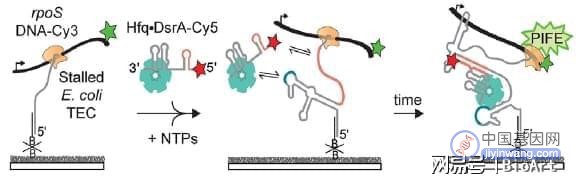
图1 smCoCoA研究系统
为了观察Hfq·DsrA的作用,研究人员还在DsrA sRNA的5’端标记了Cy5荧光基团,并提前将Hfq与DsrA预结合在一起。在实验中,研究人员观察到,Cy5荧光和Cy3荧光信号在PIFE信号出现之前发生共定位。另外,在rpoS的5’ UTR区完全转录完毕之前,约67%的rpoS 5’ UTR转录本至少和一个Hfq·DsrA-Cy5分子结合。接下来,研究人员发现,截去(AAN)上游300 bp左右的核苷酸序列(rpoS301)并不影响Hfq-DsrA复合体对rpoS 5’ UTR的转录调控,依然有至少77%的rpoS 5’ UTR转录本至少和一个Hfq·DsrA-Cy5分子结合。于是,接下来的实验中,研究人员选用rpoS301作为研究对象。
利用极大似然估计(maximum likelihood analysis),研究人员检测到Hfq·DsrA-Cy5与rpoS301转录本存在三个不同的时间(0.5 s; 5 s; >100 s),这说明至少存在三种不同形式的Hfq·DsrA·rpoS复合体。根据以前的研究推测,检测时间>100 s代表了DsrA和rpoS mRNA之间形成了稳定结合的长期存在复合体(long-lived complex)。研究人员检测发现,全长rpoS 5’ UTR转录本和rpoS301转录本中长期存在复合体形成的比率相近,分别为48%和50%,这也说明了两种不同截断体的转录效率是相似的。
Hfq能够导致rpoS 5’ UTR区域结构重塑,以促进DsrA与抑制性颈环上结合位点之间的退火配对,并因此释放RBS序列。结构重塑会对sRNA的靶向配对以及之后的基因表达产生动力学阻碍。那么,转录期间的靶向配对是否会比转录后的靶向配对更有利于基因表达呢。研究人员通过实验发现,当对转录后的靶向配对进行检测时,稳定复合体形成的比率比转录期间的要低很多,说明转录期间的靶向配对更有效率。
接下来,研究人员对DsrA是否受到转录速度的影响进行了检测。研究人员将大肠杆菌RNAP替换为转录速度快4-7倍的T7 RNAP,发现Hfq·DsrA的结合降低了2倍左右。另外,研究人员发现在5’ UTR上游的AAN motif是Hfq结合必须的,删除这段序列会降低DsrA稳定复合体的形成比率,同时,这段AAN基序必须在靶点序列的上游才能发挥作用,将这段序列转移到靶点序列的下游同样会导致稳定配对的失败。除此之外,将AAN基序和下游靶点序列之间的碱基删除同样会导致稳定结合效率的降低。
为了探究稳定复合体形成的具体位点,研究人员在靶点序列后面又添加了一个Cy3基团,实验显示,当靶点序列被转录成功后,稳定复合体的形成比率超过了80%,这说明稳定复合体的形成主要依赖于靶点序列的转录成功。
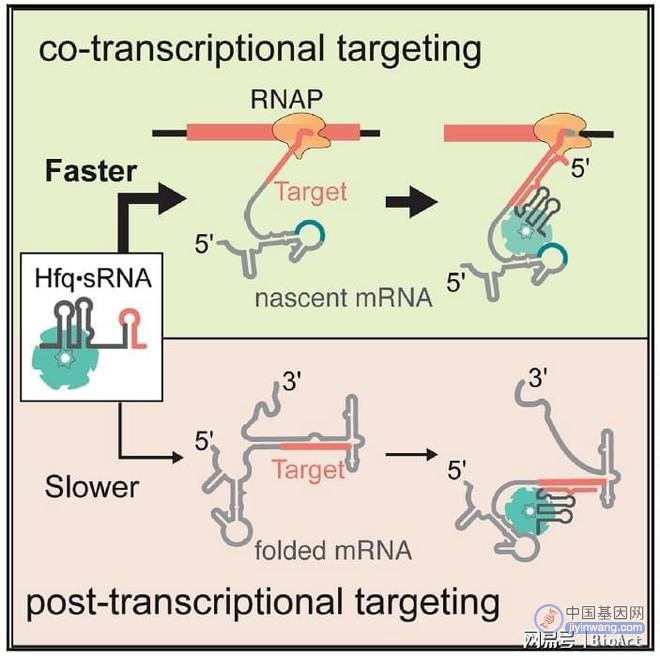
综上所述,本研究对大肠杆菌中Hfq·DsrA·rpoS系统进行了详细的研究,发现了Hfq·DsrA通过改变新生RNA二级结构进而调控目的基因表达的分子机制。
https://doi.org/10.1016/j.molcel.2023.04.003
参考文献
1. Gerovac, M., Vogel, J., and Smirnov, A. (2021). The world of stable ribonucleoproteins and its mapping with grad-seq and related approaches.Front. Mol. Biosci.8, 661448.
2. Gorski, S.A., Vogel, J., and Doudna, J.A. (2017). RNA-based recognition and targeting: sowing the seeds of specificity.Nat. Rev. Mol. Cell Biol.18, 215–228.
3. Oesterreich, F.C., Herzel, L., Straube, K., Hujer, K., Howard, J., and Neugebauer, K.M. (2016). Splicing of nascent RNA coincides with intron exit from RNA polymerase II.Cell165, 372–381.
4. Steitz, J.A., and Tycowski, K.T. (1995). Small RNA chaperones for ribosome biogenesis.Science270, 1626–1627.
5. Rodgers, M.L., and Woodson, S.A. (2021). A roadmap for rRNA folding and assembly during transcription.Trends Biochem. Sci.46, 889–901.
6. Scull, C.E., Dandpat, S.S., Romero, R.A., and Walter, N.G. (2020). Transcriptional riboswitches integrate timescales for bacterial gene expression control.Front. Mol. Biosci.7, 607158.
声明:本站所有文章资源内容,如无特殊说明或标注,均为采集网络资源。如若本站内容侵犯了原著者的合法权益,可联系本站删除。