Cell综述:张泽民总结肿瘤基因组学的发展历程以及未来发展方向
肿瘤基因组学(Cancer genomics)旨在系统性地发现肿瘤发生发展过程中的分子驱动机制,理解肿瘤异质性的生物学基础,并据此提供个体化的精准治疗策略。过去20年,得益于高通量测序技术的发展与普及,肿瘤基因组学推动了肿瘤生物学的革命性变化,极大丰富了我们对癌细胞自身、以及整个肿瘤微环境的理解,并为肿瘤治疗提供了新的可行方案。

2023年4月13日,北京大学生物医学前沿创新中心(BIOPIC)张泽民教授受邀于Cell发表了题为Accelerating the understanding of cancer biology through the lens of genomics 的综述文章,全面总结了肿瘤基因组学的发展历程以及其对理解肿瘤驱动机制与异质性、促进个体化精准肿瘤治疗的重要贡献,并强调了目前肿瘤基因组学研究视野的转化,即从对癌细胞本身特性的关注提升到对整个肿瘤“生态系统”的研究,最后讨论了肿瘤基因组学未来在推动基础肿瘤生物学理解与临床转化应用方面潜在的发展方向。

该工作首先总结了肿瘤基因组学在理解癌细胞致癌机制、分子特征以及癌症的分型与精准治疗方面的里程碑式发现。2010年,张泽民教授团队在国际上首次完成人类实体瘤的全基因组测序,使得在全基因组水平上研究不同类型的致癌突变成为可能【1】。根据致癌突变对患者进行分型推动了精准、靶向治疗的发展,例如具有EGFR突变的非小细胞肺癌患者、BRAF突变的黑素瘤患者可以对相应靶向药产生临床响应。国际合作计划TCGA、ICGC通过整合大规模、多组学测量的基因组数据,在泛癌种水平上揭示了主要的致癌规律与癌症的分子分型。基于前期的发展,文章总结了肿瘤基因组学的发展趋势,强调了样本规模、测量维度与检测分辨率对理解癌症分子机制的重要意义。
肿瘤异质性是阻碍有效肿瘤治疗的关键因素,理解肿瘤内部的分子异质性是肿瘤基因组学的核心问题。传统bulk样本能够揭示不同患者间致癌突变的差异,却难以精确定义样本内部的异质性。2012年,对同一患者肿瘤的多区域采样与分析揭示了肿瘤内部致癌突变分布的空间异质性。而单细胞全基因组测序技术,例如LIANTI【2】等技术的发展为肿瘤异质性研究带来了新的机遇。自此,我们得以从单细胞水平上追踪癌细胞的演化历程与致癌基因的异质性,这为理解并攻克肿瘤患者对不同治疗方式的抗药性提供了新的可能性。
2011年,肿瘤免疫检查点抑制疗法正式被FDA批准并在临床取得巨大成功。在理解癌细胞异质性的基础上,研究整个肿瘤生态系统尤其是免疫微环境的特征与异质性成为肿瘤基因组学新的发展方向。在2009年,单细胞水平上全转录组测序【3】的实现则为全面刻画肿瘤微环境的功能组成提供了技术支撑。自张泽民教授回国工作以来,课题组在肿瘤免疫微环境研究方面取得了国际上领先的成果,先后在肝癌、肺癌、结直肠癌以及泛癌种水平上研究了肿瘤浸润T细胞、髓系细胞等的功能状态、组分、动态演化机制、相互作用关系以及异质性(图1)【4-7】,鉴定了多种肿瘤特异的免疫细胞类型,例如CXCL13+ T细胞、SPP1+ 肿瘤相关巨噬细胞、LAMP3+ 树突状细胞等,并阐述了它们与患者预后以及肿瘤免疫治疗响应的关联,为全面理解肿瘤生态系统的异质性奠定了基础。
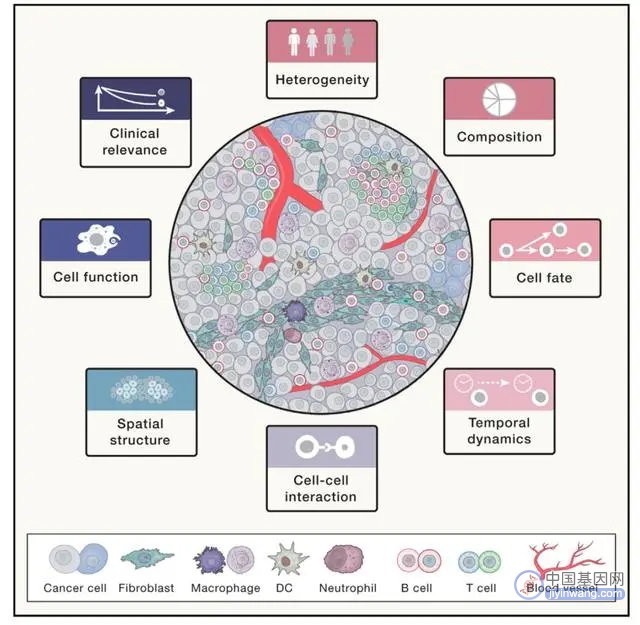
图1 刻画肿瘤微环境的特征
当前,肿瘤基因组学仍处在迅猛发展之中。随着单细胞测序样本的累积,大规模整合患者数据并对其肿瘤微环境分型成为可能,有望帮助揭示新的精准免疫治疗方案。同时,空间组学、单细胞多组学、以及结合CRISPR功能干扰的单细胞测序技术在逐渐普及,这些将进一步丰富我们对肿瘤微环境组成与功能机制的理解。未来几年内,肿瘤基因组学有望在基础肿瘤生物学与临床转化应用的多个方面产生重要的突破性成果。例如,在HCA、HTAN等国际合作项目的支持下,我们将逐渐掌握人类肿瘤的完整组成成分以及各个成分的相互作用关系,全面理解肿瘤发展的时空动态变化规律;与此同时,我们也将能够系统性阐述癌症发生的早期因果机制,准确定位肿瘤恶性转化的时间点,并指出不同肿瘤微环境类型产生的动态过程与影响因素,为肿瘤的精准治疗提供依据;进一步,结合液体活检技术对肿瘤微环境状态的无创探测,基因组学方法将助力癌症的早期发现与治疗。
在肿瘤治疗方面,随着研究的视野提升至整个肿瘤生态系统,文章突出强调了一种新的治疗方案开发思路。传统疗法关注肿瘤内部信号通路,通过干扰“靶基因”直接杀死癌细胞,在此基础上,文章提出肿瘤治疗的“靶细胞”理论(图2),通过识别并干预“靶细胞”,扰动肿瘤微环境中的细胞相互作用网络,阻断至今仍未知或未被有效干预的免疫逃逸机制,增强免疫系统杀伤癌细胞的能力,从而有效控制甚至消灭肿瘤。靶细胞的研究思路有望拓展当前的免疫检查点抑制疗法,为肿瘤治疗提供新的前景。
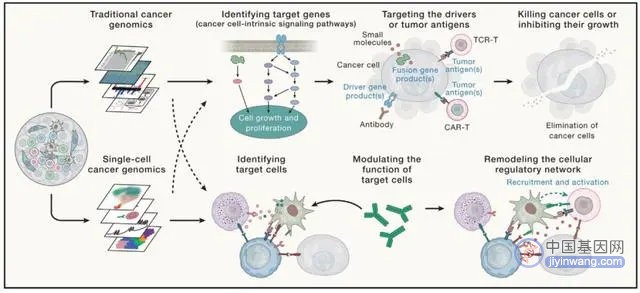
图2 基于“靶细胞”寻找新的肿瘤治疗方案
北京大学生物医学前沿中心助理研究员王东方、博士生刘宝琳为文章的并列第一作者,张泽民教授为通讯作者。
原文链接:
https://www.cell.com/cell/fulltext/S0092-8674(23)00143-5
参考文献
1. Lee, W., Jiang, Z., Liu, J., Haverty, P.M., Guan, Y., Stinson, J., Yue, P., Zhang, Y., Pant, K.P., Bhatt, D., et al. (2010). The mutation spectrum revealed by paired genome sequences from a lung cancer patient. Nature 465, 473–477. 10.1038/nature09004.
2. Chen, C., Xing, D., Tan, L., Li, H., Zhou, G., Huang, L., and Xie, X.S. (2017). Single-cell whole-genome analyses by Linear Amplification via Transposon Insertion (LIANTI). Science 356, 189–194. 10.1126/science.aak9787.
3. Tang, F., Barbacioru, C., Wang, Y., Nordman, E., Lee, C., Xu, N., Wang, X., Bodeau, J., Tuch, B.B., Siddiqui, A., et al. (2009). mRNA-Seq whole-transcriptome analysis of a single cell. Nat. Methods 6, 377–382. 10.1038/nmeth.1315.
4. Zhang, L., Yu, X., Zheng, L., Zhang, Y., Li, Y., Fang, Q., Gao, R., Kang, B., Zhang, Q., Huang, J.Y., et al. (2018). Lineage tracking reveals dynamic relationships of T cells in colorectal cancer. Nature 564, 268–272. 10.1038/s41586-018-0694-x.
5. Zhang, Q., He, Y., Luo, N., Patel, S.J., Han, Y., Gao, R., Modak, M., Carotta, S., Haslinger, C., Kind, D., et al. (2019). Landscape and Dynamics of Single Immune Cells in Hepatocellular Carcinoma. Cell 179, 829-845.e20. 10.1016/j.cell.2019.10.003.
6. Cheng, S., Li, Z., Gao, R., Xing, B., Gao, Y., Yang, Y., Qin, S., Zhang, L., Ouyang, H., Du, P., et al. (2021). A pan-cancer single-cell transcriptional atlas of tumor infiltrating myeloid cells. Cell 184, 792-809.e23. 10.1016/j.cell.2021.01.010.
7. Zheng, L., Qin, S., Si, W., Wang, A., Xing, B., Gao, R., Ren, X., Wang, L., Wu, X., Zhang, J., et al. (2021). Pan-cancer single-cell landscape of tumor-infiltrating T cells. Science 374, abe6474. 10.1126/science.abe6474.
声明:本站所有文章资源内容,如无特殊说明或标注,均为采集网络资源。如若本站内容侵犯了原著者的合法权益,可联系本站删除。