Cell:DNA甲基化在哺乳动物中的跨代遗传
在植物和一些无脊椎动物中跨代表观遗传(Transgenerational epigenetic inheritance)非常普遍【1-2】,但是在哺乳动物中表观修饰的跨代遗传是否存在以及如何进行还仍是备受争论。
2023年2月7日,美国Salk研究所与Altos Labs的Juan Carlos Izpisua Belmonte研究组在Cell发表了文章Transgenerational inheritance of acquired epigenetic signatures at CpG islands in mice,证明了启动子相关CpG岛的DNA甲基化可以从亲代传递到子代,这对于理解表观遗传在人类疾病中的作用提供了新的思考方向。

在哺乳动物中存在两个不同的阶段防止表观遗传的跨代遗传。全基因组DNA甲基化的擦除发生在原始生殖细胞中,之后胚胎植入前DNA甲基化再次被擦除。最后随着植入后胚胎开始细胞分化,基因组的DNA甲基化开始从头建立【3】。但是研究表明原始生殖细胞中某些重复的区域会逃脱基因组表观遗传的重编程【4】。
在高度甲基化的哺乳动物基因组中CpG岛是非甲基化区域中频繁出现的甲基化CpG区域,通常与启动子区域以及基因起始有关,超过一半的基因启动子与CpG岛相关【5】。对于DNA 甲基化研究的主要壁垒在于缺少DNA甲基化编辑的动物模型,靶向CpG岛对DNA进行从头甲基化编辑。
为了实现这一目标,作者们将目光集中在两个CpG岛代谢相关的基因Ankrd26(Ankyrin repeat domain 26)以及低密度脂蛋白受体Ldlr(Low-density lipoprotein receptor)的启动子区域。这两个蛋白分别敲除后会造成肥胖症以及高胆固醇血症【6-7】。作者们建立了一个CpG-free DNA的整合可稳定诱导的CpG岛从头DNA甲基化方法(图1)。据此,作者们构建了一个目标载体进行小鼠胚胎干细胞转染。对于Ankrd26的CpG岛目标,八个同源重组克隆中有四个表现出从头甲基化,其中有两个克隆出现超过55%的CpG岛甲基化,对于Ldlr的CpG岛靶标,九个同源重组的克隆中有两个表现出甲基化,有35%的CpG岛被甲基化。作者们发现这些甲基化修饰的CpG岛是稳定存在的,由于甲基化后对Ankrd26 mRNA表达抑制,导致表达量只有野生型的10%,在细胞传代25代之后依然能够保持稳定的抑制维持。
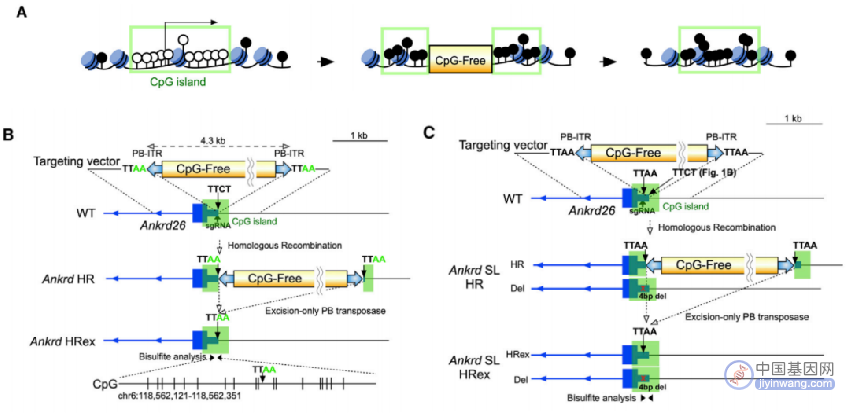
图1 CpG岛从头甲基化方法
随后,作者们将10-15个DNA甲基化修饰的小鼠胚胎干细胞移植到8细胞期的小鼠胚胎中,成功构建了嵌合体小鼠。作者们发现嵌合体小鼠的体细胞CpG岛区域的甲基化能够高度保持,与此相一致的是这些小鼠会表现出与Ankrd26基因敲除小鼠相似的表型, 体形肥胖、嗜食、血清瘦素水平升高等。另外,Ldlr胚胎干细胞移植后也能够维持CpG岛区域的甲基化。因此,作者们的方法能够做到生成DNA甲基化编辑的小鼠,并表现出对目标CpG岛稳定高甲基化且沉默下游的基因。
进一步地,作者们希望探究Ankrd26基因CpG岛区域的甲基化是否能够跨代遗传。作者们所有的F1代小鼠都能够继承甲基化,比例约为95%。而且CpG岛的甲基化的能够分别被稳定传递到父本和母本的生殖细胞系中,F3以及F4代仍然能够稳定维持CpG岛的甲基化修饰。Ldlr的CpG岛的甲基化也能够稳定遗传到至少F6代。重要的是,Ankrd26与Ldlr的CpG岛甲基化所引发的表型也在子代中稳定继承。
那么CpG岛中的DNA甲基化是如何传递到子代中的呢?通过检查生殖细胞系中的DNA甲基化状态,作者们发现其中DNA甲基化被擦除,直到E13.5天才恢复,后代中DNA甲基化是在受精后被重新建立起来的。
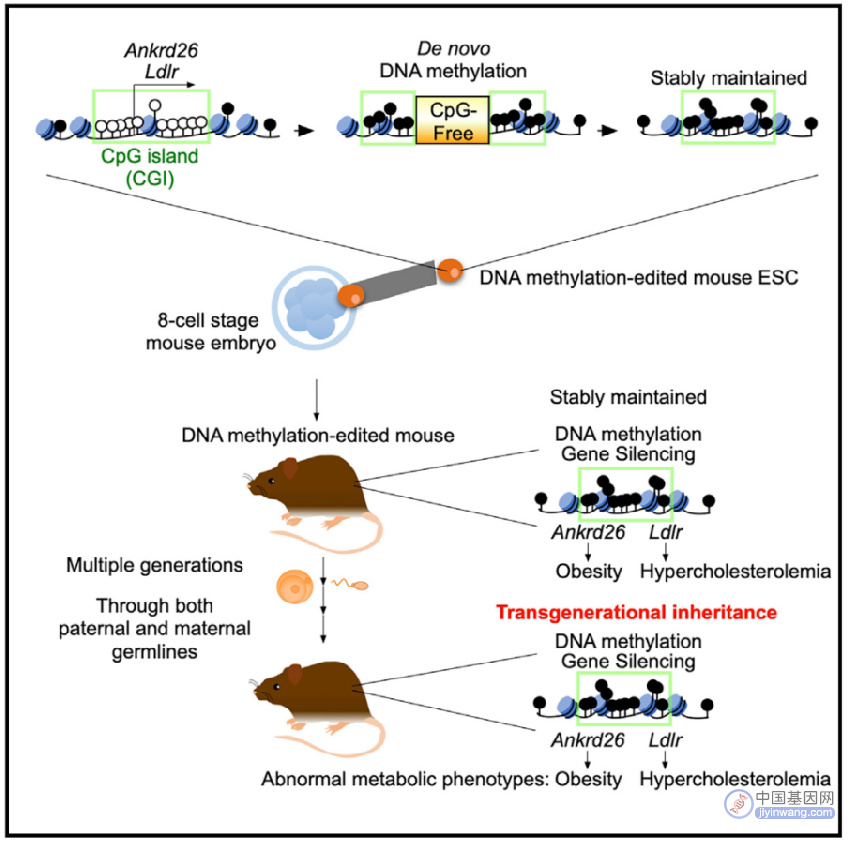
图2 工作模型
总的来说,作者们的工作建立了对DNA甲基化状态进行编辑小鼠胚胎干细胞模型以及小鼠模型,证明了小鼠中CpG岛的甲基化能够跨代遗传,这些结果为哺乳动物的表观修饰的遗传记忆提供了新的工具,也为理解非遗传疾病提供了新的理解。
原文链接:https://doi.org/10.1016/j.cell.2022.12.047
参考文献
1. Boskovic, A., and Rando, O.J. (2018). Transgenerational epigenetic inheritance. Annu. Rev. Genet. 52, 21–41. https://doi.org/10.1146/annurevgenet-120417-031404.
2. Heard, E., and Martienssen, R.A. (2014). Transgenerational epigenetic inheritance: myths and mechanisms. Cell 157, 95–109. https://doi.org/10.1016/j.cell.2014.02.045
3. Smith, Z.D., Chan, M.M., Mikkelsen, T.S., Gu, H., Gnirke, A., Regev, A., and Meissner, A. (2012). A unique regulatory phase of DNA methylation in the early mammalian embryo. Nature 484, 339–344. https://doi.org/10.1038/nature10960.
4. Hackett, J.A., SenGupta, R., Zylicz, J.J., Murakami, K., Lee, C., Down, T.A., and Surani, M.A. (2013). Germline DNA demethylation dynamics and imprint erasure through 5-hydroxymethylcytosine. Science 339, 448–452. https://doi.org/10.1126/science.1229277.
5. Saxonov, S., Berg, P., and Brutlag, D.L. (2006). A genome-wide analysis of CpG dinucleotides in the human genome distinguishes two distinct classes of promoters. Proc. Natl. Acad. Sci. USA 103, 1412–1417. https://doi.org/10.1073/pnas.0510310103.
6. Bera, T.K., Liu, X.-F., Yamada, M., Gavrilova, O., Mezey, E., Tessarollo, L., Anver, M., Hahn, Y., Lee, B., and Pastan, I. (2008). A model for obesity and gigantism due to disruption of the Ankrd26 gene. Proc. Natl. Acad. Sci. USA 105, 270–275. https://doi.org/10.1073/pnas.0710978105.
7. Ishibashi, S., Brown, M.S., Goldstein, J.L., Gerard, R.D., Hammer, R.E., and Herz, J. (1993). Hypercholesterolemia in low density lipoprotein receptor knockout mice and its reversal by adenovirus-mediated gene delivery. J. Clin. Invest. 92, 883–893. https://doi.org/10.1172/JCI116663.
声明:本站所有文章资源内容,如无特殊说明或标注,均为采集网络资源。如若本站内容侵犯了原著者的合法权益,可联系本站删除。