Nature:微生物世界的秘密基因——揭示未培养类群的功能与进化
文章概述了一项关于微生物遗传物质的研究,着重于那些未被培养和研究过的微生物。通过分析149,842个环境基因组,研究人员创建了一个包含404,085个新的、对特定未培养原核生物类别重要的基因家族(FESNov)的目录。
该研究几乎将已知细菌和古菌基因家族的数量增加了三倍。这些基因家族跨越多个物种,并展现出强烈的纯化选择信号,这表明它们是新的同源群。此外,FESNov目录还发现了1034个新的基因家族,这些家族可能代表了促进这些未培养类群进化分化的共同特征。
PART/1
研 究 背 景
过去几十年里,宏基因组学和元条形码研究颠覆了微生物基因组学,揭示了许多新的细菌和古菌谱系及其独特的基因组成。许多环境基因在已培养的生物中没有同源基因,这些所谓的“未知”基因占据了约25%至50%的比例,它们的功能性和生态学及进化学重要性尚未被充分了解。
PART/2
研 究 内 容
研究者利用基因组上下文重建和结构比对来探索FESNov家族的潜在功能角色。通过这些分析,他们为32.4%的FESNov家族预测了功能关联,其中4349个与重要的生物学过程高度相关。
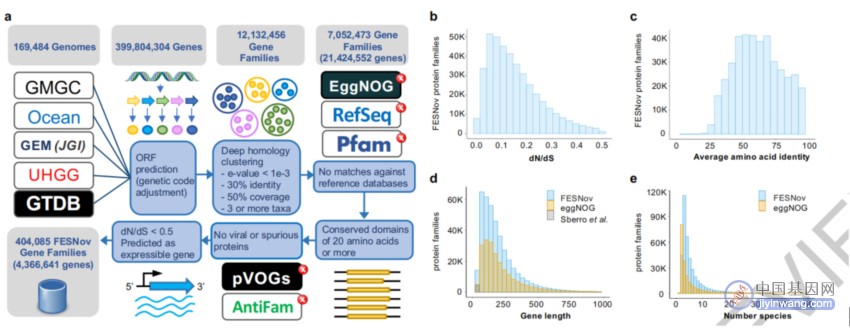
图1 基因家族发现流程和一般统计
研究者通过一系列步骤确定了功能和进化上重要的新基因家族(FESNov)。如图1所示,包括:(a) 用于识别FESNov基因家族的工作流程;(b) FESNov基因家族中非同义和同义替换(dN/dS)比率的分布;(c) FESNov基因家族平均蛋白质氨基酸特征的分布;(d) FESNov基因家族和所有细菌及古菌eggNOG同源群的蛋白长度分布对比;(e) FESNov目录与细菌和古菌eggNOG同源群每个家族的物种数量分布对比。这表明了研究者成功地确定了一系列新的、重要的基因家族。
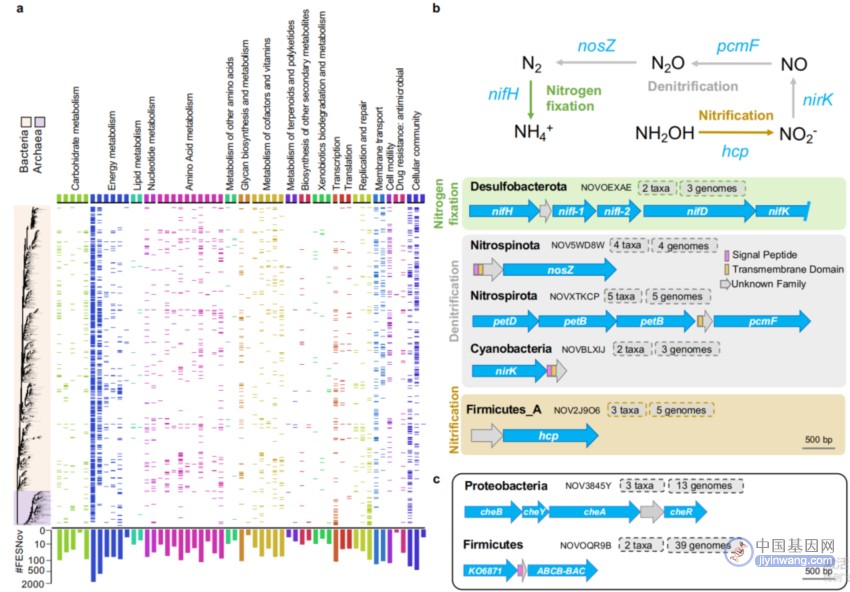
图2 与KEGG途径紧密联系的FESNov基因家族分布
研究者调查了FESNov基因家族与KEGG途径的关联。如图2所示,(a) 展示了与KEGG途径(列)相关的FESNov家族的存在/不存在矩阵,跨越细菌和古菌GTDB谱系,并在目级别上进行了分类;(b) 展示了与氮循环相关基因紧密耦合的FESNov基因家族示例;(c) 展示了实验验证的FESNov基因家族NOV3845Y(细胞运动性)和NOVOQR9B(抗菌肽)的基因组背景。因此,研究者能够确定和验证这些基因家族在关键生物途径中的作用。
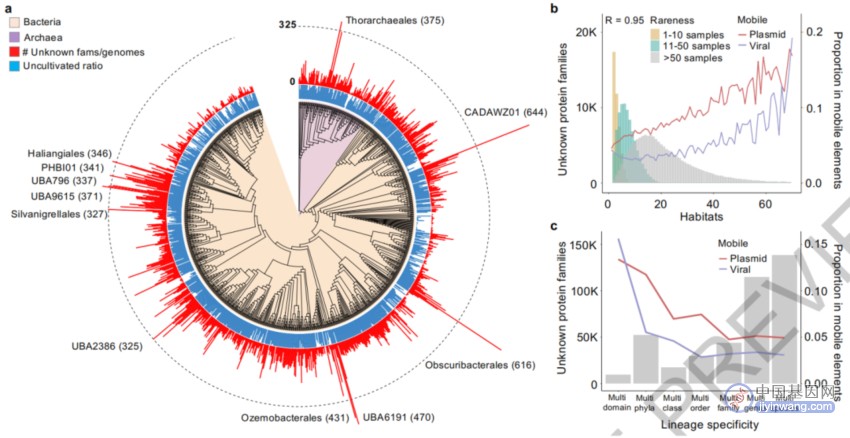
图3 FESNov基因家族遍及整个微生物谱系,覆盖多种栖息地
研究者分析了FESNov基因家族在不同栖息地和微生物谱系中的分布。如图3所示,(a) 描述了FESNov基因家族在GTDB5细菌和古菌谱系中的分布,展示了在每个目分类学上的基因组中未知基因家族的数量;(b) 描述了FESNov基因家族的生态宽度(以栖息地数量衡量)及其在样本中的检测稀有度;(c) 展示了FESNov基因家族在不同分类级别上的分布。这表明FESNov基因家族在微生物多样性中的广泛分布。
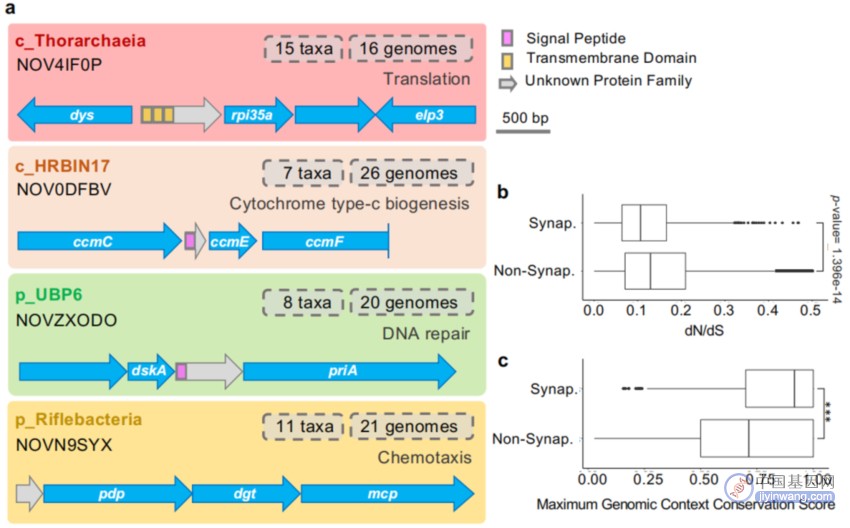
图4 在高级别分类排名中发现的FESNov同源基因家族
研究者探讨了在高级别分类排名中的FESNov同源基因家族的重要性。如图4所示,(a) 提供了四个门和纲级别同源FESNov基因家族的示意概览,这些家族位于参与关键细胞过程的保守基因簇中;(b) 比较了同源(n=1034)和非同源(n=403051)FESNov基因家族的dN/dS值;(c) 比较了同源(n=1034)和非同源(n=402987)FESNov基因家族的基因组上下文保守性。这些数据表明这些基因家族在进化上极为重要。
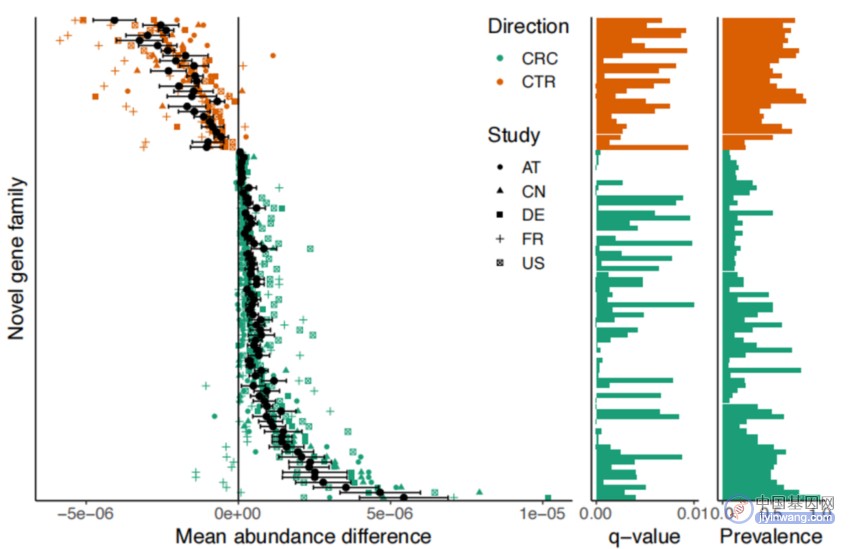
图5 FESNov基因家族对CRC预测力的贡献,包括疾病的生物标志物
研究者评估了FESNov基因家族在识别结直肠癌(CRC)方面的潜在作用。如图5所示,展示了在575个人类结直肠癌样本(来自五个不同的研究群体)中显著高表达(用绿色表示)和低表达(用橙色表示)的FESNov基因家族的相对丰度差异。这些结果强调了新基因家族在发现新的功能性生物标志物以及它们可能与观察到的差异相关的分子机制中的高效用。
PART/3
总 结 与 展 望
本研究的目标是识别和特征化未被描述的微生物基因家族,这些家族具有高度的进化和功能重要性。然而,FESNov目录仅占原始数据集中所有未知序列的一小部分,因此仍有许多未被发现的基因。研究还表明,FESNov目录可以作为假设驱动的框架,用于鉴定和实验表征未知但功能重要的基因。最后,研究强调了将这些独特的遗传资源定期纳入参考数据库和生物信息学工作流的重要性。
额外信息:新生物标志物的发现
研究还探讨了将FESNov目录纳入生物标志物发现管道的潜在好处。例如,在研究结直肠癌(CRC)检测中,通过分析575个人类肠道样本,研究人员识别出69个在CRC样本中显著过量的新基因家族,这些家族可能与肠道中CRC患者丰富的7个不同物种相关。
参考文献
[1] Rodríguez del Río, Á., Giner-Lamia, J., Cantalapiedra, C.P. et al. Functional and evolutionary significance of unknown genes from uncultivated taxa. Nature (2023).
声明:本站所有文章资源内容,如无特殊说明或标注,均为采集网络资源。如若本站内容侵犯了原著者的合法权益,可联系本站删除。