基因转录如何“踩刹车”?上海科学家揭示基因转录终止机制
北京时间2023年1月12日,中国科学院分子植物科学卓越创新中心合成生物学重点实验室张余研究团队和美国威斯康辛大学麦迪逊分校Robert Landick团队以及浙江大学冯钰团队合作在《自然》杂志上发表题为“Structural basis for intrinsic transcription termination(转录终止的结构机制)”的研究论文,该研究揭示了细菌RNA聚合酶如何识别转录终止序列终止转录的工作机制。

DNA是遗传信息的载体,遗传信息通过转录从DNA传递到RNA,随后通过翻译解码成蛋白质。
基因是DNA遗传信息的编码单元,基因的正确解码需要执行基因转录的RNA聚合酶严谨识别基因的的起始序列(启动子)和终止序列(终止子)。
RNA聚合酶在执行基因转录时类似高速行驶的汽车,以大约每秒50个核苷酸的速度合成RNA,当RNA聚合酶转录至终止序列时,需要从高速延伸的状态刹车、停止转录并释放RNA。
转录终止过程发生异常会干扰下游基因的表达,影响DNA复制,破坏基因组稳定性等。
细菌的固有转录终止序列是一段大约30-50个核苷酸碱基的序列,由能够形成发卡结构的序列和连续的Uridine(Polyuridine;多聚尿苷)组成。
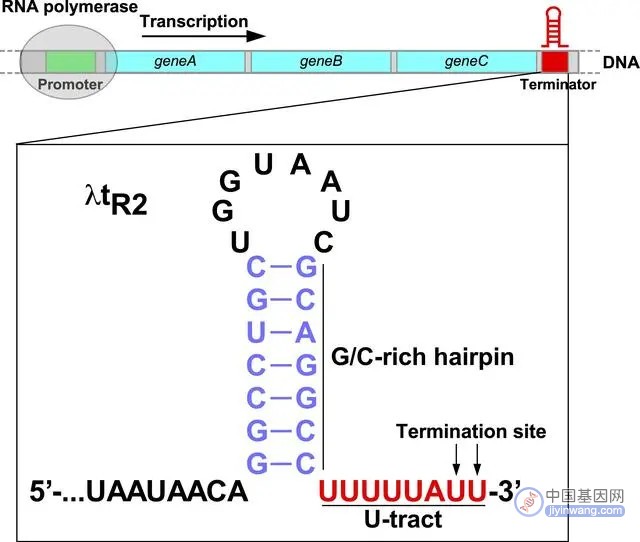
▲细菌转录终止序列示意图
研究团队捕获了RNA聚合酶转录终止的一系列中间状态,解析了RNA聚合酶在上述转录终止中间状态的冷冻电镜三维结构。
研究发现,转录终止序列的多聚尿苷使RNA聚合酶刹车,将其固定在转录暂停状态,随后RNA发卡结构折叠进入RNA聚合酶内部,促使RNA从RNA聚合酶内部解离。
该研究报导了细菌RNA聚合酶在基因转录终止序列暂停转录,RNA发夹结构折叠进RNAP内部诱导RNA释放的分子机制。该研究回答了基因表达的基础科学问题,拓展了人们对于基因表达的机制理解。
中科院分子植物科学卓越创新中心博士生尤琳琳(已毕业)为论文的第一作者,张余研究员和威斯康辛大学麦迪逊分校Robert Landick教授以及浙江大学冯钰研究员为共同通讯作者。

▲张余研究员(左)与尤琳琳博士
声明:本站所有文章资源内容,如无特殊说明或标注,均为采集网络资源。如若本站内容侵犯了原著者的合法权益,可联系本站删除。