Nature:埃博拉病毒RNA聚合酶如何玩转基因组RNA复制
Journal Club 凝聚了一批热爱科学、享受科学、传播科学的探索者和发现者。他们志同道合,他们青春飞扬,他们与科学共舞。在这里,你可以收获最新、最真的行业资讯和来自科研一线的文献解读。你关心的就是我们要说的,我们将用最质朴、最客观的文字书写有深度、有温度的科学。Journal Club,与你畅游Journal世界,和你共享科学蔚蓝。
负链RNA病毒(negative-strand RNA virus, NSV)包括在人和动物中广泛流行的一系列病原体,如埃博拉病毒(EBOV)、拉沙病毒(LASV)、尼帕病毒、狂犬病毒(RABV)、流感病毒和人类呼吸道合胞病毒(HRSV)1。作为全球经济和公共卫生的重大威胁,理解NSV复制的分子机制,以及开发针对NSV感染的新型抗病毒药物,对抵抗其感染至关重要。
根据病毒基因组的组成,NSV可以进一步分为非分段NSV(non-segmented NSV, nsNSV)和分段NSV (segmented NSV, sNSV),前者如EBOV,后者如流感病毒。nsNSV和sNSV的单链RNA基因组与病毒核蛋白组装成具有螺旋结构的病毒核糖核蛋白(viral ribonucleoprotein, vRNP)复合物2。在sNSV中,每个基因组RNA片段可以通过部分互补的5'和3'末端形成一个伪环,这些末端在同一组病毒中高度保守。而对于nsNSV,vRNA的5'和3'末端通常分别位于两个相反的末端2。
所有NSV都可以编码一个包含RNA依赖的RNA聚合酶(RNA-dependent RNA polymerase, RdRP)结构域的大型RNA聚合酶,并在vRNP中对病毒RNA基因组进行复制或转录2。nsNSV的聚合酶在基因组的3'-末端启动转录,并顺序将前导区和内部基因转录到前导RNA和具有5'-帽子和3'-poly(A)尾的单顺式mRNA中2。据推测,nsNSV的转录遵循一种起始-终止机制(start-stop mechanism):通过识别每个病毒基因两侧的基因起始(gene start, GS)和基因终止(gene end, GE)信号。在相应的GE信号处,当转录本的转录终止时,聚合酶应该要么从模板中解离,要么在其GS信号处继续合成下游转录本。对于基因组复制来说,聚合酶也从基因组的3'-末端开始,产生一个完整长度的互补基因组RNA,基因组中所有的GS和GE信号都会被聚合酶忽略。然而,聚合酶的功能在转录和复制之间切换的分子机制目前仍然不清楚,目前认为这种切换可能受到P蛋白的磷酸化状态或其他转录激活因子的调控3。
作为nsNSVs的代表,EBOV对人类具有高度的致病性,可以引起严重的临床症状,包括出血热和多器官功能衰竭。2023年9月13日,中科院的施一教授和高福教授共同通讯,在Nature上以加速预览的形式发表了题为Molecular mechanism of de novo replication by the Ebola virus polymerase的科研论文,报道了第一个L蛋白识别基因组启动元件机制的结构。在EBOV中, L-VP35复合物是发挥高效RNA聚合活性的基本功能单元,结合冷冻电镜,研究者解析了EBOV L-VP35复合物的整体结构,同时清楚地揭示了与辅因子VP35四聚体紧密相互作用的L蛋白活性位点的结构特征。
本研究共表达的EBOV L蛋白和VP35能够形成可溶且均一的L-VP35,在功能实验中,在RNA引物缺失的情况下,L-VP35能够以EBOV基因组RNA 3’端前导序列的18-mer RNA生成RNA产物,提示该复合物具有从头复制的活性。此外,研究者发现,当反应体系中只存在单一GTP底物时,L-VP35能够利用其3’端延伸活性对18-mer 前导RNA模板的3’端进行修饰,而当4种NTP底物同时存在时,这种活性却会减弱,鉴于类似的现象也在其他nsNSV中被观察到,提示这可能是nsNSV聚合酶共有的机制。此外,研究者发现,L-VP35的从头复制活性存在前导序列特异性,当前导序列被其他序列或其他nsNSV的前导序列取代时,其从头复制都无法正常进行。在以非前导序列作为模板时,3-mer或4-mer 才能驱动产物的生产,2-mer引物则无法发挥相同的作用,进一步实验提示,非特定RNA序列依赖性的复制过程是由RNA引物延伸所产生的。
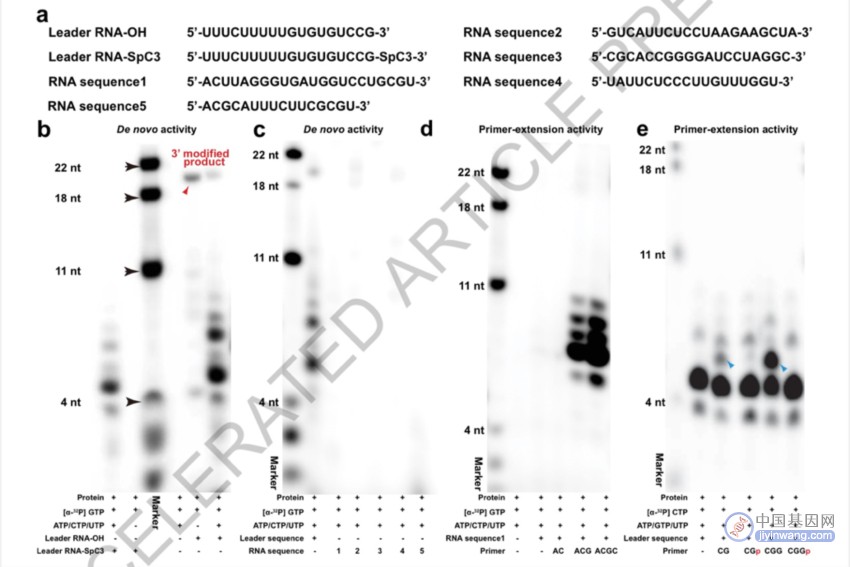
图1. EBOV L-VP35具有从头转录活性
借助冷冻电镜,本文研究者进一步解析了L-VP35与 18-mer前导RNA形成的复合物结构,该结构整体分辨率达3.0 Å 。在本文结构中,L蛋白由N端结构域 (N-terminal domain, NTD)、RdRp结构域(含有典型的fingers-palm-thumb结构)以及GDP多核糖核苷转移酶 (GDP polyribonucleotidyltransferase, PRNTase)结构域组成,3’ RNA位于由fingers、NTD和PRNTase结构域所形成的模板进入通道(template entry channel)内。在复合物中,VP35四聚体以两种构象存在,与无底物结合的L-VP35相比,VP35四聚体的参与寡聚化的部分存在摆动现象,然而在目前的结构中,这种现象出现的原因尚且无法被阐明。
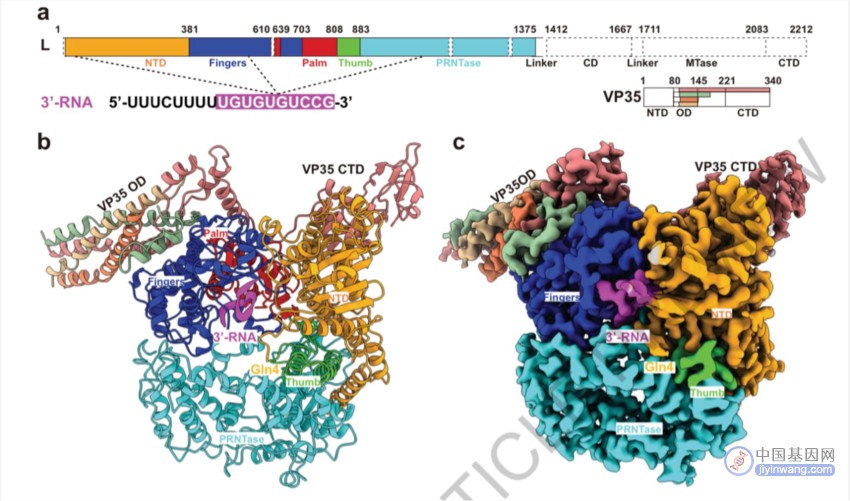
图2. EBOV L-VP35-RNA复合物的整体结构
3’前导RNA在L蛋白的模板进入通道中以一种弯折的构象结合,并形成许多分子内相互作用以稳定这种构象。3’端G1、C2和C3互相堆叠在一起,U4、G5和U6堆叠在一起,两部分的连接处存在弯折。G7翻转到相反的方向,其位置由3对氢键稳定,U8与G7位于同一方向上,而G9又翻转到相反的方向。作为结构中最后一个可以被看见的核苷酸,U10进一步弯折,以适应RNA链在整个通道中所需要采取的构象。其中,G7是整个RNA链中构象特征最为显著的部分,且是唯一与U4形成相互作用的核苷酸。当G7被突变为A、U或C时,聚合酶会失去从头复制活性。
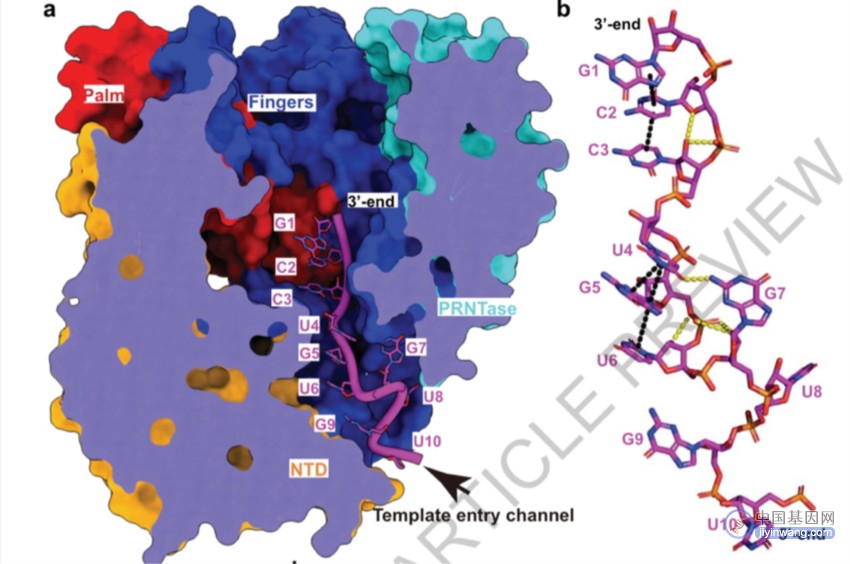
图3. 3’前导RNA的弯折构象
通过比对RNA结合的L-VP35结构与无底物的L-VP35结构,研究者发现除了NTD的尖端由于与G9结合发生了构象变化外,L蛋白的绝大部分在两个结构中可以被很好地吻合在一起。蛋白与RNA之间的相互作用主要通过氢键和π-π相互作用维持。
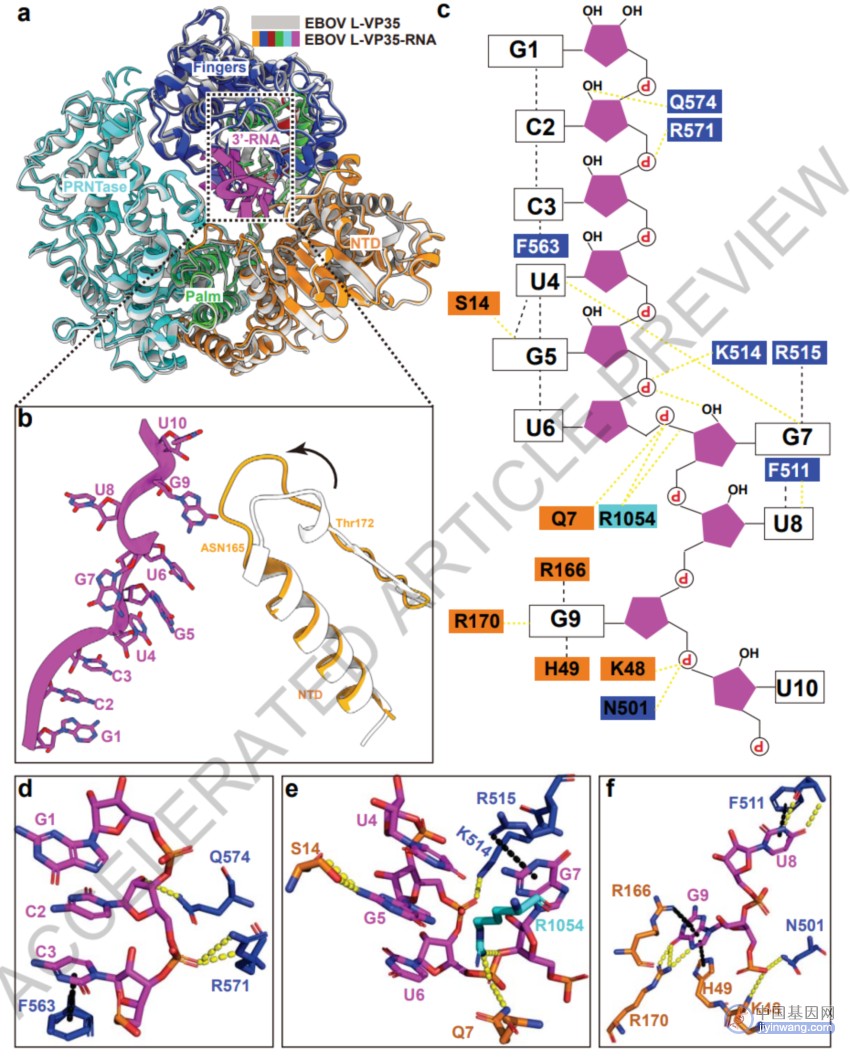
图4. EBOV聚合酶与前导RNA之间的相互作用
为进一步探究关键残基(NTD上的H49、R166、R170以及fingers️ domain上的R515)对L蛋白从头复制活性的影响,研究者将这些残基突变为丙氨酸,结果显示,单个/两个氨基酸残基的突变并不影响L-VP35与RNA链之间的高亲和力结合,然而,当4个氨基酸残基同时突变时,聚合酶对RNA链的亲和力下降了1,000倍之多。进一步功能分析提示,H49A突变不影响聚合酶从头复制活性,而R166A或R170A则会导致聚合酶活性下降,R515A会导致抑制复制活性,而4个突变同时存在时,复制活性完全消失。结合该酶在引物延伸方面的活性分析,研究者认为,这种特定的RNA构象对酶的从头复制活性有更大的影响。
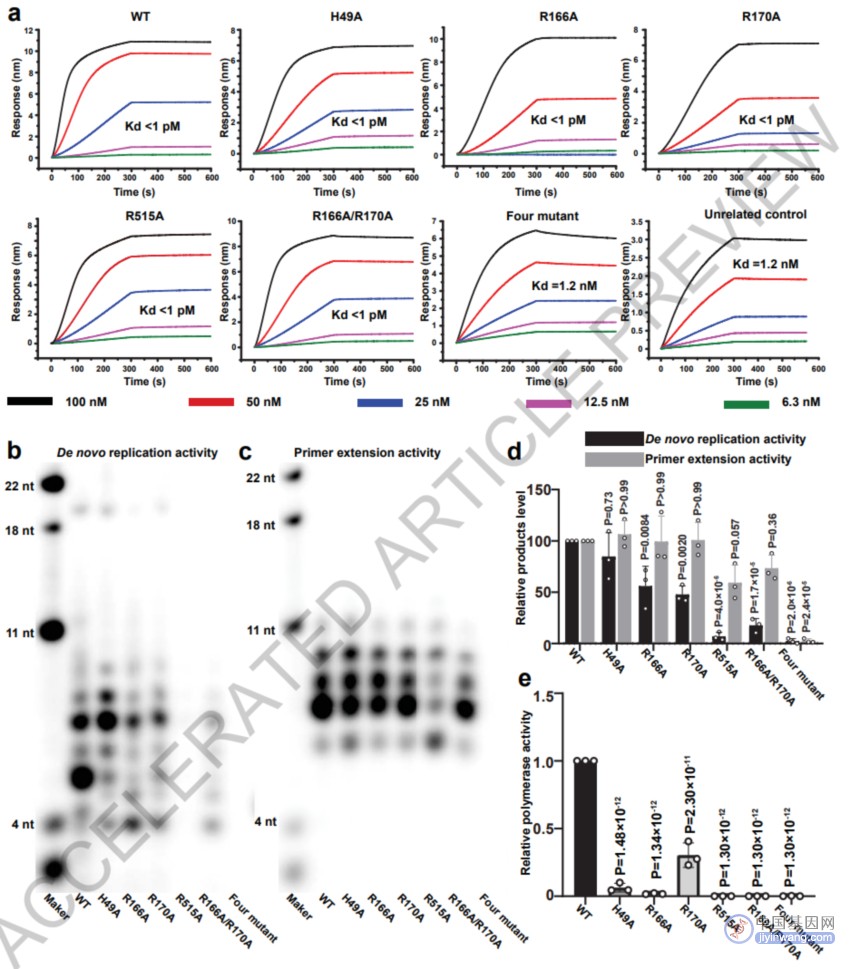
图5. L蛋白突变对RNA结合以及合成的影响
综上,本文的结构揭示了EBOV RNA聚合酶识别基因组RNA以及复制的独特机制,对理解nsNSV RNA聚合酶作用的发挥提供了更多的细节信息。未来针对EBOV等nsNSV复制机制的研究,或许能给新型抗病毒药物的研发带来更多新思路。
原文链接
https://doi.org/10.1038/s41586-023-06608-1
参考文献
1. Ouizougun-Oubari, M. & Fearns, R. Structures and Mechanisms of Nonsegmented, Negative-Strand RNA Virus Polymerases. Annual Review of Virology (2023).
2. te Velthuis, A. J. W., Grimes, J. M. & Fodor, E. Structural insights into RNA polymerases of negative-sense RNA viruses (Jan, 10.1038/s41579-020-00501-8, 2021). Nature Reviews Microbiology 19, 220-220 (2021).
3. Liang, B. Structures of the Mononegavirales Polymerases. J Virol 94 (2020).
声明:本站所有文章资源内容,如无特殊说明或标注,均为采集网络资源。如若本站内容侵犯了原著者的合法权益,可联系本站删除。