Mol Cell:DNA双链断裂引起基因组插入发生的分子机制
基因组插入(Genomic insertions)是一种基因组重排事件,指的是一个或者多个供体序列插入到同一个染色体的非相邻基因座或者非同源的染色体中。这些基因组重排事件可能代表了高达2%的种系拷贝数量变异(copy-number variations, CNVs)事件,且常常与发育异常相关【1,2】。插入(Insertions)和删除(deletions)是肿瘤基因组的一个常见特征【3-5】。基因组插入一般来自错误修复的DNA损伤,比如DNA双链断裂(double-strand breaks, DSBs)。然而,这种事件发生的具体分子机制还未阐释清楚。

2023年7月3日,来自美国哥伦比亚大学的Jean Gautier团队在Molecular Cell杂志上发表了文章Mechanisms of insertions at a DNA double-strand break 。他们建立了一种对基因组插入和删除等DNA重排事件进行测序的新方法Indel-seq(insertion and deletion sequencing),并以此方法对基因组中DNA插入和删除等分子重排事件发生的分子机制进行了详细的检测,找到了这类重排事件发生的关键调控因素。

基因组中DSBs事件的发生是基因组插入和删除发生的关键因素,因此,研究人员首先建立了DSBs诱导发生的细胞模型。即在U2OS细胞系中将AsiSI核酸内切酶融合表达与雌激素受体结合结构域,通过外源4OHT(4-hydroxy-tamoxifen)诱导AsiSI核酸内切酶表达,并在基因组中产生DSBs,构建成功的细胞系命名为DIvA细胞系。检测发现4OHT在DIvA细胞系中诱导产生了超过100个DSBs,并在TRIM37基因座发生了分子重排。TRIM37基因座是基因组中第二个最容易发生DNA断裂的位点。
接下来,研究人员开发了一种高通量的靶向测序方法,并结合了一种强大的计算方法,命名为Indel-seq。通过与sanger测序方法进行比对,发现Indel-seq能够准确检测几乎所有的插入事件,检测结果表明,基因组平均插入长度为120 bp。需要注意的是,39%的插入事件发生在重复序列区域中,包括端粒、着丝粒、逆转录元件等。Indel-seq检测到增加的插入事件,28%来自端粒区域,这也说明了U2OS细胞端粒的不稳定性。
为了探索插入序列起源和含有AsiSI活性位点基因座之间的联系,研究人员将供体序列和AsiSI活性位点之间的距离进行了划分:主要分为3个,即(1)距离AsiSI位点非常近的区域(0-2 kb);(2)距离AsiSI相对近的区域(100 kb范围内);以及(3)距离AsiSI较远的区域(大于100 kb)。统计发现,供体序列来源在这三个区域内分别占据1/3的总数量,另外,来源于(1)和(2)区域的供体序列负责在AsiSI诱导产生的DSBs处进行插入事件,而远端的供体序列主要负责在基因组中自然存在的DNA损伤处进行的插入事件。
接下来,研究人员利用DNA-PK抑制剂NU7441限制NHEJ的发生,发现插入事件发生减少,删除事件增加,尤其是近端(1)和(2)处插入事件减少,而远端(3)处插入事件并未受到太大影响,说明NHEJ介导的末端连接对于插入事件的发生是必要的。而通过POLQ抑制剂抑制微同源末端连接(microhomology-mediated end joining, MMEJ)对插入事件的发生没有影响,说明插入事件的发生不经过MMEJ过程。
DSBs能够诱导损伤基因座的聚集,这个过程由WASP-Arp2/3-Actin通路介导。研究人员发现Arp2/3复合物形成抑制剂CK-666能够降低插入事件发生的频率,说明插入事件的发生涉及到DSBs聚集过程的发生,与损伤基因座之间的相互作用密切相关。基因组插入事件发生与基因重组过程有关,敲低RAD51能够明显抑制近端(1)和(2)处插入事件的发生。ChIP-seq实验也显示,Rad51结合区域与供体序列区域密切相关。
在退火以及插入事件发生过程中,不可避免的会产生大量的微同源序列(microhomologies, MHs),基因组插入的发生一般起源于微同源介导的链侵袭过程,并以NHEJ链接过程为终点。另外,DNA末端切除产生的ssDNA可以作为供体序列的模板,促进基因组插入事件的进程。
除此之外,研究人员还发现,70%的供体序列起源于启动子区或者基因转录区。起源于启动子区的插入事件一般在AsiSI活性位点的1 kb范围之内,而起源于基因转录区的插入位点一般在AsiSI活性位点较远的位置。这表明,基因组插入事件可能和基因转录相关。实验结果显示,RNA聚合酶抑制剂能够抑制启动子区附近DSBs的发生频率,证实了上述推测。除此之外,RNA合成过程中产生的DNA-RNA分子杂交也促进了基因组插入事件的发生频率。
最后,研究人员利用CRISPR/Cas9及其不同的突变系统探究了DSBs末端不同形态对插入事件发生的影响,发现3’和5’粘性末端能够促进DNA末端切除引起的基因组插入事件的发生,基因组插入事件的发生不受基因组位点的影响。
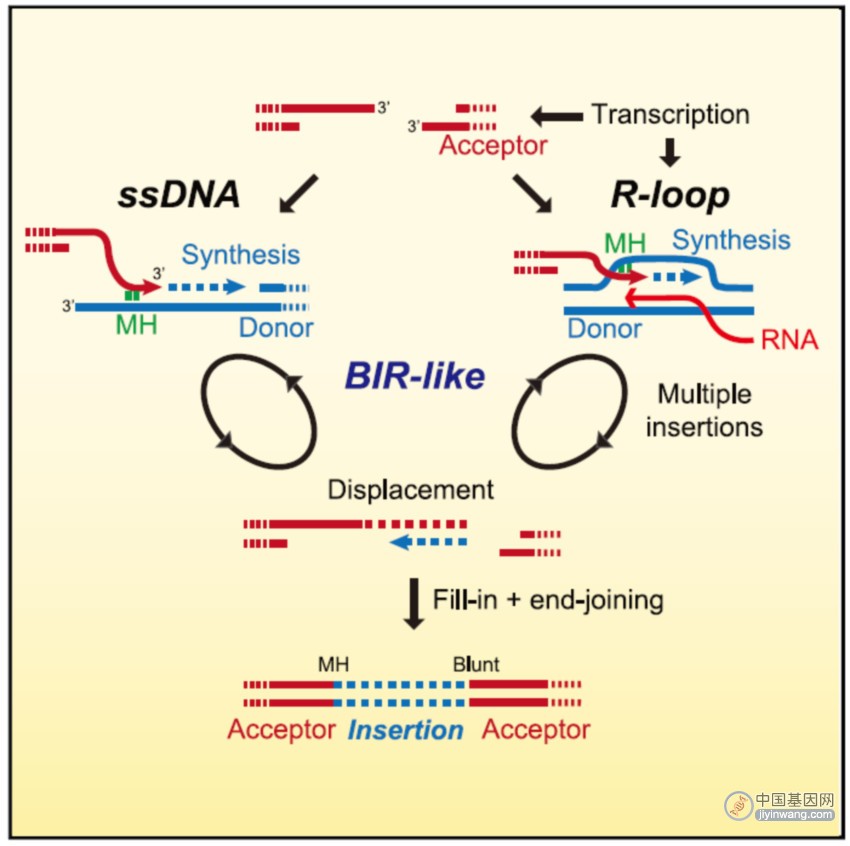
总之,本研究系统检测了DNA双链断裂引起的基因组插入事件发生的分子机制,发现这一事件的发生与NHEJ、DNA末端切除等过程密切相关,对于我们理解基因组的重排以及突变奠定了理论基础。
原文链接:
https://doi.org/10.1016/j.molcel.2023.06.016
参考文献:
1. Carvalho, C.M., and Lupski, J.R. (2016). Mechanisms underlying structural variant formation in genomic disorders. Nat. Rev. Genet. 17, 224–238.
2. Gu, S., Szafranski, P., Akdemir, Z.C., Yuan, B., Cooper, M.L., Magrin˜ a´, M.A., Bacino, C.A., Lalani, S.R., Breman, A.M., Smith, J.L., et al. (2016). Mechanisms for complex chromosomal insertions. PLoS Genet. 12, e1006446.
3. Corte´ s-Ciriano, I., Lee, J.J., Xi, R., Jain, D., Jung, Y.L., Yang, L., Gordenin, D., Klimczak, L.J., Zhang, C.Z., Pellman, D.S., et al. (2020). Comprehensive analysis of chromothripsis in 2,658 human cancers using whole-genome sequencing. Nat. Genet. 52, 331–341.
4. Li, Y., Roberts, N.D., Wala, J.A., Shapira, O., Schumacher, S.E., Kumar, K., Khurana, E., Waszak, S., Korbel, J.O., Haber, J.E., et al. (2020). Patterns of somatic structural variation in human cancer genomes. Nature 578, 112–121.
5. Alexandrov, L.B., Kim, J., Haradhvala, N.J., Huang, M.N., Tian Ng, A.W., Wu, Y., Boot, A., Covington, K.R., Gordenin, D.A., Bergstrom, E.N., et al. (2020). The repertoire of mutational signatures in human cancer. Nature 578, 94–101.
声明:本站所有文章资源内容,如无特殊说明或标注,均为采集网络资源。如若本站内容侵犯了原著者的合法权益,可联系本站删除。