绘制肝内胆管癌致癌驱动基因突变图谱
编者按:肝内胆管癌(iCCA)是第二高发的原发性肝脏恶性肿瘤,肿瘤异质性高,5年生存率低于10%。当前大部分iCCA患者发现时即为晚期,手术切除率低,同时尚缺乏有效的靶向、免疫治疗方案,因此iCCA的临床治疗面临着巨大挑战。大量研究表明,iCCA具有高度异质性的基因组突变和肿瘤微环境,这可能与其高侵袭性和不良预后相关。人们通过绘制更精确的肿瘤分子学图谱,为系统理解肿瘤异质性及实现个体化治疗提供理论依据。
我国复旦大学附属中山医院樊嘉院士、中国科学院上海药物研究所周虎研究员、中国科学院分子细胞科学卓越创新中心高大明研究员、复旦大学附属中山医院高强教授课题组合作,于2022年初在Cancer Cell上发表过一项研究,基于来自262例iCCA患者癌组织的基因组、转录组、蛋白质组和磷酸化蛋白质组等多组学数据,绘制了中国iCCA患者的多维分子图谱,为iCCA的发生发展机制、分子分型、预后监测和个性化治疗策略提供了新思路。
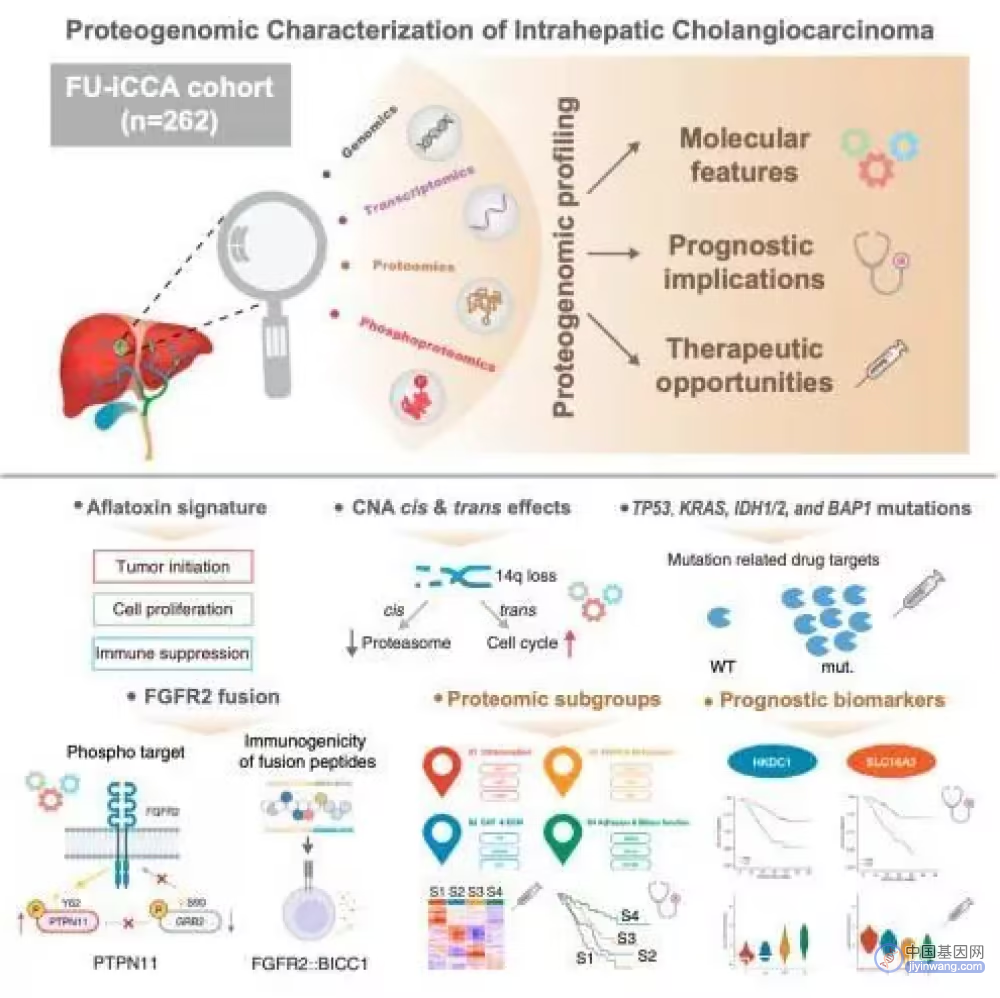
文章相关图片摘要 (https://doi.org/10.1016/j.ccell.2021.12.006)
该项研究发现,TP53、KRAS、FGFR2、IDH1/2、BAP1突变(融合)iCCA的主要驱动基因变异。此外,研究发现在中国人群iCCA样本中存在大量黄曲霉素突变指纹,且这些致癌物指纹与更高的肿瘤突变负荷、NK细胞高浸润等显著相关。研究者进一步分析了iCCA染色质拷贝数变异的多组学特征,发现抑癌基因丢失(如ARID1A、MAP2K4、MLH1)和致癌基因扩增(如STK19、HIST1H1E、MCL1、MDM4)是主要事件,且近40%的肝内胆管癌存在可药物靶向的基因改变。
无独有偶,近日JOH杂志在线发表了德国汉诺威医学院Arndt Vogel教授团队的一项大样本量的基因组学研究。该项回顾性研究共纳入来自Foundation CORE数据库的6130例iCCA患者,对>300个肿瘤相关基因的短变异/融合重排和拷贝数改变进行了评估。
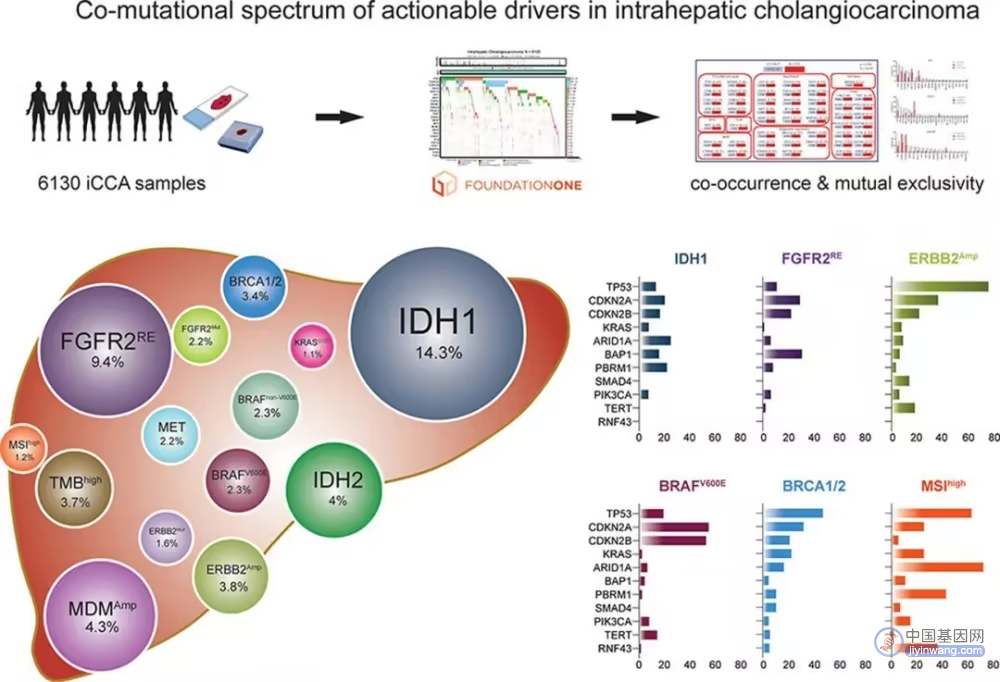
文章相关图片摘要 (https://doi.org/10.1016/j.jhep.2022.11.030)
最终,研究人员发现了七个与iCCA治疗应答相关的致癌驱动基因的共突变谱:IDH1/2、FGFR2、ERBB2、BRAF、MDM2、BRCA1/2、MET和KRASG12C。此外,研究人员在IDH1/2和FGFR2突变的患者中观察到RTK/RAS/ERK通路共突变的负向调节,以及相关表观遗传修饰物(如ARID1A和BAP1)的富集。RNF43和KMT2D突变在微卫星不稳定性(MSI)和肿瘤突变负荷(TMB)较高的iCCA患者中发生频率较高。
总 结
近年来,不断涌现出基于iCCA临床大样本的多组学研究,它们通过全景视角绘制iCCA的多维分子图谱,揭示了iCCA相关的多个肿瘤分子特征及潜在的治疗策略,为我们探究其发生发展机制、分子分型、预后监测和个性化治疗策略提供了新思路。
参考文献:
1. Dong L, Lu D, Chen R, et al. Proteogenomic characterization identifies clinically relevant subgroups of intrahepatic cholangiocarcinoma. Cancer Cell. 2022 Jan 10;40(1):70-87.e15.
2. Kendre G, Murugesan K, Brummer T, et al. Charting co-mutation patterns associated with actionable drivers in intrahepatic cholangiocarcinoma. J Hepatol. 2022 Dec 14:S0168-8278(22)03328-1.
3. Lee-Law PY, Olaizola P, Caballero-Camino FJ, et al. Targeting UBC9-mediated protein hyper-SUMOylation in cystic cholangiocytes halts polycystic liver disease in experimental models. J Hepatol. 2021 Feb;74(2):394-406.
4. Jusakul A, Cutcutache I, Yong CH, et al. Whole-Genome and Epigenomic Landscapes of Etiologically Distinct Subtypes of Cholangiocarcinoma. Cancer Discov. 2017 Oct;7(10):1116-1135.
5. Meric-Bernstam F, Hurwitz H, Raghav KPS, et al. Pertuzumab plus trastuzumab for HER2-amplified metastatic colorectal cancer (MyPathway): an updated report from a multicentre, open-label, phase 2a, multiple basket study. Lancet Oncol. 2019 Apr;20(4):518-530.
6. Skoulidis F, Byers LA, Diao L, et al. Co-occurring genomic alterations define major subsets of KRAS-mutant lung adenocarcinoma with distinct biology, immune profiles, and therapeutic vulnerabilities. Cancer Discov. 2015 Aug;5(8):860-77.
7. Liu R, Rizzo S, Waliany S, et al. Systematic pan-cancer analysis of mutation-treatment interactions using large real-world clinicogenomics data. Nat Med. 2022 Aug;28(8):1656-1661.
8. Sicklick JK, Kato S, Okamura R, et al. Molecular profiling of cancer patients enables personalized combination therapy: the I-PREDICT study. Nat Med. 2019 May;25(5):744-750.
9. Nakamura IT, Kohsaka S, Ikegami M, et al. Comprehensive functional evaluation of variants of fibroblast growth factor receptor genes in cancer. NPJ Precis Oncol. 2021 Jul 16;5(1):66.
10. Chen H, Ma J, Li W, et al. A molecular brake in the kinase hinge region regulates the activity of receptor tyrosine kinases. Mol Cell. 2007 Sep 7;27(5):717-30.
声明:本站所有文章资源内容,如无特殊说明或标注,均为采集网络资源。如若本站内容侵犯了原著者的合法权益,可联系本站删除。