一代、二代、三代、四代基因测序技术的发展简介与优缺点对比
基因测序,即通过对生物体的核酸序列进行检测,从而在分子和基因层面实现对生物体的进阶分析与解读。对于人类而言,究其根本都含有23对染色体、2.5万个基因编码、30亿个碱基对组成的bio-data综合体。通过对基因层面的解析,我们得以高效探究自己身体内终极密码。测序技术的不断进步使人类可以破解生命的密码,是分子生物学迅速发展的强大推动力之一。
近半世纪以来,基因测序技术飞速发展,从一代发展至四代,发生了日新月异的变化。

一代测序
一代测序是上世纪70年代由Sanger和Coulson开创的DNA双脱氧链终止法测序,也称为Sanger测序。在1977年完成第一个基因组序列(噬菌体X174),全长共计5375个碱基。2001年完成的首个人类基因组图谱就是以改进了的Sanger法为其测序基础。
Sanger测序原理:在4个DNA合成反应体系(含dNTP)中分别加入一定比例带有标记的ddNTP(分为:ddATP、ddCTP、ddGTP和ddTTP),通过凝胶电泳和放射自显影后可以根据电泳带的位置确定待测分子的DNA序列。由于ddNTP的2’和3’都不含羟基,其在DNA的合成过程中不能形成磷酸二酯键,因此可以用来中断DNA合成反应。

一代测序虽然准确度十分高,且该技术在当下依然被广泛应用(比如构建载体做克隆,基因敲除等实验都可以用到),但是通量太低,所以对于大片段或者全外显子等非常耗时,且很多情况下成本较高。
二代测序
二代测序技术,又称为Next Generation Sequencing(NGS)技术,是为了改进一代测序通量过低的问题而出现的,能够同时对上百万甚至数十亿个DNA分子进行测序,实现了大规模、高通量测序的目标。刚面世时主要包括Roche公司的454技术、ABI公司的Solid技术和Illumina公司的Solexa技术。这三种技术都极大的提高了测序的通量,大大降低了测序成本和周期。
二代测序技术在大幅提高了测序速度的同时,还大大地降低了测序成本,并且保持了高准确性,以前完成一个人类基因组的测序需要3年时间,而使用二代测序技术则仅仅需要1周,但其序列读长方面比起第一代测序技术则要短很多,大多只有100bp-150bp。
目前illumina的测序仪占全球75%以上,以NextSeq、HiSeq、NovaSeq等系列为主。它的机器采用的都是边合成边测序的方法,读长短(50-300bp);准确度达99.9%;通量很高。
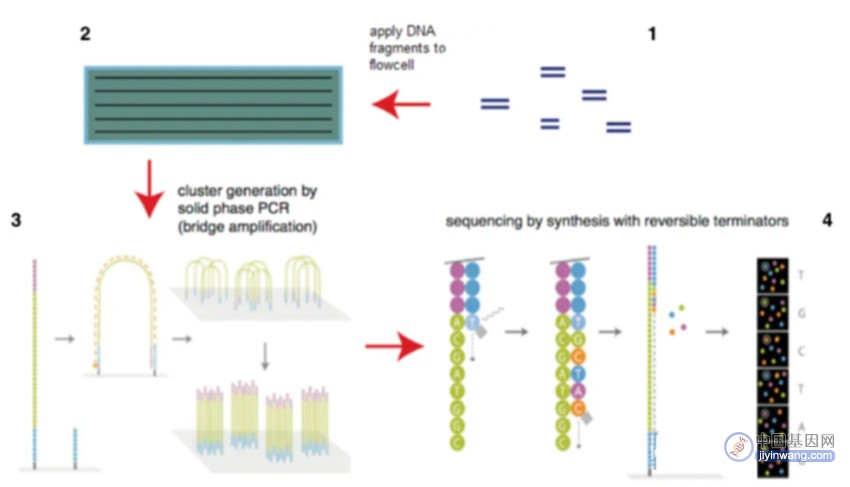
二代测序技术虽然通量很高,成本低廉,但是读长实在太短,主流的Illumina测序仪,常规模式只能测PE150的长度,靠着软件算法上的进步才得以可用。由此三代测序走上了历史舞台。
三代测序
三代测序主要有两种技术:PacBio公司的SMRT和Oxford Nanopore Technologies的纳米孔单分子测序技术,这两种技术的测序读长都可以达到几十kb的级别,远远高于二代测序技术。与前两代相比,他们最大的特点就是单分子测序,测序过程无需进行PCR扩增。实现了对每一条DNA分子的单独测序。单分子测序可以更准确地检测串联重复扩增等。这对于无参物种的分子生物学研究大有帮助,长读长对于基因组拼接、全长基因序列的获取提供了巨大的便利。
SMRT技术的测序速度很快,每秒约10个dNTP。但这么快的测序速度也带来了一些明显的缺点——测序错误率比较高(这几乎是目前单分子测序技术的通病),可以达到10%-15%,而且以随机的缺失序列和错位居多。
三代测序设备在DNA 序列片段读长上优于二代设备,但在准确度上较二代设备差,此外单样本的测序成本一直居高不下。
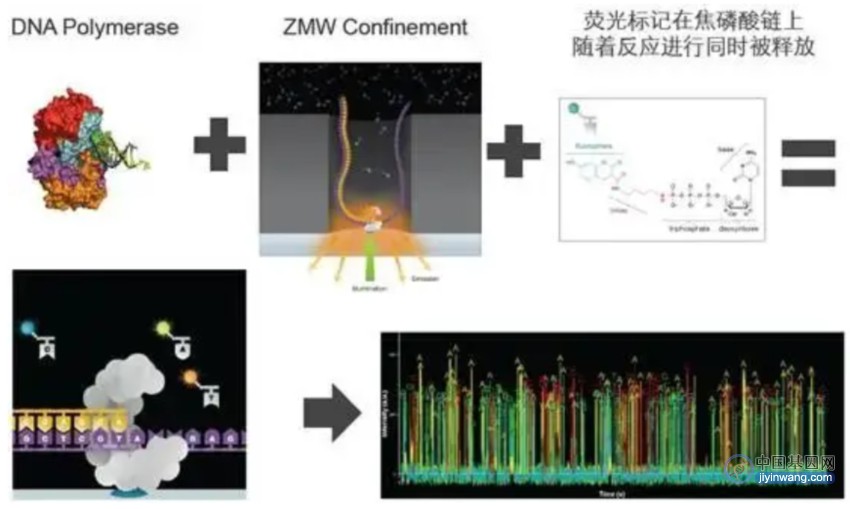
PacBio SMRT测序原理
四代测序-纳米孔测序
以Oxford Nanopore Technologies为代表的纳米孔测序技术与其他测序技术不同的是,它基于电信号而不是光信号。经历了三个主要的技术革新:一、单分子DNA从纳米孔通过;二、纳米孔上的酶对于测序分子在单核苷酸精度的控制;三、单核苷酸的测序精度控制。
将在某一面上含有一对电极的特殊脂质双分子层置于一个微孔之上,该双分子层中含有很多由α溶血素蛋白组成的纳米孔(直径2.6nm),只能容纳一个核苷酸通过,并且每个纳米孔会结合一个核酸外切酶。当DNA模板进入孔道时,孔道中的核酸外切酶会“抓住”DNA分子,顺序剪切掉穿过纳米孔道的DNA碱基,每一个碱基通过纳米孔时都会产生一个阻断,根据阻断电流的变化就能检测出相应碱基的种类,从而进行实时测序,最终得到DNA分子的序列。
Oxford Nanopore的特点是单分子测序,测序读长长(超过150kb),测序速度快,测序数据实时监控,机器方便携带等,但其单芯片测序成本还是在几百美金以上。
各代测序技术的对比
一代测序
优点:方法简便,分辨率高,测序片段长,流程细致,质控环节多,污染低,结果直观可视,假性结果极低。在单个基因测序方面具有优势。金标准。
缺点:测序试剂昂贵,通量低,处理比较长的同聚物时也有自身的问题。
二代测序
优点:通量高,一次可对几十万到几百万条核酸分子进行序列测定;单条序列成本低。
缺点:序列读长短;建库过程中有PCR过程,会引入错配碱基;要想得到准确和长度较长的拼接结果,需要较高的测序覆盖率,会增加成本和错误率。
三代测序
优点:
连贯性:连贯性对基因组的组装非常重要,如果连贯性比较好,能够准确的反应出基因结构之间的关系(外显子、基因簇、转移元件、调节序列等)。
完整性:如果一个基因组的测序深度>50X,理论上每一个碱基都会被测到。但实际上,基因组仍然会有很多缺失区域,比如即便是最新的人类参考基因组,其中仍然会有超过百万的“N”。长的提升能够有效提高基因组组装的完整性。
准确性:基因组组装的准确性可以在核酸水平或者结构变异水平进行描述。Illumina的三代测序技术的准确性非常高,每个碱基准确性大于99.9%,PacBio和Nanopore的准确性在足够测序深度的情况下,经过算法校正之后也能够达到99.9%。
缺点:单读长的错误率偏高,需要重复测序以纠错;依赖DNA聚合酶的活性;成本较高;生信分析软件不够丰富,数据积累少。
四代测序-纳米孔测序
优点:
超高的便携性,MinION可以做到即插即用,非常适合流行病疫区的即时检测;
超长的读长,MinION可以达到150kb,可用来填补之前测序的gap,可用于从头组装基因组,可变剪切等;
广泛的应用,可以直接对DNA,RNA和蛋白质序列进行测序;碱基判读准确率较高,R10纳米孔数据质量值超过Q40(即错误识别的概率是0.01%,即错误率0.01%),一致性(Identity)质量值达Q50 (99.999%的碱基准确率)。
缺点:低准确度,尤其是2D reads,虽然可以通过降低DNA通过速度或一致性检测提高准确度,但仍无法和二代测序相比。
基因测序技术的迅速发展为农业、环境和基础科学研究领域带来了新的突破和机遇。通过基因测序技术,科学家们可以深入研究遗传机制、改良农作物和家畜品种、修复环境和保护生态系统。随着技术的不断推进,基因测序技术有望在更多领域中发挥作用,为人类社会的可持续发展做出更大的贡献。
未来测序将更加专一化,具有更高的通量,测序设备微型化、高度集成,测序数据分析能力进一步提高,测序对象范围扩展,测序成本更低。
声明:本站所有文章资源内容,如无特殊说明或标注,均为采集网络资源。如若本站内容侵犯了原著者的合法权益,可联系本站删除。