全面解读融合基因检测:DNA or RNA? PCR or NGS?
融合基因是一类驱动肿瘤发生发展非常重要的变异形式。FDA或NMPA针对ALK、ROS1、RET、FGFR2和NTRK1/2/3等融合靶点已经批准了多款相应的靶向药物。融合基因的检测方法众多,临床上如何选择最合适的方法精准检测融合基因使更多患者获益呢?
本文通过对国内外研究数据的深入分析,全面解读DNA和RNA水平检测融合基因的生物学背景差异,融合基因检测该基于DNA水平还是RNA水平?以及首选检测技术平台是PCR还是NGS?

DNA和RNA用于融合基因检测的生物学原理
融合基因检测相关的基因序列特点
在DNA水平上,融合断点位置通常发生在较长的内含子区域,且融合的断裂点不同患者可能不同,故用传统的PCR直接扩增断裂点是不易实现的,采用NGS的方法设计探针去抓取断裂点是一种可行的检测方法,但从DNA水平检测融合基因不可避免存在如下局限性和挑战:1)融合基因检测要覆盖非常冗长且含有大量重复序列的内含子区域才能准确地找到融合断裂点;2)高GC含量不利于探针有效捕获目标区域片断;3)不同基因的内含子含有非常相似的重复序列,这一特征不利于序列准确对比,影响检测准确性;4)复杂的转录或转录后的剪接加工过程,可能会影响基因的融合。因此,不难理解的是在RNA水平检测融合基因是相对高效、精准的。
相比DNA水平,RNA水平上融合基因表现为前后两个基因外显子之间的衔接,融合点相对固定。这一特征为精准设计探针或引物提供了先天优势。因此,根据融合基因序列特点,在RNA水平上检测融合基因比DNA水平更易实现。
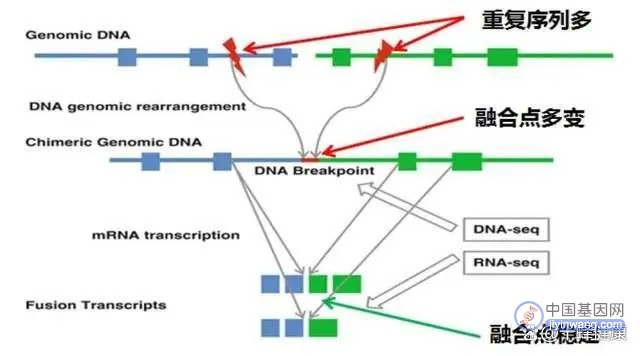
图1 DNA水平和RNA水平基因融合示意图
融合基因在RNA水平上“模板”急剧增多,更易检测
早在2012年Genome Res发表了一项关于肺腺癌的转录特征和突变特征的研究[1],该研究检测了200例肺腺癌手术标本的基因改变,其中87例进行RNA测序。研究显示不管是ALK融合、ROS1融合还是RET融合,融合基因的RNA表达量都显著上调(图2)。其原因是融合基因保留了其激酶催化结构域的3’端和融合伴侣启动子的5’端,融合伴侣在自身发生转录的同时,其下游激酶区域同时发生了转录。即在生理状态下原来不应该转录的激酶区域,在形成融合基因后,其激酶区域转录被激活产生了大量的mRNA。因此,在RNA水平上检测融合基因比DNA水平更敏感。
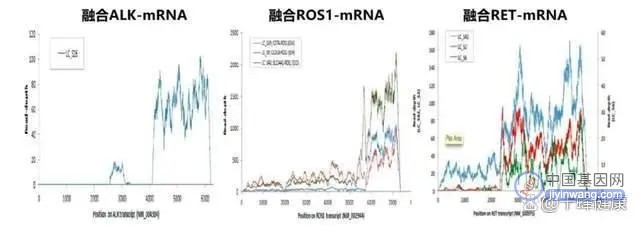
图2 RNA测序-融合基因分子数示意图
融合基因检测:用DNA还是RNA?
纽约纪念斯隆凯特琳癌症研究中心(MSKCC)的一项真实世界肺腺癌大队列研究中(图3)[2],DNAseq(MSK-IMPACT 468 genes DNA panel)检测的驱动基因阳性率为76.6%(1933/2522),其中融合阳性率为7.7%(195/2522)。DNAseq检测为驱动基因变异阴性的232例患者中,经RNAseq可检测到更多融合及MET 14号外显子跳跃15.5%(36/232)(图4),其中融合阳性患者为30例,将融合阳性率从7.7%(195/2522)提高到10.4%(225/2165),可见,RNAseq可挽回DNAseq漏检的融合。
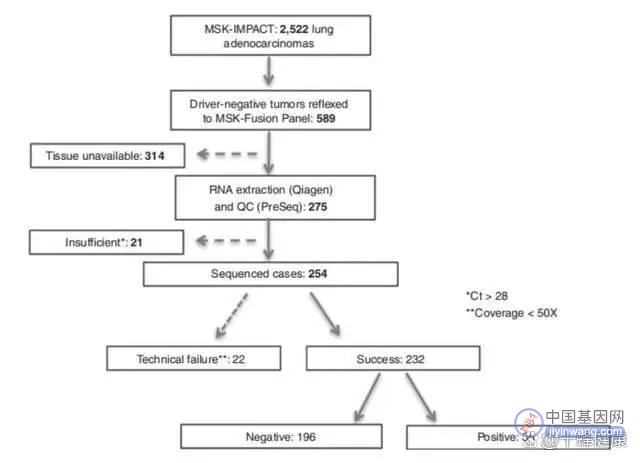
图3 肺腺癌队列
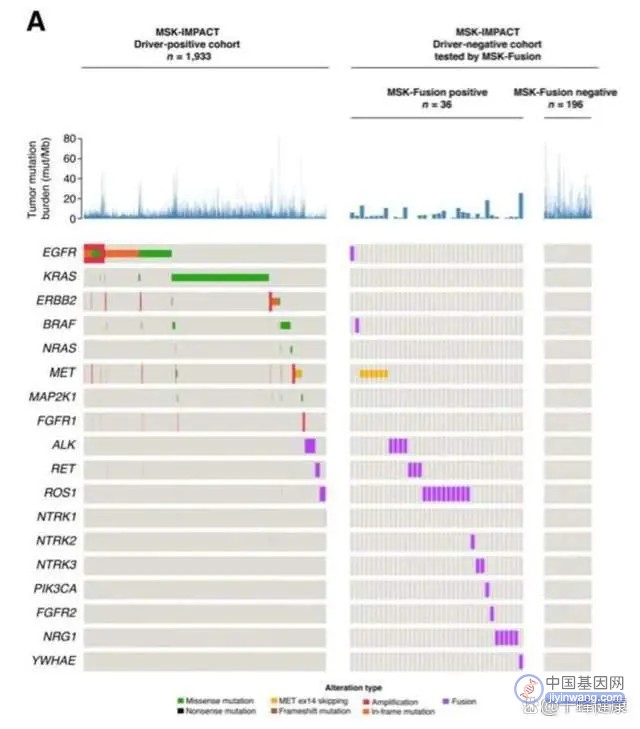
图4 DNAseq和RNAseq的驱动突变
在专业权威杂志J Thorac Oncol上发表一篇文章中[3],比较了DNAseq和RNAseq两种方法检测肺癌MET 14号外显子跳跃的检出率,两组数据结果显示,相较DNAseq,RNAseq更敏感(表一)。
表一 MET 14号外显子跳跃检测:DNAseq vs RNAseq
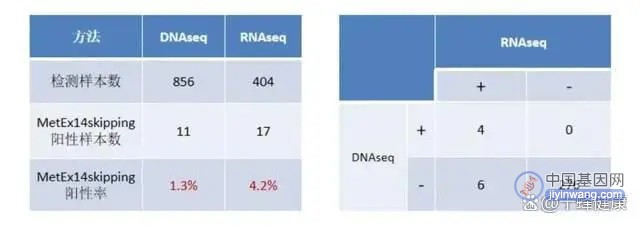
左侧表格为非头对头研究数据,右侧表格为头对头研究数据
泛癌人群的基因组图谱研究中,发表在国际顶级期刊Nat Biotechnol上的一篇研究对500例不同肿瘤类型的患者样本进行DNAseq和RNAseq[4],研究结果提示在泛癌人群中,相比DNAseq,RNAseq可检测出更多融合基因(图5)。
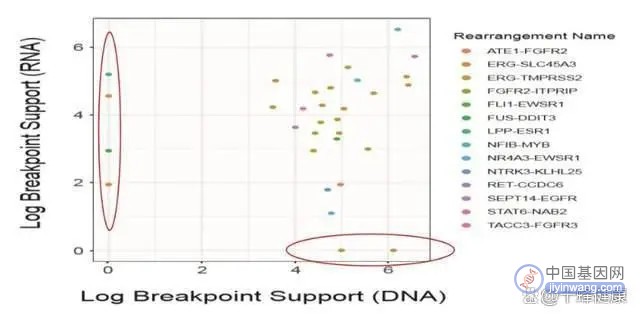
图5 DNAseq和RNAseq检测的融合基因比较
在实际检测中,DNA水平检测相比RNA水平的检测能够检出更多的稀有融合,但DNA水平检测到的融合基因是否真实参与了翻译和转录过程并最终表达出有功能的融合蛋白?是否真正驱动肿瘤的发生?哪种水平的检测更能预测相应靶向药物疗效?中国医学科学院肿瘤医院应建明主任团队在J Thorac Oncol发表的一篇文章很好地回答了这些问题[5],该研究收集了DNAseq检测到的24例稀有ALK融合,其中20例融合被RNAseq或IHC确认,4例未能被RNAseq或IHC确认,被RNAseq或IHC确认融合的患者相比未能被确认的患者具有更长克唑替尼的反应时间(PFS 11.0 mo vs 2.0 mo; 图6),即RNA/蛋白水平能确认的融合患者,靶向治疗才有效。
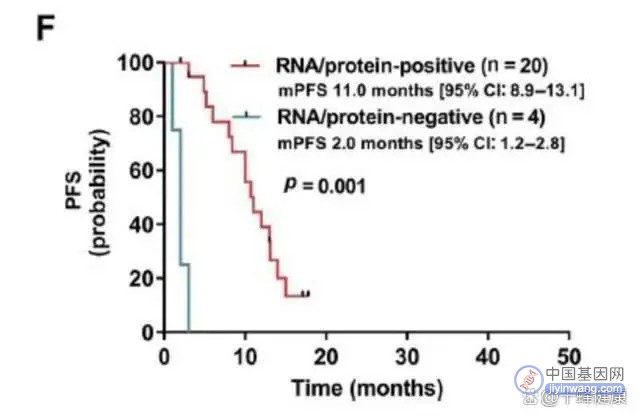
图6 ALK稀有融合患者的生存曲线
融合基因检测:首选PCR还是NGS?
FISH最初作为融合基因检测的金标准,但多项研究已显示敏感度不如RT-PCR[6-9],而且常规IHC和FISH均受主观因素影响。在一项比较IHC、FISH 和RT-PCR三种方法检测ALK融合的研究中,研究者认为RT-PCR应纳入融合基因检测的标准方法[9]。目前RT-PCR检测ALK、ROS1融合已写入CSCO及NCCN指南中,艾德RT-PCR是成熟的RNA融合检测技术,目前已有多篇研究显示出了其在融合基因检测中的优势。
基于RNA水平的 NGS方法 VS 基于RNA水平的RT-PCR方法
湘雅医院肖德胜主任团队发表的一篇研究[10],比较了不同检测方法(包括基于DNA水平的NGS方法,和基于RNA水平的艾德RT-PCR方法)检测非小细胞肺癌EML4-ALK融合基因的阳性率。研究结果显示,基于RNA水平的艾德RT-PCR方法比基于DNA水平的NGS方法敏感许多(表二)。
表二 不同检测方法检测EML4-ALK融合基因阳性率比较

基于RNA水平的融合检测:NGS VS RT-PCR
在2020 WCLC及CSCO会议上,日本国家癌症中心的Koichi Goto教授展示了亚洲肺癌基因组筛查项目(LC-SCRUM-Asia)中艾德PCR-9基因产品与Oncomine™ Comprehensive Assay(OCA,161 genes NGS-panel,ThermoFisher)v3在非小细胞肺癌驱动基因变异检测的直接对比结果。因Oncomine™同样是在RNA水平上检测融合,因此该研究具有极强的可比性。结果显示,在融合基因检测能力上,艾德PCR-9基因产品显示出更优异的检测能力。原因有二,其一为RT-PCR对短片段及末端融合的检测能力优于NGS;其二,RT-PCR有着更高的检测灵敏度,既更低的最低检测限。(图7)。
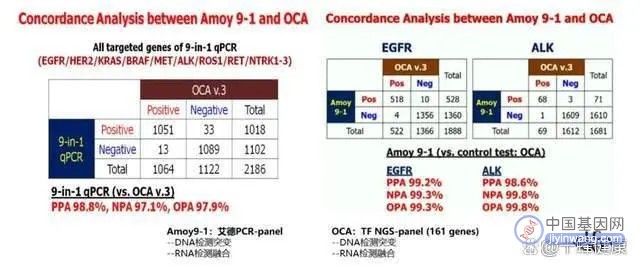
图7 Amoy 9-1和OCA检测一致性比较
Koichi Goto教授在此次2020 WCLC中还进一步比较了艾德PCR-9基因产品、OPA(50genes NGS-panel,ThermoFisher)和OCA 3个产品的检测失败率及报告时间(TAT)等基本参数(图8)。结果显示艾德PCR-9基因产品在检测成功率,尤其在RNA水平上的检测成功率、报告时间及普及度上均显示出了绝对优势。更高的检测成功率意味着将有更多的患者获得靶向药物治疗的机会;而更短的报告周期,免去了患者“焦急”等待检测报告时的“煎熬”,能够快速获得精准治疗。因此,基于艾德PCR-9基因的临床检测,可让更多患者更“从容”地享受当今精准医学发展带来的福利!
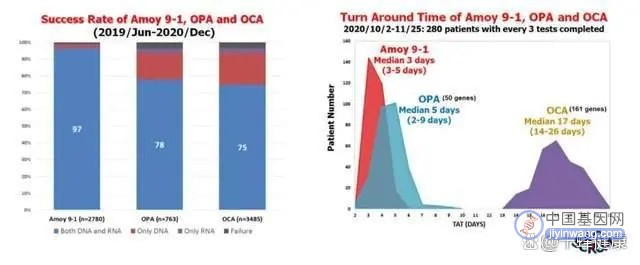
图8 Amoy 9-1、OPA和OCA检测失败率及报告时间比较
综合以上研究结论可概括为以下两点
√ 融合基因DNA和RNA水平上的结构特征决定了融合基因检测,RNA水平远比DNA水平更敏感,更准确。
√ 基于RNA检测融合基因,RT-PCR与NGS检测敏感度相当。相比NGS,RT-PCR在检测成功率、检测时间和技术普及度均优势显著。
声明:本站所有文章资源内容,如无特殊说明或标注,均为采集网络资源。如若本站内容侵犯了原著者的合法权益,可联系本站删除。