基于成像的CRISPR筛选绘制人必需基因的表型图谱
解析必需基因在不同细胞过程中的作用是理解细胞生长、增殖和生物学功能所必需的。近日,来自怀特黑德生物医学研究所的Iain M. Cheeseman和来自麻省理工学院博德研究所的Paul C. Blainey团队合作,共同在Cell上发表了Resource文章The phenotypic landscape of essential human genes。研究将CRISPR-Cas9为基础的功能筛选和显微镜成像结合,鉴定了5,072适应性基因中,超过90%基因敲除的显著表型,提示其在特定生物学过程中发挥作用,揭示基因间的关联,提供了人基础基因的功能全景。

基于CRISPR-Cas9的混合基因筛选能够干扰成千上万个遗传因子,革新了我们测试细胞增殖和存活需求对应功能的能力【1】。目前大多数的筛选方法基于荧光激活的细胞分选(FACS)【2,3】,它总结了每个扰动在群体水平上对细胞表型的贡献,而定义必需基因的特异贡献需要对复杂的细胞表型进行定量分析。近期的研究将混合功能遗传筛选和单细胞转录本丰度分析结合【4-6】,但对应核心细胞功能的很多表型,如亚细胞定位,需要显微镜进行直接的具象化,阵列筛选一定程度上解决了这一点,但是很难规模化【7】。为此,最近的混合筛选方法利用原位扰动基因分型【8】或基于图像表型物理筛选细胞【9】,但这一富集方法针对预先定义的表型,不能对做深度的关联探索。通过成千上万的基因组扰动,查询和系统比较大量和多样化的细胞生物学表型,特别是形态学和空间组织能力,是功能研究一个重要的未实现的目标。
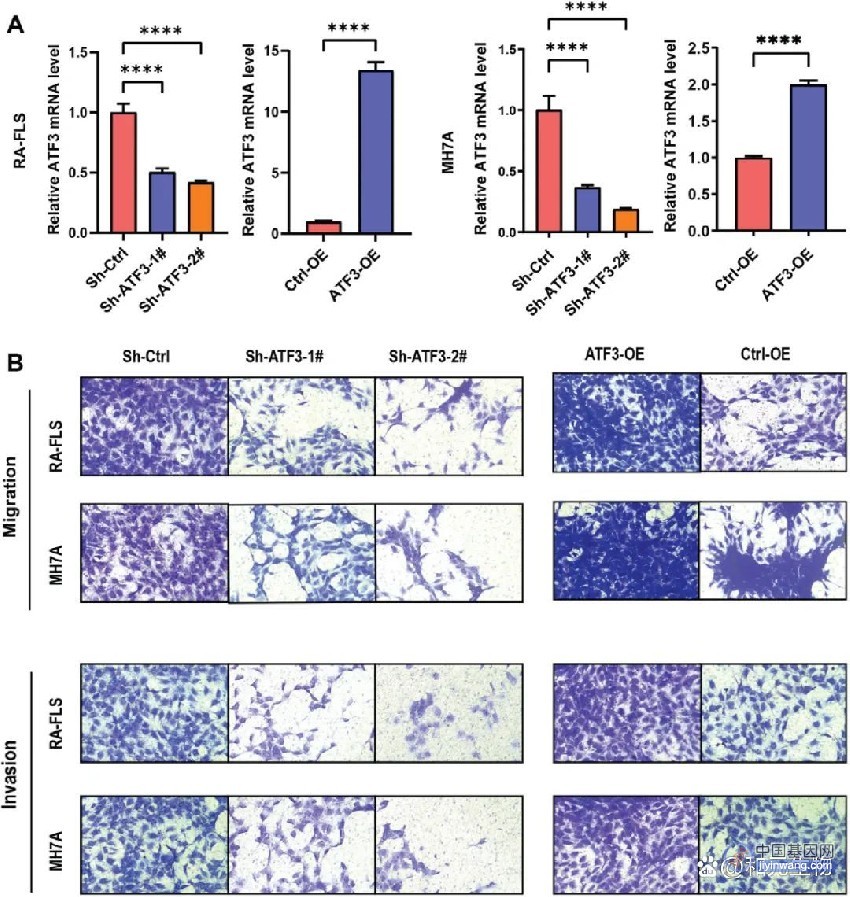
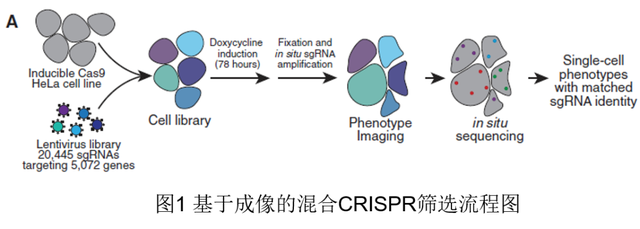
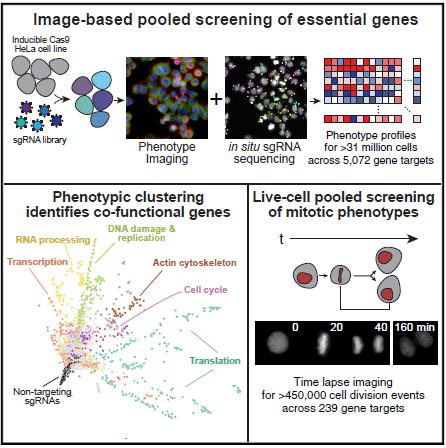
为解决以上问题,作者首先基于多轮Cas9和转座子遗传筛选,定义了5,072个适应性基因和一个包含20,445个sgRNAs的文库。将此文库递送至含有Cas9的HeLa细胞,78h后固定细胞进行原位sgRNA序列放大,DAPI、DNA损伤应答、微管和丝状肌动蛋白染色成像,原位测序,作者从每个单独的细胞图像中得到1,084个表型参数,基于此作者将其分为间期或有丝分裂期并进行下游分析。这一方法产生了31,884,270个单细胞的显微图像,其表型都与sgRNA相对应,每组四种sgRNAs的每个基因靶细胞中位数为6,119个细胞。
为了识别维持基因组完整性所必需的基因,作者分析了间期核参数的表型评分,观察到DNA损伤(平均γH2AX核强度)和DNA含量(整合DAPI核强度)之间存在明显的相关性。接下来,作者想要识别发挥细胞骨架功能的基因,他们发现破坏肌动蛋白的骨架,黏附分子或整合素亚单元都会扰乱细胞黏连和改变细胞形态。与之前研究不同,KCTD10在调节肌动蛋白装配上一直起作用,而不仅限于细胞迁移或发育编程的细胞融合时。此外,作者也发现了调节间期微管蛋白水平的多个因素。除染色参数外,作者还分析了细胞核和细胞区域的形态学参数,他们注意到靶向翻译相关基因导致细胞面积减少,而靶向DNA复制修复相关基因导致细胞面积增加。类似地,细胞核大小也存在显著差异。
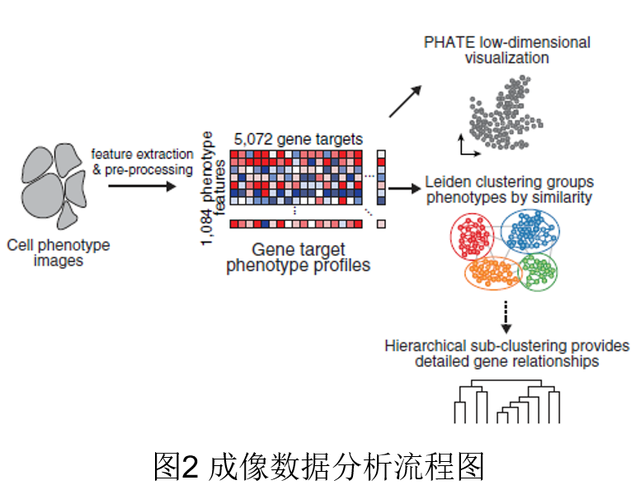
基于成像数据总结的表型得分,作者创建了每个基因的表型概况,识别出了具有相似间期表型的基因和一些功能亚单元基因,比如翻译相关会分9群,不同类群基因的敲除都会导致细胞核和细胞面积的减少,但其它表型参数会不同。意外地是,尽管没有针对细胞器的膜进行染色标记,作者仍发现了与囊泡运输相关的基因,说明间接的作用也能被筛选到。
至此,作者一共识别出4,665个展现可测量间期表型的基因。通过正交方法验证以及与之前注释比较,研究提供了一个特定蛋白质对核心细胞过程不同功能贡献的更为精细的图像,其可以应用于基因功能和途径关联的探究,识别关键生物学过程中共同作用的因子,包括之前刻画很少的基因,如C7orf26。
对于2.6%被划分为有丝分裂期的细胞,作者做了类似分析,识别了大量有丝分裂功能相关的基因类群,可以区分类群和单个基因的特异作用,发现了799个在MitoCheck数据中不存在的新基因,比如ZNF335促进纺锤体功能,在中心体功能中也发挥间接作用,这说明新的方法在灵敏度和有效性上都有了很大提升。
随后,作者进行了二次混合活细胞筛选,选取了228个认为与有丝分裂表型无关的基因和11个阳性对照,其中197个基因呈现出有丝分裂持续时间的改变,进一步验证了在固定细胞分析中识别不同有丝分裂分子的能力。出乎意料的是,作者鉴定出两种膜转运结合蛋白AQP7和ATP1A1,单独敲除其中任意一个都会导致染色体排列的延迟和有丝分裂持续时间的延长,但是在纺锤体或着丝粒装配中没有异常。PEG3000的加入发现AQP7和ATP1A1是创造内部渗透细胞环境所必需的,这一环境能促进染色体分离,揭示了渗透性及其调节在有丝分裂保真度上的作用。
进一步的研究发现有丝分裂表型的基础是着丝粒的功能,其在细胞分裂过程中介导着丝粒DNA和微管聚合物之间的相互作用。敲除LIN52, CLP1和RNPC3会带来关键有丝分裂分子产生的选择性缺陷,从而导致染色体错误分离和着丝粒功能障碍。
综合来看,研究通过显微镜对数千万个基因敲除细胞进行分析,发现了由此产生的细胞表型对核心生物过程的特定贡献,构建了一个全面的基因型——表型资源库,定义了大量未被阐释的人类必需基因的功能。
原文链接:
https://doi.org/10.1016/j.cell.2022.10.017
参考文献
1. Wang, T., Wei, J.J., Sabatini, D.M., and Lander, E.S. (2014). Genetic screens in human cells using the CRISPR-Cas9 system. Science 343, 80–84. https://doi.org/10.1126/science.1246981.
2. Condon, K.J., Orozco, J.M., Adelmann, C.H., Spinelli, J.B., van der Helm, P.W., Roberts, J.M., Kunchok, T., and Sabatini, D.M. (2021). Genome-wide CRISPR screens reveal multitiered mechanisms through which mTORC1 senses mitochondrial dysfunction. Proc. Natl. Acad. Sci. USA 118. e2022120118. https://doi.org/10.1073/pnas.2022120118.
3. Nieuwenhuis, J., Adamopoulos, A., Bleijerveld, O.B., Mazouzi, A., Stickel, E., Celie, P., Altelaar, M., Knipscheer, P., Perrakis, A., Blomen, V.A., and Brummelkamp, T.R. (2017). Vasohibins encode tubulin detyrosinating activity. Science 358, 1453–1456. https://doi.org/10.1126/science.aao5676.
4. Dixit, A., Parnas, O., Li, B., Chen, J., Fulco, C.P., Jerby-Arnon, L., Marjanovic, N.D., Dionne, D., Burks, T., Raychowdhury, R., et al. (2016). Perturb-Seq: dissecting molecular circuits with scalable single-cell RNA profiling of pooled genetic screens. Cell 167, 1853–1866.e17. https://doi.org/10.1016/j.cell.2016.11.038.
5. Replogle, J.M., Saunders, R.A., Pogson, A.N., Hussmann, J.A., Lenail, A., Guna, A., Mascibroda, L., Wagner, E.J., Adelman, K., Lithwick-Yanai, G., et al. (2022). Mapping information-rich genotype-phenotype landscapes with genome-scale Perturb-seq. Cell 185, 2559–2575.e28. https://doi.org/10.1016/j.cell.2022.05.013.
6. Datlinger, P., Rendeiro, A.F., Schmidl, C., Krausgruber, T., Traxler, P., Klughammer, J., Schuster, L.C., Kuchler, A., Alpar, D., and Bock, C. (2017). Pooled CRISPR screening with single-cell transcriptome readout. Nat. Methods 14, 297–301. https://doi.org/10.1038/nmeth.4177.
7. Neumann, B., Walter, T., He riche , J.-K., Bulkescher, J., Erfle, H., Conrad, C., Rogers, P., Poser, I., Held, M., Liebel, U., et al. (2010). Phenotypic profiling of the human genome by time-lapse microscopy reveals cell division genes. Nature 464, 721–727. https://doi.org/10.1038/nature08869.
8. Feldman, D., Singh, A., Schmid-Burgk, J.L., Carlson, R.J., Mezger, A., Garrity, A.J., Zhang, F., and Blainey, P.C. (2019). Optical pooled screens in human cells. Cell 179, 787–799.e17. https://doi.org/10.1016/j.cell.2019.09.016.
9. Hasle, N., Cooke, A., Srivatsan, S., Huang, H., Stephany, J.J., Krieger, Z., Jackson, D., Tang, W., Pendyala, S., Monnat, R.J., Jr., et al. (2020). Highthroughput, microscope-based sorting to dissect cellular heterogeneity. Mol. Syst. Biol. 16. e9442. https://doi.org/10.15252/msb.20209442.
声明:本站所有文章资源内容,如无特殊说明或标注,均为采集网络资源。如若本站内容侵犯了原著者的合法权益,可联系本站删除。